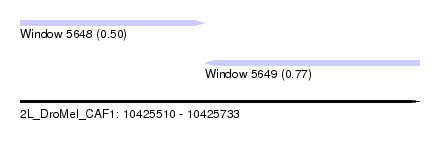
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 10,425,510 – 10,425,733 |
| Length | 223 |
| Max. P | 0.774399 |

| Location | 10,425,510 – 10,425,613 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 73.03 |
| Mean single sequence MFE | -40.38 |
| Consensus MFE | -26.84 |
| Energy contribution | -28.12 |
| Covariance contribution | 1.28 |
| Combinations/Pair | 1.12 |
| Mean z-score | -0.95 |
| Structure conservation index | 0.66 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
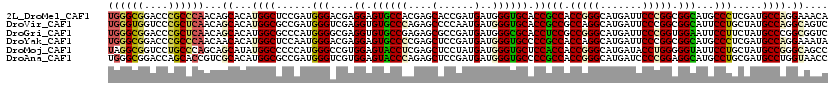
>2L_DroMel_CAF1 10425510 103 + 22407834 U------------A---GAUUGUACUUUAUACU--UAAUAGCCACCCCUGCCCGGUGGUGGUGCUCCACGGAUCAUGCUGGGCGGCGGUCCGCGCAUCGGUGGCGGAGCACCGCGUCCCA .------------.---(((.((..........--.....((((((.......))))))((((((((.((((((..(((....)))))))))(((....)))..)))))))))))))... ( -39.00) >DroGri_CAF1 2366 114 + 1 UUACUACAAAGAUAGAUGAGUG--CUGA-UAAUUUUAGUAGCCACCCCGUCCCGGCGGUGGUGCGCCACGCAUCAU---UGGUGGCGGUCCUCUCAUGGGUGGCGGUGCACCGCGUCCCA ...............(((((((--((((-.....))))))((((((((((....))))(((((((...))))))).---.))))))......)))))(((..((((....))))..))). ( -46.00) >DroEre_CAF1 1772 103 + 1 U------------A---AAUUUUACUUUAUACU--UAAUAACCACCCCUGCCUGGUGGUGGUGCUCCACGGAUCAUGCUGGGCGGCGGACCGCGCAUCGGUGGGGGAGCACCACGUCCCA .------------.---................--.....((((((.......))))))((((((((.(..(((((((...((((....)))))))).)))..)))))))))........ ( -36.60) >DroYak_CAF1 1727 103 + 1 U------------A---GUUUGUACUUUAUAUU--UAAUAGCCACCCCUGCCCGGUGGUGGUGCUCCACGGAUCAUGCUGGGCGGCGGACCGCGCAUCGGAGGGGGAGCACCGCGUCCCA .------------.---(..(((..........--.....((((((.......))))))((((((((.....((.((.((.((((....)))).)).))))...)))))))))))..).. ( -38.00) >DroMoj_CAF1 2097 115 + 1 UUACUAUGAAGAUAAGCGAAUGGGCUGUUUAAU--UAAUAUCCACCUCGUCCCGGUGGUGGUGCGCCGCGCAUCAU---UGGUGGUGGUCCUCGCAUGGGUGGUGGUGCACCCCGCCCAA ...............((((.((((.((((....--.))))))))..))))...(((((.((((((((((.((((..---..((((......))))...)))))))))))))))))))... ( -44.50) >DroAna_CAF1 1673 103 + 1 U------------G---GAAAAAAGUAUAAAAU--UAAUAUCCUCCCCUGCCCGGUGGCGGUGCCCCGCGAAUCAUGCUCGGCGGCGGUCCGCGCAUCGGUGGAGGUGCACCGCGUCCAA (------------(---((..............--......(((((.....((((((((((.(((((((((.......)).)))).))))))).)))))).))))).((...)).)))). ( -38.20) >consensus U____________A___GAUUGUACUUUAUAAU__UAAUAGCCACCCCUGCCCGGUGGUGGUGCUCCACGGAUCAUGCUGGGCGGCGGUCCGCGCAUCGGUGGCGGAGCACCGCGUCCCA ..........................................((((.......))))((((((((((....(((((((...((((....)))))))).)))...))))))))))...... (-26.84 = -28.12 + 1.28)
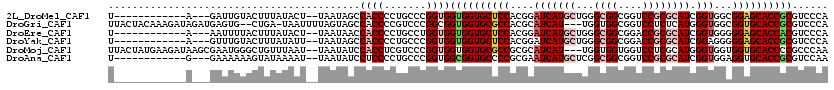
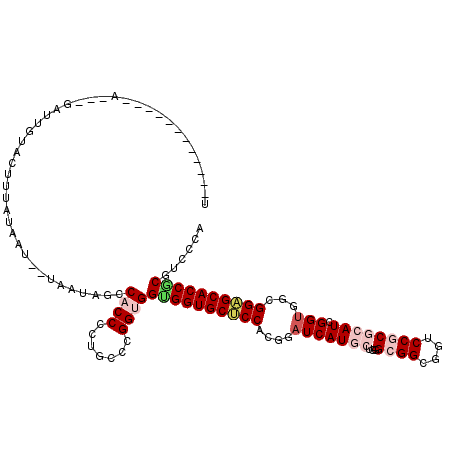
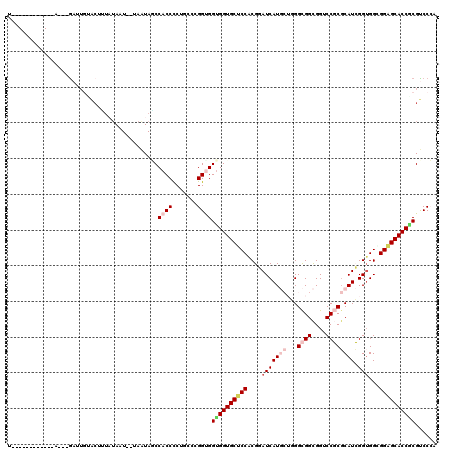
| Location | 10,425,613 – 10,425,733 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.28 |
| Mean single sequence MFE | -57.15 |
| Consensus MFE | -37.68 |
| Energy contribution | -37.85 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.54 |
| SVM RNA-class probability | 0.774399 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
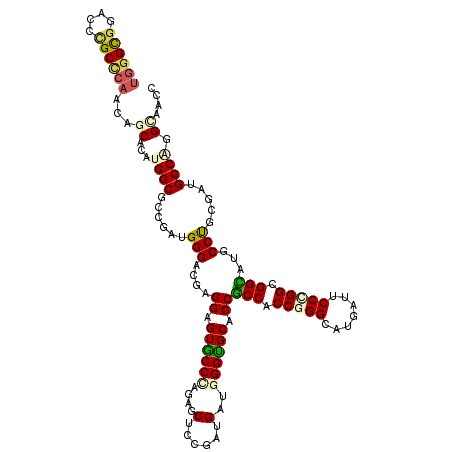
>2L_DroMel_CAF1 10425613 120 - 22407834 UGGGCGGACCCGCCCAACAGCACAUGGCUCCGAUGGGACGAGGAGUGCCACGAGCACCGAUGAUGGGUGCACCGCCACCGGGCAUGAUUCCCGGCGGCAUGCCCUCGAUGCCAGGAAACA ((((((....))))))........((((..(((.(((.((.((....)).)).(((((.......)))))...(((.(((((.......))))).)))...))))))..))))(....). ( -56.10) >DroVir_CAF1 2933 120 - 1 UGGGUGGUCCCGCUCAACAGCACAUGGCGCCGAUGGGUCGAGGUGUGCCCAGAGCCCCAAUGAUGGGUGCACCGCCGCCAGGCAUGAUUCCCGGCGGCAUUCCUGCUAUGCCAGGCAGUC ((((((....))))))...((...((((((((..((((((.((((..((((............))))..))))(((....))).)))))).))))((((....))))..)))).)).... ( -54.90) >DroGri_CAF1 2480 120 - 1 UGGGCGGACCCGCUCAACAGCACAUGGCGCCCAUGGGGCGAGGUGUGCCGAGAGCGCCGAUGAUGGGCGCACCUCCGCCGGGCAUGAUUCCCGGUGGAAUUCCUUCUAUGCCCGGCGGUC ((((((....)))))).(.((((((..(((((...)))))..)))))).)...(((((.......)))))....(((((((((((((((((....))))))......))))))))))).. ( -65.70) >DroYak_CAF1 1830 120 - 1 UGGGCGGACCCGCCCAACAACACAUGGCUCCAAUGGGACGAGGAGUGCCCCGAGCUCCGAUGAUGGGUGCCCCGCCACCAGGCAUGAUUCCCGGCGGCAUGCCCUCGAUGCCAGGAAAUA ((((((....))))))........(((((((....)))(((((.((((((((.((.((.......)).))...(((....)))........))).)))))..)))))..))))....... ( -50.60) >DroMoj_CAF1 2212 120 - 1 UAGGCGGUCCUGCCCAGCAGCAUAUGGCCCCCAUGGGCCGUGGAGUACCUCGAGCUCCUAUGAUGGGUGCUCCACCACCGGGCAUGAUACCUGGGGGUAUUCCUGCUAUGCCGGGCAGCC ..(((.((((.((..((((....(((((((....)))))))(((((((((.((((.(((.....))).)))).....(((((.......))))))))))))))))))..)).)))).))) ( -59.70) >DroAna_CAF1 1776 120 - 1 UGGGCGGACCAGCACCGUCGCACAUGGCGCCGAUGGGUCGUGGAGUACCCAGAGCUCCGAUGAUGGGUGCCCCGCCACCGGGCAUGAUCCCCGGAGGCAUGCCUGCGAUGCCUGGUAACC .((....(((((...(((((((((((.(.(((..(((((((((.(((((((...(......).))))))).))(((....)))))))))).))).).))))..))))))).)))))..)) ( -55.90) >consensus UGGGCGGACCCGCCCAACAGCACAUGGCGCCGAUGGGACGAGGAGUGCCCAGAGCUCCGAUGAUGGGUGCACCGCCACCGGGCAUGAUUCCCGGCGGCAUGCCUGCGAUGCCAGGCAACC ((((((....))))))...((...((((......(((....((.((((((....(......)..)))))).))(((.(((((.......))))).)))...))).....)))).)).... (-37.68 = -37.85 + 0.17)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:55:58 2006