
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 10,390,619 – 10,390,736 |
| Length | 117 |
| Max. P | 0.987377 |

| Location | 10,390,619 – 10,390,736 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.03 |
| Mean single sequence MFE | -29.97 |
| Consensus MFE | -26.78 |
| Energy contribution | -27.00 |
| Covariance contribution | 0.22 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.95 |
| SVM RNA-class probability | 0.983794 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 10390619 117 + 22407834 AAUCCCCCAGCCGUGUUCAAUUCGCCGCAGGCCCACAACCAUAACCACACAGUACAAGCACAGCACAGUGCACUGUCCACGGCUGGACCGCUUGGCCAUCACU---CGCUCAACCACACG ......(((((((((........(((...)))................(((((....((((......))))))))).)))))))))...(.(((((.......---.)).))).)..... ( -31.00) >DroSec_CAF1 5530 117 + 1 AAUCCCCCAGCCGUGUUCAAUUCGCCGCAGGCCCACAACCAUAACCACACAGUACAAGCACAGCACAGUGCACUGUCCACGGCUGGACCGCUUGGCCAUCACU---CGCUCAACCACACG ......(((((((((........(((...)))................(((((....((((......))))))))).)))))))))...(.(((((.......---.)).))).)..... ( -31.00) >DroSim_CAF1 5450 117 + 1 AAUCCCCCAGCCGUGUUCAAUUCGCCGCAGGCCCACAACCAUAACCACACAGUACAAGCACAGCACAGUGCACUGUCCACGGCUGGACCGCUUGGCCAUCACU---CGCUCAACCACACG ......(((((((((........(((...)))................(((((....((((......))))))))).)))))))))...(.(((((.......---.)).))).)..... ( -31.00) >DroEre_CAF1 5931 117 + 1 AAUCCCCCAGCCGUGUUCAACUCUCCGCAGGCCCACAACCAUAACCACACAGUACAAGCACAGCACAGUGCACUGUCCACGGCUGGACCGCUUGGCCAUCACU---CGCUCAACCACACG ......(((((((((..............((.......))........(((((....((((......))))))))).)))))))))...(.(((((.......---.)).))).)..... ( -29.30) >DroYak_CAF1 6042 117 + 1 AAUCCCCCAGCCGUGUUCAAUUCGCCGCAGGCCCACAACCAUAACCACACAGUACAAGCACAGCACAGUGCACUGUCCACGGCUGGACCGCUUGGCCAUCACU---CGCUCAACCACACG ......(((((((((........(((...)))................(((((....((((......))))))))).)))))))))...(.(((((.......---.)).))).)..... ( -31.00) >DroAna_CAF1 4656 114 + 1 AAUACCCCAGCCGUGUUCAACUCGCCGCAGCCCCACAA------CCACGCAGUACAAGCUCAGCACAGUGCAUUGUCCACGGCUGGACCGCUUGGCCAUCACUCAUCGCUCAACCAUACG ......(((((((((..(((..(((.((.((.......------....))(((....)))..))...)))..)))..)))))))))...(.(((((.((.....)).)).))).)..... ( -26.50) >consensus AAUCCCCCAGCCGUGUUCAAUUCGCCGCAGGCCCACAACCAUAACCACACAGUACAAGCACAGCACAGUGCACUGUCCACGGCUGGACCGCUUGGCCAUCACU___CGCUCAACCACACG ......(((((((((........(((...)))................(((((....((((......))))))))).)))))))))...(.(((((...........)).))).)..... (-26.78 = -27.00 + 0.22)
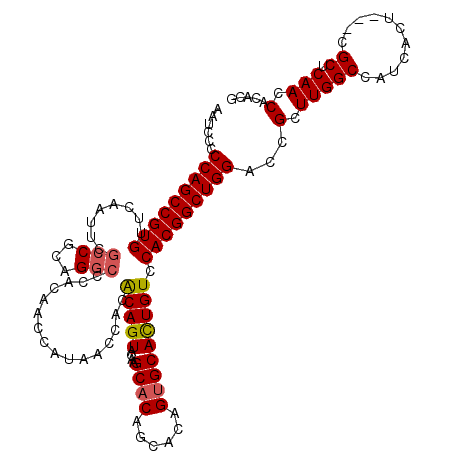

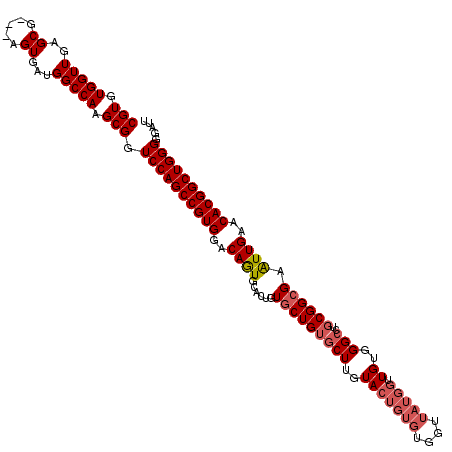
| Location | 10,390,619 – 10,390,736 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.03 |
| Mean single sequence MFE | -45.08 |
| Consensus MFE | -43.11 |
| Energy contribution | -44.08 |
| Covariance contribution | 0.97 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.48 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.08 |
| SVM RNA-class probability | 0.987377 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 10390619 117 - 22407834 CGUGUGGUUGAGCG---AGUGAUGGCCAAGCGGUCCAGCCGUGGACAGUGCACUGUGCUGUGCUUGUACUGUGUGGUUAUGGUUGUGGGCCUGCGGCGAAUUGAACACGGCUGGGGGAUU (((.(((((.....---......))))).))).((((((((((.((((....))))((((((((..(((((((....))))).))..)))..)))))........))))))))))..... ( -45.30) >DroSec_CAF1 5530 117 - 1 CGUGUGGUUGAGCG---AGUGAUGGCCAAGCGGUCCAGCCGUGGACAGUGCACUGUGCUGUGCUUGUACUGUGUGGUUAUGGUUGUGGGCCUGCGGCGAAUUGAACACGGCUGGGGGAUU (((.(((((.....---......))))).))).((((((((((.((((....))))((((((((..(((((((....))))).))..)))..)))))........))))))))))..... ( -45.30) >DroSim_CAF1 5450 117 - 1 CGUGUGGUUGAGCG---AGUGAUGGCCAAGCGGUCCAGCCGUGGACAGUGCACUGUGCUGUGCUUGUACUGUGUGGUUAUGGUUGUGGGCCUGCGGCGAAUUGAACACGGCUGGGGGAUU (((.(((((.....---......))))).))).((((((((((.((((....))))((((((((..(((((((....))))).))..)))..)))))........))))))))))..... ( -45.30) >DroEre_CAF1 5931 117 - 1 CGUGUGGUUGAGCG---AGUGAUGGCCAAGCGGUCCAGCCGUGGACAGUGCACUGUGCUGUGCUUGUACUGUGUGGUUAUGGUUGUGGGCCUGCGGAGAGUUGAACACGGCUGGGGGAUU (((.(((((.....---......))))).))).((((((((((..((..((.((.(((.(.(((..(((((((....))))).))..)))).))).)).))))..))))))))))..... ( -44.30) >DroYak_CAF1 6042 117 - 1 CGUGUGGUUGAGCG---AGUGAUGGCCAAGCGGUCCAGCCGUGGACAGUGCACUGUGCUGUGCUUGUACUGUGUGGUUAUGGUUGUGGGCCUGCGGCGAAUUGAACACGGCUGGGGGAUU (((.(((((.....---......))))).))).((((((((((.((((....))))((((((((..(((((((....))))).))..)))..)))))........))))))))))..... ( -45.30) >DroAna_CAF1 4656 114 - 1 CGUAUGGUUGAGCGAUGAGUGAUGGCCAAGCGGUCCAGCCGUGGACAAUGCACUGUGCUGAGCUUGUACUGCGUGG------UUGUGGGGCUGCGGCGAGUUGAACACGGCUGGGGUAUU (((.(((((..((.....))...))))).))).((((((((((..((((......(((((((((....(..((...------.))..))))).))))).))))..))))))))))..... ( -45.00) >consensus CGUGUGGUUGAGCG___AGUGAUGGCCAAGCGGUCCAGCCGUGGACAGUGCACUGUGCUGUGCUUGUACUGUGUGGUUAUGGUUGUGGGCCUGCGGCGAAUUGAACACGGCUGGGGGAUU (((.(((((..((.....))...))))).))).((((((((((..((((......(((((((((..(((((((....))))).))..)))..)))))).))))..))))))))))..... (-43.11 = -44.08 + 0.97)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:55:42 2006