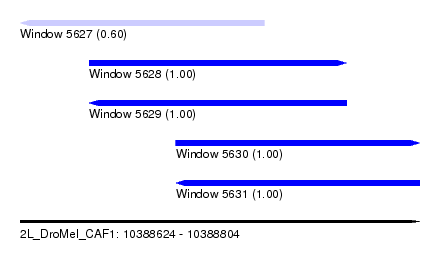
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 10,388,624 – 10,388,804 |
| Length | 180 |
| Max. P | 0.999962 |

| Location | 10,388,624 – 10,388,734 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 90.04 |
| Mean single sequence MFE | -23.85 |
| Consensus MFE | -18.80 |
| Energy contribution | -19.00 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.596250 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 10388624 110 - 22407834 CAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAUACAUUUAAGAGGAAUUAAUUCGUACAUAAGGCUGU-AUACGCACGGGUAUAUACACACAUUCAAC-AAAUAA ..((((((((((((..(((((((....))))).))..)))))......(((.......)))))))))).(..(((-(((..(....)..))))))..).......-...... ( -22.30) >DroSec_CAF1 3546 111 - 1 CAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAUACAUUUAAGAGGAAUUAAUUCGUACAUAAGGCUGUUGUACCCACGGGUAUAUACACACAUUCAAC-AAAUAA ..((((((((((((..(((((((....))))).))..)))))......(((.......)))))))))).(..(((((((((....)))))).)))..).......-...... ( -25.10) >DroSim_CAF1 3429 111 - 1 CAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAUACAUUUAAGAGGAAUUAAUUCGUACAUAAGGCUGUUGUACCCACGGGUAUAUACACACAUUCAAC-AAAUAA ..((((((((((((..(((((((....))))).))..)))))......(((.......)))))))))).(..(((((((((....)))))).)))..).......-...... ( -25.10) >DroEre_CAF1 3608 110 - 1 CAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAUACAUUUAAGACAAAUUAAUUCGUACAUAAGGCUGU-GUACCCACGGGUAUAUACACACAUUCAAC-AAAUAA .(((((((((((((..(((((((....))))).))..)))))))............................(((-(((((....)))))))).)))))).....-...... ( -26.10) >DroYak_CAF1 3650 110 - 1 CAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAUACAUUUAAGAGGAAUUAAUUCGUACAUAAGGCUGU-AUACCCACGGGUAUAUACACACAUUCAAC-AAAUAA ..((((((((((((..(((((((....))))).))..)))))......(((.......)))))))))).(..(((-(((((....))))))))..).........-...... ( -26.10) >DroAna_CAF1 3197 105 - 1 CAUGUGUAUGUAUGCAGAAUCAUGAAAAU-AUAUCCACAUACAUUUAAACGGAAUUAAUUCGUGCAUAGCUUCGUUGUACA------UAUAUAUUUAUAUCUACCGAAAUCC .(((((.((((((.((......))...))-)))).))))).........(((.........(((((.........)))))(------((((....)))))...)))...... ( -18.40) >consensus CAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAUACAUUUAAGAGGAAUUAAUUCGUACAUAAGGCUGU_GUACCCACGGGUAUAUACACACAUUCAAC_AAAUAA ..((((((((((((..(((((((....))))).))..)))))..................................(((((....))))))))))))............... (-18.80 = -19.00 + 0.20)

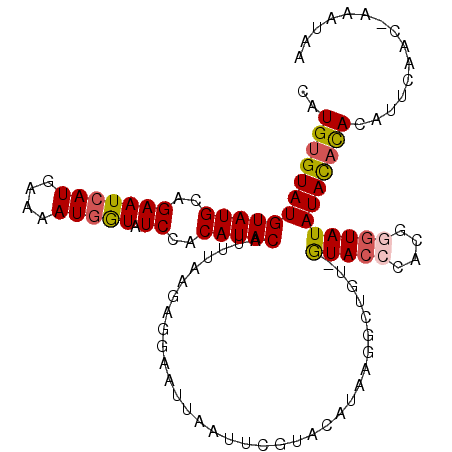
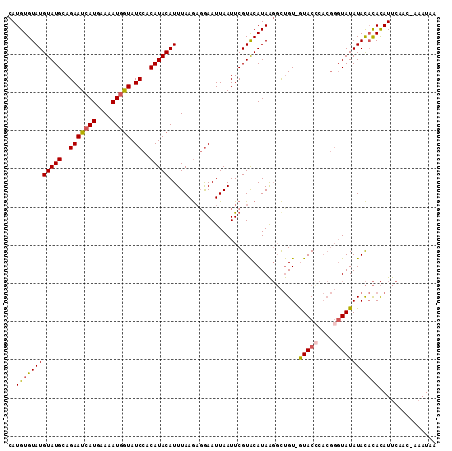
| Location | 10,388,655 – 10,388,771 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 94.58 |
| Mean single sequence MFE | -28.50 |
| Consensus MFE | -25.50 |
| Energy contribution | -25.50 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.03 |
| Structure conservation index | 0.89 |
| SVM decision value | 3.51 |
| SVM RNA-class probability | 0.999319 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 10388655 116 + 22407834 GUAU-ACAGCCUUAUGUACGAAUUAAUUCCUCUUAAAUGUAUGUGGAUACCAUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGUAUAUACGAAUGCUCGUAAACAA ((.(-((((((.((((((((((((............(((...((((...))))...))))))))(((((((((((....)))))))))))...))))))).)...))).))).)).. ( -28.00) >DroSec_CAF1 3577 117 + 1 GUACAACAGCCUUAUGUACGAAUUAAUUCCUCUUAAAUGUAUGUGGAUACCAUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGCAUGUACGAAUGCUCGUAAACAA ................(((((....((((.......(((((((..((...(((....)))..))..)))))))..((((((((((....))))))))))..))))..)))))..... ( -28.90) >DroSim_CAF1 3460 117 + 1 GUACAACAGCCUUAUGUACGAAUUAAUUCCUCUUAAAUGUAUGUGGAUACCAUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGCAUGUACGAAUGCUCGUAAACAA ................(((((....((((.......(((((((..((...(((....)))..))..)))))))..((((((((((....))))))))))..))))..)))))..... ( -28.90) >DroEre_CAF1 3639 116 + 1 GUAC-ACAGCCUUAUGUACGAAUUAAUUUGUCUUAAAUGUAUGUGGAUACCAUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGUAUGUACAAAUGCUCGUAAACAA ((..-(((((.((.((((((................(((((((..((...(((....)))..))..)))))))..((((((((....)))))))).)))))))).))).))..)).. ( -27.90) >DroYak_CAF1 3681 116 + 1 GUAU-ACAGCCUUAUGUACGAAUUAAUUCCUCUUAAAUGUAUGUGGAUACCAUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGCAUGUACGAAUGCUCGUAAACAA ((.(-(((((.((.((((((((....))).......(((((((((((...(((....)))..)))((((((((((....))))))))))))))))))))))))).))).))).)).. ( -29.40) >DroAna_CAF1 3223 116 + 1 GUACAACGAAGCUAUGCACGAAUUAAUUCCGUUUAAAUGUAUGUGGAUAU-AUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGCAUGUACGAAUGCUCGUAAACAA .....((((.((((((((...........(((..((((((((....))))-))))..)))....(((((((((((....)))))))))))..)))))).......))))))...... ( -27.91) >consensus GUAC_ACAGCCUUAUGUACGAAUUAAUUCCUCUUAAAUGUAUGUGGAUACCAUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGCAUGUACGAAUGCUCGUAAACAA ................(((((....((((.......(((((((..((...(((....)))..))..)))))))..((((((((((....))))))))))..))))..)))))..... (-25.50 = -25.50 + 0.00)
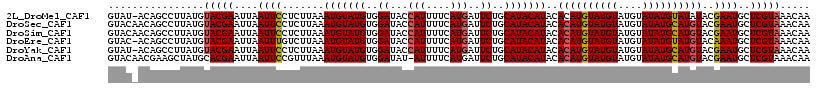
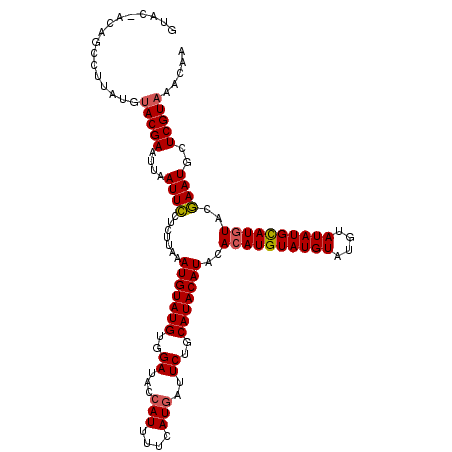
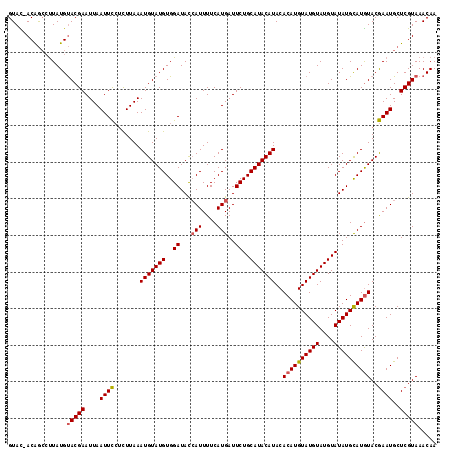
| Location | 10,388,655 – 10,388,771 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 94.58 |
| Mean single sequence MFE | -27.08 |
| Consensus MFE | -26.25 |
| Energy contribution | -25.37 |
| Covariance contribution | -0.89 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.97 |
| SVM decision value | 4.46 |
| SVM RNA-class probability | 0.999901 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 10388655 116 - 22407834 UUGUUUACGAGCAUUCGUAUAUACAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAUACAUUUAAGAGGAAUUAAUUCGUACAUAAGGCUGU-AUAC .(((.((((......))))...))).....(((((..(.((((((((((((..(((((((....))))).))..)))))......(((.......)))))))))).)..)))-)).. ( -25.30) >DroSec_CAF1 3577 117 - 1 UUGUUUACGAGCAUUCGUACAUGCAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAUACAUUUAAGAGGAAUUAAUUCGUACAUAAGGCUGUUGUAC ((((.((((((.((((...(((((((((((((......)))))))))))))....(((((....)))))...................))))...)))))).))))........... ( -26.90) >DroSim_CAF1 3460 117 - 1 UUGUUUACGAGCAUUCGUACAUGCAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAUACAUUUAAGAGGAAUUAAUUCGUACAUAAGGCUGUUGUAC ((((.((((((.((((...(((((((((((((......)))))))))))))....(((((....)))))...................))))...)))))).))))........... ( -26.90) >DroEre_CAF1 3639 116 - 1 UUGUUUACGAGCAUUUGUACAUACAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAUACAUUUAAGACAAAUUAAUUCGUACAUAAGGCUGU-GUAC ((((.((((((.((((((.(((((((((((((......)))))))))))))....(((((....))))).................))))))...)))))).))))......-.... ( -28.60) >DroYak_CAF1 3681 116 - 1 UUGUUUACGAGCAUUCGUACAUGCAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAUACAUUUAAGAGGAAUUAAUUCGUACAUAAGGCUGU-AUAC ..((.(((.(((....((((.((((..(((((((((....)))))))))))))(((((((....))))).(((..............)))......)))))).....)))))-).)) ( -27.34) >DroAna_CAF1 3223 116 - 1 UUGUUUACGAGCAUUCGUACAUGCAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAU-AUAUCCACAUACAUUUAAACGGAAUUAAUUCGUGCAUAGCUUCGUUGUAC ..((..((((((((((...(((((((((((((......)))))))))))))..))))((((((...-((.(((..............)))))...))))))....)).))))...)) ( -27.44) >consensus UUGUUUACGAGCAUUCGUACAUGCAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAUACAUUUAAGAGGAAUUAAUUCGUACAUAAGGCUGU_GUAC ((((.((((((.((((...(((((((((((((......)))))))))))))....(((((....)))))...................))))...)))))).))))........... (-26.25 = -25.37 + -0.89)

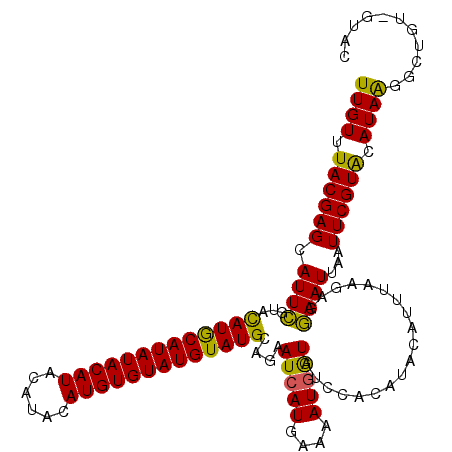
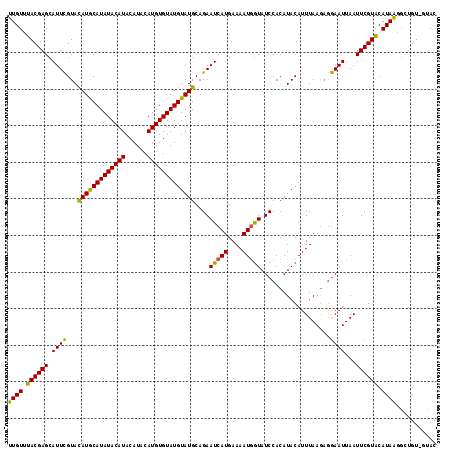
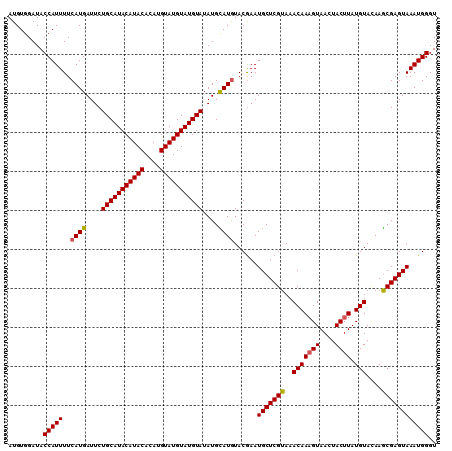
| Location | 10,388,694 – 10,388,804 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 97.64 |
| Mean single sequence MFE | -30.76 |
| Consensus MFE | -30.26 |
| Energy contribution | -30.26 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.59 |
| Structure conservation index | 0.98 |
| SVM decision value | 4.93 |
| SVM RNA-class probability | 0.999962 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 10388694 110 + 22407834 AUGUGGAUACCAUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGUAUAUACGAAUGCUCGUAAACAAAGUAACUACUUAUGUACAAACGAGUAAAUGGGU .........((((((.....(((((((((((((((....)))))))))))..(((((((((((....)))))....((((....)))).))))))...)))))))))).. ( -28.50) >DroSec_CAF1 3617 110 + 1 AUGUGGAUACCAUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGCAUGUACGAAUGCUCGUAAACAAUGUAACUACUUAUGUACAAGCGAGUAAAUGGGU .........(((((..((((....(((((((((((....)))))))))))....))))......(((((((.....((((((....))))))....)))))))))))).. ( -30.20) >DroSim_CAF1 3500 110 + 1 AUGUGGAUACCAUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGCAUGUACGAAUGCUCGUAAACAAAGUAACUACUUAUGUACAAGCGAGUAAAUGGGU .........(((((..((((....(((((((((((....)))))))))))....))))......(((((((..(((((((....)))).)))....)))))))))))).. ( -32.30) >DroEre_CAF1 3678 110 + 1 AUGUGGAUACCAUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGUAUGUACAAAUGCUCGUAAACAAAGUAACUACUUAUGUACAAGCGAGUAAAUGGGU .........(((((..((((....(((((((((((....)))))))))))....))))......(((((((..(((((((....)))).)))....)))))))))))).. ( -30.50) >DroYak_CAF1 3720 110 + 1 AUGUGGAUACCAUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGCAUGUACGAAUGCUCGUAAACAAAGUAACUACUUAUGUAUAAGCGAGUAAAUGGGU .........(((((..((((....(((((((((((....)))))))))))....))))......(((((((..(((((((....)))).)))....)))))))))))).. ( -32.30) >consensus AUGUGGAUACCAUUUUCAUGAUUCUGCAUACAUACACAUGUAUGUAUGUAUAUGCAUGUACGAAUGCUCGUAAACAAAGUAACUACUUAUGUACAAGCGAGUAAAUGGGU .........(((((..((((....(((((((((((....)))))))))))....))))......(((((((..(((((((....)))).)))....)))))))))))).. (-30.26 = -30.26 + -0.00)


| Location | 10,388,694 – 10,388,804 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 97.64 |
| Mean single sequence MFE | -30.92 |
| Consensus MFE | -31.40 |
| Energy contribution | -30.88 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.11 |
| Mean z-score | -4.56 |
| Structure conservation index | 1.02 |
| SVM decision value | 4.33 |
| SVM RNA-class probability | 0.999873 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
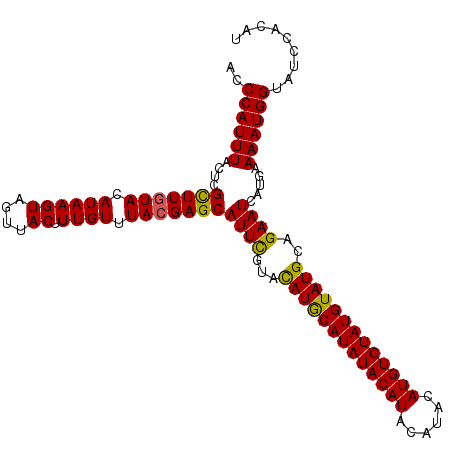
>2L_DroMel_CAF1 10388694 110 - 22407834 ACCCAUUUACUCGUUUGUACAUAAGUAGUUACUUUGUUUACGAGCAUUCGUAUAUACAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAU ..((((((.(((((....(((.((((....)))))))..))))).((((...(((((((((((((......)))))))))))))..)))).....))))))......... ( -28.80) >DroSec_CAF1 3617 110 - 1 ACCCAUUUACUCGCUUGUACAUAAGUAGUUACAUUGUUUACGAGCAUUCGUACAUGCAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAU ..((((((....(((((((.((((((....)).)))).)))))))((((...(((((((((((((......)))))))))))))..)))).....))))))......... ( -32.80) >DroSim_CAF1 3500 110 - 1 ACCCAUUUACUCGCUUGUACAUAAGUAGUUACUUUGUUUACGAGCAUUCGUACAUGCAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAU ..((((((....(((((((.((((((....)).)))).)))))))((((...(((((((((((((......)))))))))))))..)))).....))))))......... ( -32.80) >DroEre_CAF1 3678 110 - 1 ACCCAUUUACUCGCUUGUACAUAAGUAGUUACUUUGUUUACGAGCAUUUGUACAUACAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAU ..((((((....(((((((.((((((....)).)))).))))))).((((..(((((((((((((......))))))))))))))))).......))))))......... ( -31.50) >DroYak_CAF1 3720 110 - 1 ACCCAUUUACUCGCUUAUACAUAAGUAGUUACUUUGUUUACGAGCAUUCGUACAUGCAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAU ..((((((....((((..(((.((((....)))))))....))))((((...(((((((((((((......)))))))))))))..)))).....))))))......... ( -28.70) >consensus ACCCAUUUACUCGCUUGUACAUAAGUAGUUACUUUGUUUACGAGCAUUCGUACAUGCAUAUACAUACAUACAUGUGUAUGUAUGCAGAAUCAUGAAAAUGGUAUCCACAU ..((((((....(((((((.((((((....)).)))).)))))))((((...(((((((((((((......)))))))))))))..)))).....))))))......... (-31.40 = -30.88 + -0.52)

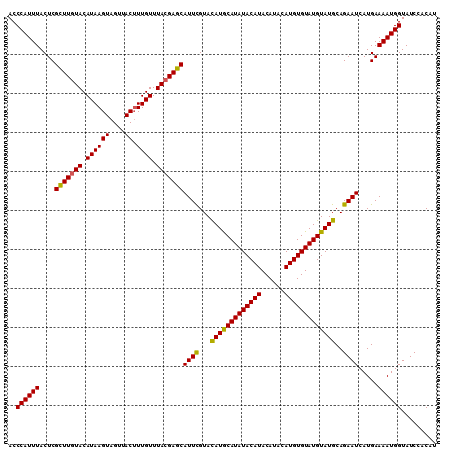
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:55:39 2006