
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 10,372,533 – 10,372,736 |
| Length | 203 |
| Max. P | 0.860996 |

| Location | 10,372,533 – 10,372,628 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 95 |
| Reading direction | forward |
| Mean pairwise identity | 92.77 |
| Mean single sequence MFE | -42.20 |
| Consensus MFE | -35.85 |
| Energy contribution | -37.52 |
| Covariance contribution | 1.67 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.28 |
| SVM RNA-class probability | 0.665693 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 10372533 95 + 22407834 CUGCACGACUGCGACGGCAGCUGAUUGGCCACACAGGUGGCAAAGCAGUCGCUGUUCGGAGCCGGAGAGGGCGGUGGCACGCCCCUAUUGGCAUC ........(((.((((((.((((....(((((....)))))....)))).))))))))).(((((((.(((((......))))))).)))))... ( -44.50) >DroSec_CAF1 3933 95 + 1 CUGCACGACUGCGACGGCAGCUGAUUGGCCACACAGGUGGCAAAGCAGUCGCUGUUCGGAGCCGGAGAGGGCGGUGGCACGCCCCUAUUGGCAUC ........(((.((((((.((((....(((((....)))))....)))).))))))))).(((((((.(((((......))))))).)))))... ( -44.50) >DroSim_CAF1 3939 95 + 1 CUGCACGACUGCGACGGCAGCUGAUUGGCCACACAGGUGGCAAAGCAGUCGCUGUUCGGAGCCGGAGAGGGCGGUGGCACGCCCCUAUUGGCAUC ........(((.((((((.((((....(((((....)))))....)))).))))))))).(((((((.(((((......))))))).)))))... ( -44.50) >DroEre_CAF1 4035 95 + 1 CUGCACGACUGAGAUGGCAGCUGAUUGGCCACACAGGUGGCAAAGCAGUCGCUAUUCGGAGCCGGAGAGGGCGGUGGCACGCCCCUAUUGGCAUC ........(((((.((((.((((....(((((....)))))....)))).))))))))).(((((((.(((((......))))))).)))))... ( -43.50) >DroYak_CAF1 4024 95 + 1 CUGCACGACUGAGACGGCAGCUGAUUGGCCACACAGGUGGCAAAGCAAUCGCUAUUCGGAGCCGGAGAGGGCGGUGGCACGCCCCUAUUGGCAUC ((((.((.......))))))((((...(((((....)))))..(((....)))..)))).(((((((.(((((......))))))).)))))... ( -37.40) >DroAna_CAF1 4561 93 + 1 --GCACGGCUGCGCGGGCAUCUGAUUGGCCACACAGGUGGCAAAGCAAUCACUGUUGGGUGCCGGCGAGGGAGGCGGUACGCCCCUGUUGGGAUC --...((((..(((.(((((((.....(((((....)))))..((((.....))))))))))).))).(((.((((...)))))))))))..... ( -38.80) >consensus CUGCACGACUGCGACGGCAGCUGAUUGGCCACACAGGUGGCAAAGCAGUCGCUGUUCGGAGCCGGAGAGGGCGGUGGCACGCCCCUAUUGGCAUC ........(((.((((((.((((....(((((....)))))....)))).))))))))).(((((((.(((((......))))))).)))))... (-35.85 = -37.52 + 1.67)

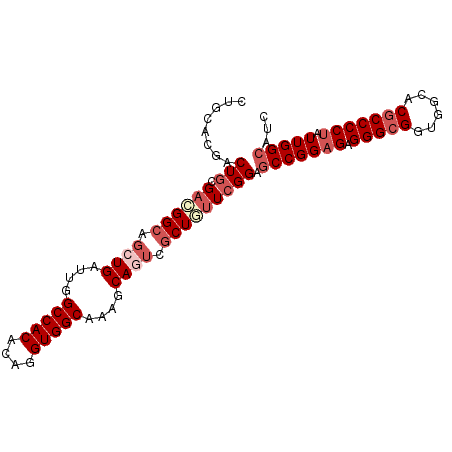

| Location | 10,372,557 – 10,372,668 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 93.03 |
| Mean single sequence MFE | -48.37 |
| Consensus MFE | -40.39 |
| Energy contribution | -41.87 |
| Covariance contribution | 1.48 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.21 |
| SVM RNA-class probability | 0.637624 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 10372557 111 + 22407834 UUGGCCACACAGGUGGCAAAGCAGUCGCUGUUCGGAGCCGGAGAGGGCGGUGGCACGCCCCUAUUGGCAUCGUCGUACUUCUCCAGCGGCGACAGCUUCAGCUUGCCGCCA ...........(((((((((((.(((((((((.((((((((((.(((((......))))))).)))))...(.....)...)))))))))))).))).....)))))))). ( -51.80) >DroSec_CAF1 3957 111 + 1 UUGGCCACACAGGUGGCAAAGCAGUCGCUGUUCGGAGCCGGAGAGGGCGGUGGCACGCCCCUAUUGGCAUCGUCGUACUUCUCCAGCGGCGACAGCUUCAGCUUGCCGCCA ...........(((((((((((.(((((((((.((((((((((.(((((......))))))).)))))...(.....)...)))))))))))).))).....)))))))). ( -51.80) >DroSim_CAF1 3963 110 + 1 UUGGCCACACAGGUGGCAAAGCAGUCGCUGUUCGGAGCCGGAGAGGGCGGUGGCACGCCCCUAUUGGCAUCGUCGUACUUCUCCAGCGGCGACAGCUUCAGCUUGCCG-GA ...(((((....)))))...((((((((((((.((((((((((.(((((......))))))).)))))...(.....)...))))))))))))(((....))))))..-.. ( -47.90) >DroEre_CAF1 4059 111 + 1 UUGGCCACACAGGUGGCAAAGCAGUCGCUAUUCGGAGCCGGAGAGGGCGGUGGCACGCCCCUAUUGGCAUCGUCGUACUUCUCCAGCGGCGAUAGCUUUAGCUUGCCGCCA ...........((((((((.((.(..(((((.((..((.((((((((((..((...(((......))).))..))).))))))).))..)))))))..).)))))))))). ( -48.40) >DroYak_CAF1 4048 111 + 1 UUGGCCACACAGGUGGCAAAGCAAUCGCUAUUCGGAGCCGGAGAGGGCGGUGGCACGCCCCUAUUGGCAUCGUCGUACUUCUCCAGCGGCGAUAACUUCAGCUUGCCGCCA ...........((((((((.((..............(((((((.(((((......))))))).)))))((((((((.........)))))))).......)))))))))). ( -46.20) >DroAna_CAF1 4583 111 + 1 UUGGCCACACAGGUGGCAAAGCAAUCACUGUUGGGUGCCGGCGAGGGAGGCGGUACGCCCCUGUUGGGAUCAUCGUAUUUCUCCAACGGCGACAGCUUAAGCUUGCCGGAA ...(((((....)))))........((((....))))((((((((..((((.((.((((...(((((((..........)).))))))))))).))))...)))))))).. ( -44.10) >consensus UUGGCCACACAGGUGGCAAAGCAGUCGCUGUUCGGAGCCGGAGAGGGCGGUGGCACGCCCCUAUUGGCAUCGUCGUACUUCUCCAGCGGCGACAGCUUCAGCUUGCCGCCA ...........(((((((((((.(((((((((.((((((((((.(((((......))))))).)))))...(.....)...)))))))))))).))).....)))))))). (-40.39 = -41.87 + 1.48)

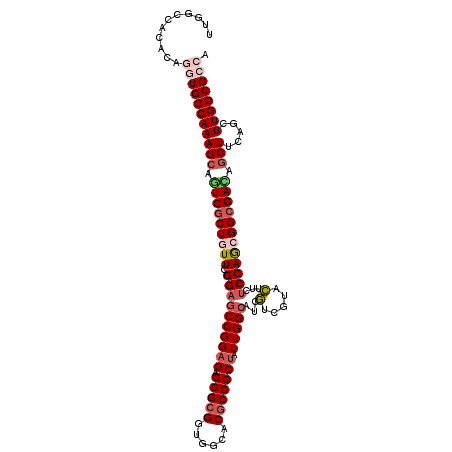

| Location | 10,372,557 – 10,372,668 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 93.03 |
| Mean single sequence MFE | -44.68 |
| Consensus MFE | -39.31 |
| Energy contribution | -39.28 |
| Covariance contribution | -0.03 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.38 |
| Structure conservation index | 0.88 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.660191 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 10372557 111 - 22407834 UGGCGGCAAGCUGAAGCUGUCGCCGCUGGAGAAGUACGACGAUGCCAAUAGGGGCGUGCCACCGCCCUCUCCGGCUCCGAACAGCGACUGCUUUGCCACCUGUGUGGCCAA (((((((((((.(..(((((((..((((((((.(((......)))......(((((......)))))))))))))..)).)))))..).)).)))))..(.....))))). ( -48.30) >DroSec_CAF1 3957 111 - 1 UGGCGGCAAGCUGAAGCUGUCGCCGCUGGAGAAGUACGACGAUGCCAAUAGGGGCGUGCCACCGCCCUCUCCGGCUCCGAACAGCGACUGCUUUGCCACCUGUGUGGCCAA (((((((((((.(..(((((((..((((((((.(((......)))......(((((......)))))))))))))..)).)))))..).)).)))))..(.....))))). ( -48.30) >DroSim_CAF1 3963 110 - 1 UC-CGGCAAGCUGAAGCUGUCGCCGCUGGAGAAGUACGACGAUGCCAAUAGGGGCGUGCCACCGCCCUCUCCGGCUCCGAACAGCGACUGCUUUGCCACCUGUGUGGCCAA ..-.(((((((.(..(((((((..((((((((.(((......)))......(((((......)))))))))))))..)).)))))..).)).))))).((.....)).... ( -44.70) >DroEre_CAF1 4059 111 - 1 UGGCGGCAAGCUAAAGCUAUCGCCGCUGGAGAAGUACGACGAUGCCAAUAGGGGCGUGCCACCGCCCUCUCCGGCUCCGAAUAGCGACUGCUUUGCCACCUGUGUGGCCAA (((((((((((....(((((((..((((((((.(((......)))......(((((......)))))))))))))..)).)))))....)).)))))..(.....))))). ( -46.10) >DroYak_CAF1 4048 111 - 1 UGGCGGCAAGCUGAAGUUAUCGCCGCUGGAGAAGUACGACGAUGCCAAUAGGGGCGUGCCACCGCCCUCUCCGGCUCCGAAUAGCGAUUGCUUUGCCACCUGUGUGGCCAA (((((((((((....(((((((..((((((((.(((......)))......(((((......)))))))))))))..)).)))))....)).)))))..(.....))))). ( -43.70) >DroAna_CAF1 4583 111 - 1 UUCCGGCAAGCUUAAGCUGUCGCCGUUGGAGAAAUACGAUGAUCCCAACAGGGGCGUACCGCCUCCCUCGCCGGCACCCAACAGUGAUUGCUUUGCCACCUGUGUGGCCAA ....(((..((..((((.(((((.(((((.........(((.((((....)))))))...(((.........)))..))))).))))).)))).))(((....)))))).. ( -37.00) >consensus UGGCGGCAAGCUGAAGCUGUCGCCGCUGGAGAAGUACGACGAUGCCAAUAGGGGCGUGCCACCGCCCUCUCCGGCUCCGAACAGCGACUGCUUUGCCACCUGUGUGGCCAA ....(((((((....(((((((..((((((((.(((......)))......(((((......)))))))))))))..)).)))))....)).))))).((.....)).... (-39.31 = -39.28 + -0.03)

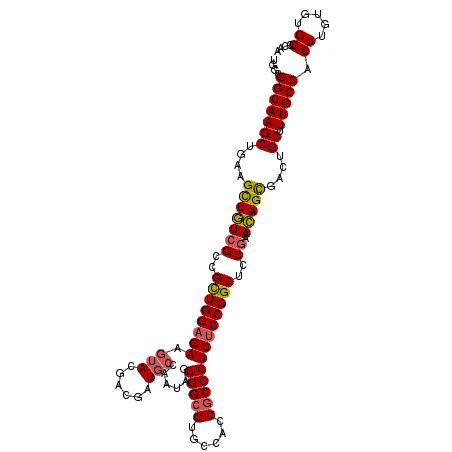
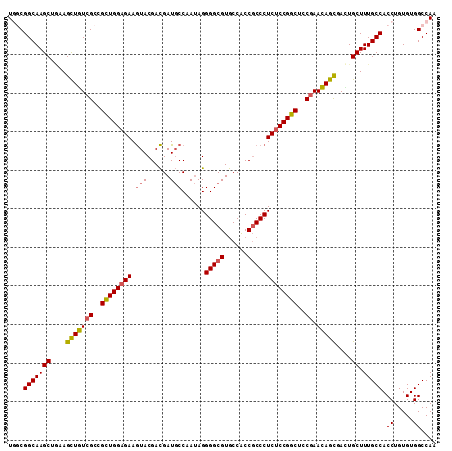
| Location | 10,372,628 – 10,372,736 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 79.63 |
| Mean single sequence MFE | -44.97 |
| Consensus MFE | -19.58 |
| Energy contribution | -22.75 |
| Covariance contribution | 3.17 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.86 |
| Structure conservation index | 0.44 |
| SVM decision value | 0.83 |
| SVM RNA-class probability | 0.860996 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 10372628 108 + 22407834 GUCGUACUUCUCCAGCGGCGACAGCUUCAGCUUGCCGCCAUGUGGCGGGAAGUUCUCGCCGUUGGGAGCGCGAAAGCGGCCCGACGACAGGCUGCAGUACAUCACAUC ...(((((....(((((((((.((((((......(((((....))))))))))).)))))((((((..(((....))).)))))).....)))).)))))........ ( -53.10) >DroPse_CAF1 10663 108 + 1 GUCAAUCUUUUCCAGCGGCGACAGCUUCAGCUUUCCAGCGUUGGGCUGGAAAGCAUCACUAGACUGCGCAUUUAAGCGAGAAGAUUGCAAACUGCAGUACAUCACAUC (.((((((((((..((.(((.(((((...((((((((((.....))))))))))......)).))))))......))))))))))))).................... ( -37.20) >DroSim_CAF1 4034 107 + 1 GUCGUACUUCUCCAGCGGCGACAGCUUCAGCUUGCCG-GAUGUGGCGGGAAGUUCUCGCCGUUGGGAGCGCGAAAACGGCCCGACGACAGGCUGCAGUACAUCACAUC ...(((((((.((((((((((.((((((....(((((-....))))).)))))).)))))))))))).........(((((........))))).)))))........ ( -44.50) >DroEre_CAF1 4130 108 + 1 GUCGUACUUCUCCAGCGGCGAUAGCUUUAGCUUGCCGCCAUGUGGCGGGAAGUUAUCCCCGUUCGGAGCGCGAAAGCGGCCGGACGACAGGCUGCAGUACAUCACAUC ...(((((....((((((.((((((((...((((((((...))))))))))))))))))(((((((..(((....))).)))))))....)))).)))))........ ( -49.60) >DroYak_CAF1 4119 108 + 1 GUCGUACUUCUCCAGCGGCGAUAACUUCAGCUUGCCGCCAUGUGGCGGGAAGUUCUCGCCGUUAGGAGCGCGAAAGCGACCCGACGACAGGCUGCAGUACAUCACAUC (((((....((((((((((((.((((((......(((((....))))))))))).)))))))).))))(((....))).....))))).................... ( -48.20) >DroPer_CAF1 10516 108 + 1 GUCAAUCUUUUCCAGCGGCGACAGCUUCAGCUUUCCAGCGUUGGGCUGGAAAGCAUCACUAGACUGCGCAUUUAAGCGAGAAGAUUGCAAACUGCAGUACAUCACAUC (.((((((((((..((.(((.(((((...((((((((((.....))))))))))......)).))))))......))))))))))))).................... ( -37.20) >consensus GUCGUACUUCUCCAGCGGCGACAGCUUCAGCUUGCCGCCAUGUGGCGGGAAGUUAUCGCCGUUCGGAGCGCGAAAGCGACCCGACGACAGGCUGCAGUACAUCACAUC (((((....(((((((((.((.((((((....((((((...)))))).)))))).)).))))).))))(((....))).....))))).................... (-19.58 = -22.75 + 3.17)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:55:31 2006