
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 1,109,440 – 1,109,557 |
| Length | 117 |
| Max. P | 0.915932 |

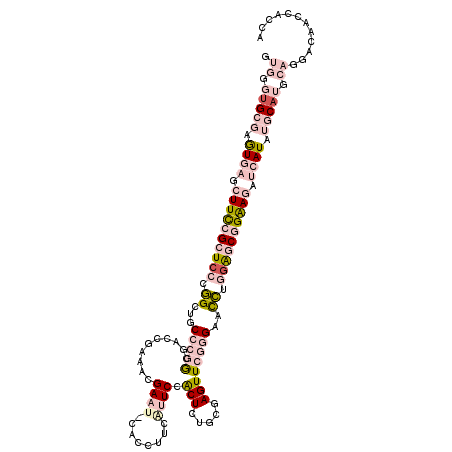
| Location | 1,109,440 – 1,109,557 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.85 |
| Mean single sequence MFE | -46.20 |
| Consensus MFE | -22.16 |
| Energy contribution | -25.88 |
| Covariance contribution | 3.73 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.11 |
| Structure conservation index | 0.48 |
| SVM decision value | 0.60 |
| SVM RNA-class probability | 0.794090 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
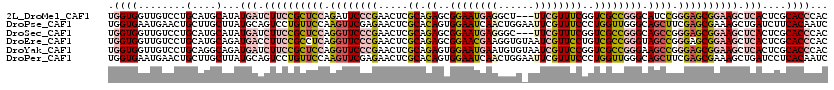
>2L_DroMel_CAF1 1109440 117 + 22407834 GUGGGUGCGAGUGAGCUUCCGCUCCCGGAUGCCCGGCGACCGAAACGAA---AGCCUCAUUCCGCUCUGCGAGUUCGGGAAUCUGGAGCGGAAGAUCAUAUGCAUGCAGGACAACCACCA .((.(((((.((((.((((((((((.((((.((((((....((..(...---.)..))....(((...))).).))))).)))))))))))))).)))).))))).))((....)).... ( -50.40) >DroPse_CAF1 38750 120 + 1 GAUUGUGAAGAUCAGCUUUCGCUCGAAGCUGCCCAACCAGGGAAACGAAUUCCAGUUGAUUCCACUGUGCGAGUUCUCGAACUUGGAACAGGACUGCAUAAGCAAGCAGUUCAUUCACCA ....(((((...(((((((.....)))))))..((((...((((.....)))).)))).(((((..((.(((....))).)).)))))..(((((((........))))))).))))).. ( -35.60) >DroSec_CAF1 27986 117 + 1 GUGGGUGCGAGUGAGCUUCCGCUCCCGGCUGCCCGGCGACCGAAACGAA---GCCCUCAUUCCGCUCUGCGAGUUCGGGAACCUGGAGCGGAAGAUCAUAUGCAUGCAGGACAACCACCA .((.(((((.((((.((((((((((.((...((((((..(......)..---))).......(((...))).....)))..)).)))))))))).)))).))))).))((....)).... ( -48.70) >DroEre_CAF1 30774 120 + 1 GUGGGUGCGAGUGAGCUUCCGCUCCCGGCUACCCGGCGACAGGAACGAUUACACCUUCGUUCCGCUCUGCGAGUUCGGGAACCUGAGGCGGAAGGUCAUCUGCAUGCAGGACAACCACCA .((.(((((..(((.(((((((..(.((...(((((.....(((((((........)))))))((((...)))))))))..)).)..))))))).)))..))))).))((....)).... ( -52.90) >DroYak_CAF1 29051 120 + 1 GUGGGUGCGAGUGAGCUUCCGCUCCCGGCUUCCCGGCGACCGGAACGAUUACACAUUCAUUCCACUCUGCGAGUUCGGGAACCUGGAGCGGAAGAUCAUCUGCCUGCAGGACAACCACCA (..((((.((.(((.((((((((((.((.(((((((.....((((.((........)).))))((((...))))))))))))).)))))))))).)))))))))..).((....)).... ( -53.00) >DroPer_CAF1 38668 120 + 1 GAUUGUGAGGAUCAGCUUUCGCUCGAAGCUGCCCAACCAGGGAAACGAAUUCCAGUUGAUUCCACUGUGCGAGUUCUCGAACUUGGAACAGGACUGCAUAAGCAAGCAGUUCAUUCACCA ....(((((((.(((((((.....)))))))..((((...((((.....)))).))))..))).((((.((((((....))))))..))))((((((........))))))...)))).. ( -36.60) >consensus GUGGGUGCGAGUGAGCUUCCGCUCCCGGCUGCCCGGCGACCGAAACGAAU_CACCUUCAUUCCACUCUGCGAGUUCGGGAACCUGGAGCGGAAGAUCAUAUGCAUGCAGGACAACCACCA .((.(((((.((((.((((((((((.((...(((((..........((((........)))).(((.....))))))))..)).)))))))))).)))).))))).))............ (-22.16 = -25.88 + 3.73)

| Location | 1,109,440 – 1,109,557 |
|---|---|
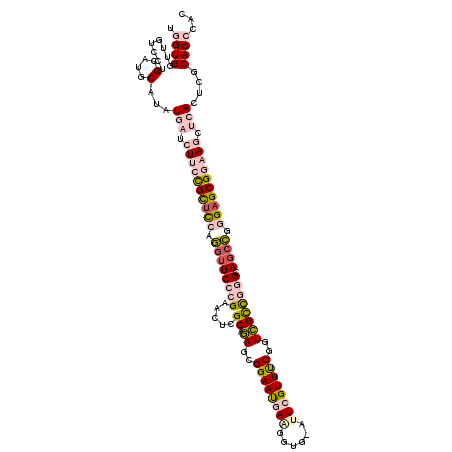
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.85 |
| Mean single sequence MFE | -47.10 |
| Consensus MFE | -29.22 |
| Energy contribution | -31.00 |
| Covariance contribution | 1.78 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.62 |
| SVM decision value | 1.10 |
| SVM RNA-class probability | 0.915932 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 1109440 117 - 22407834 UGGUGGUUGUCCUGCAUGCAUAUGAUCUUCCGCUCCAGAUUCCCGAACUCGCAGAGCGGAAUGAGGCU---UUCGUUUCGGUCGCCGGGCAUCCGGGAGCGGAAGCUCACUCGCACCCAC ..((((.((.....))(((...(((.((((((((((.(((.((((.....((.((.(((((((((...---))))))))).)))))))).)))..)))))))))).)))...))).)))) ( -48.60) >DroPse_CAF1 38750 120 - 1 UGGUGAAUGAACUGCUUGCUUAUGCAGUCCUGUUCCAAGUUCGAGAACUCGCACAGUGGAAUCAACUGGAAUUCGUUUCCCUGGUUGGGCAGCUUCGAGCGAAAGCUGAUCUUCACAAUC ..(((((((((((((((((....))).....((((((.((.(((....))).))..))))))(((((((((.....))))..)))))))))).))))(((....)))....))))).... ( -37.90) >DroSec_CAF1 27986 117 - 1 UGGUGGUUGUCCUGCAUGCAUAUGAUCUUCCGCUCCAGGUUCCCGAACUCGCAGAGCGGAAUGAGGGC---UUCGUUUCGGUCGCCGGGCAGCCGGGAGCGGAAGCUCACUCGCACCCAC ..((((.((.....))(((...(((.((((((((((.((((((((.....((.((.(((((((((...---))))))))).)))))))).)))).)))))))))).)))...))).)))) ( -52.40) >DroEre_CAF1 30774 120 - 1 UGGUGGUUGUCCUGCAUGCAGAUGACCUUCCGCCUCAGGUUCCCGAACUCGCAGAGCGGAACGAAGGUGUAAUCGUUCCUGUCGCCGGGUAGCCGGGAGCGGAAGCUCACUCGCACCCAC ..((((.(((.(((....))).(((.((((((((((.((((((((.....((.((..(((((((........)))))))..)))))))).))))))).))))))).)))...))).)))) ( -49.40) >DroYak_CAF1 29051 120 - 1 UGGUGGUUGUCCUGCAGGCAGAUGAUCUUCCGCUCCAGGUUCCCGAACUCGCAGAGUGGAAUGAAUGUGUAAUCGUUCCGGUCGCCGGGAAGCCGGGAGCGGAAGCUCACUCGCACCCAC ..((((.(((.(((....))).(((.((((((((((.((((((((.....((.((.((((((((........)))))))).)))))))).)))).)))))))))).)))...))).)))) ( -57.10) >DroPer_CAF1 38668 120 - 1 UGGUGAAUGAACUGCUUGCUUAUGCAGUCCUGUUCCAAGUUCGAGAACUCGCACAGUGGAAUCAACUGGAAUUCGUUUCCCUGGUUGGGCAGCUUCGAGCGAAAGCUGAUCCUCACAAUC ..((((.((((((((((((....))).....((((((.((.(((....))).))..))))))(((((((((.....))))..)))))))))).))))(((....))).....)))).... ( -37.20) >consensus UGGUGGUUGUCCUGCAUGCAUAUGAUCUUCCGCUCCAGGUUCCCGAACUCGCAGAGCGGAAUGAAGGUG_AUUCGUUUCGGUCGCCGGGCAGCCGGGAGCGGAAGCUCACUCGCACCCAC .((((........(....)...(((.((((((((((.((((((((.....((.((..((((((((......))))))))..)))))))).)))).)))))))))).)))....))))... (-29.22 = -31.00 + 1.78)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:33:47 2006