
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 9,846,017 – 9,846,115 |
| Length | 98 |
| Max. P | 0.980781 |

| Location | 9,846,017 – 9,846,115 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 78.99 |
| Mean single sequence MFE | -38.33 |
| Consensus MFE | -26.86 |
| Energy contribution | -27.12 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.10 |
| Mean z-score | -4.55 |
| Structure conservation index | 0.70 |
| SVM decision value | 1.87 |
| SVM RNA-class probability | 0.980781 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
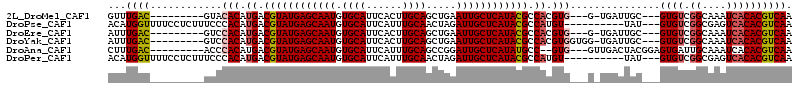
>2L_DroMel_CAF1 9846017 98 + 22407834 GUUUGAC---------GUACACAUGACGUAUGAGCAAUGUGCAUUCACUUGCAGCUGAAUUGCUCAUACGCCACGUG---G-UGAUUGC---GUGUCGGCAAAUCACACGUCAA ..(((((---------((.(((.((.((((((((((((.((((......)))).....)))))))))))).)).)))---(-(((((((---......)).))))))))))))) ( -42.90) >DroPse_CAF1 57792 101 + 1 ACAUGGUUUUCCUCUUUCCCACAUGACGUAUGAGCAAUGUGCAUUCAUUUGCAACUAGAUUGCUCAUACGCCAUGU----------UAU---GUGUCGGCGAGUCACACGUCAA ((((((....))........(((((.((((((((((((.((((......)))).....)))))))))))).)))))----------.))---))...((((.......)))).. ( -32.50) >DroEre_CAF1 57315 98 + 1 AUUUGAC---------GUCCACAUGACGUAUGAGCAAUGUGCAUUCACUUGCAGCUGAAUUGCUCAUACGCCACGUG---G-UGAUUGC---GUGUCGGCAAAUCACACGUCAA ..(((((---------((.(((.((.((((((((((((.((((......)))).....)))))))))))).)).)))---(-(((((((---......)).))))))))))))) ( -42.10) >DroYak_CAF1 57582 101 + 1 AUUUGAC---------GUCCACAUGACGUAUGAGCAAUGUGCAUUCACUUGCAGCUGAAUUGCUCAUACGCCACGUGGUGG-UGAUUGC---GUGUCGGCAAAUCACACGUCAA ..(((((---------((((((.((.((((((((((((.((((......)))).....)))))))))))).)).))))..(-(((((((---......)).))))))))))))) ( -44.50) >DroAna_CAF1 61189 100 + 1 CUUUGAC---------ACCCACAUGACGUAUGAGCAAUGUGCAUUCAUUUGCAGCCGGAUUGCUCAUAUGCC--GUG---GUUGACUACGGAGUGAUUGCAAAUCACACGUCAA ..(((((---------..........(((((((((((((((((......)))).)...))))))))))))((--(((---(....)))))).(((((.....)))))..))))) ( -35.50) >DroPer_CAF1 58318 101 + 1 ACAUGGUUUUCCUCUUUCCCACAUGACGUAUGAGCAAUGUGCAUUCAUUUGCAACUAGAUUGCUCAUACGCCAUGU----------UAU---GUGUCGGCGAGUCACACGUCAA ((((((....))........(((((.((((((((((((.((((......)))).....)))))))))))).)))))----------.))---))...((((.......)))).. ( -32.50) >consensus AUUUGAC_________GCCCACAUGACGUAUGAGCAAUGUGCAUUCACUUGCAGCUGAAUUGCUCAUACGCCACGUG___G_UGAUUAC___GUGUCGGCAAAUCACACGUCAA ...((((............(((.((.((((((((((((.((((......)))).....)))))))))))).)).)))...............((((.((....)))))))))). (-26.86 = -27.12 + 0.25)

| Location | 9,846,017 – 9,846,115 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 78.99 |
| Mean single sequence MFE | -42.08 |
| Consensus MFE | -31.27 |
| Energy contribution | -32.77 |
| Covariance contribution | 1.50 |
| Combinations/Pair | 1.03 |
| Mean z-score | -5.44 |
| Structure conservation index | 0.74 |
| SVM decision value | 1.58 |
| SVM RNA-class probability | 0.965181 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 9846017 98 - 22407834 UUGACGUGUGAUUUGCCGACAC---GCAAUCA-C---CACGUGGCGUAUGAGCAAUUCAGCUGCAAGUGAAUGCACAUUGCUCAUACGUCAUGUGUAC---------GUCAAAC (((((((((((((.((......---)))))))-)---(((((((((((((((((((...(.((((......)))))))))))))))))))))))).))---------))))).. ( -47.40) >DroPse_CAF1 57792 101 - 1 UUGACGUGUGACUCGCCGACAC---AUA----------ACAUGGCGUAUGAGCAAUCUAGUUGCAAAUGAAUGCACAUUGCUCAUACGUCAUGUGGGAAAGAGGAAAACCAUGU ...((((((..(((.((.....---...----------((((((((((((((((((...((.(((......))))))))))))))))))))))).))...)))..)...))))) ( -36.30) >DroEre_CAF1 57315 98 - 1 UUGACGUGUGAUUUGCCGACAC---GCAAUCA-C---CACGUGGCGUAUGAGCAAUUCAGCUGCAAGUGAAUGCACAUUGCUCAUACGUCAUGUGGAC---------GUCAAAU (((((((.(((((.((......---)))))))-(---(((((((((((((((((((...(.((((......)))))))))))))))))))))))))))---------))))).. ( -48.80) >DroYak_CAF1 57582 101 - 1 UUGACGUGUGAUUUGCCGACAC---GCAAUCA-CCACCACGUGGCGUAUGAGCAAUUCAGCUGCAAGUGAAUGCACAUUGCUCAUACGUCAUGUGGAC---------GUCAAAU (((((((((((((.((......---)))))))-)..((((((((((((((((((((...(.((((......)))))))))))))))))))))))))))---------))))).. ( -51.60) >DroAna_CAF1 61189 100 - 1 UUGACGUGUGAUUUGCAAUCACUCCGUAGUCAAC---CAC--GGCAUAUGAGCAAUCCGGCUGCAAAUGAAUGCACAUUGCUCAUACGUCAUGUGGGU---------GUCAAAG ((((((.(((((.....))))).).........(---(((--(((.((((((((((...(.((((......))))))))))))))).)))..))))..---------))))).. ( -32.10) >DroPer_CAF1 58318 101 - 1 UUGACGUGUGACUCGCCGACAC---AUA----------ACAUGGCGUAUGAGCAAUCUAGUUGCAAAUGAAUGCACAUUGCUCAUACGUCAUGUGGGAAAGAGGAAAACCAUGU ...((((((..(((.((.....---...----------((((((((((((((((((...((.(((......))))))))))))))))))))))).))...)))..)...))))) ( -36.30) >consensus UUGACGUGUGAUUUGCCGACAC___GCAAUCA_C___CACGUGGCGUAUGAGCAAUCCAGCUGCAAAUGAAUGCACAUUGCUCAUACGUCAUGUGGAC_________GUCAAAU .((((((((.........))))...............(((((((((((((((((((...(.((((......))))))))))))))))))))))))............))))... (-31.27 = -32.77 + 1.50)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:51:37 2006