
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 9,669,365 – 9,669,495 |
| Length | 130 |
| Max. P | 0.999845 |

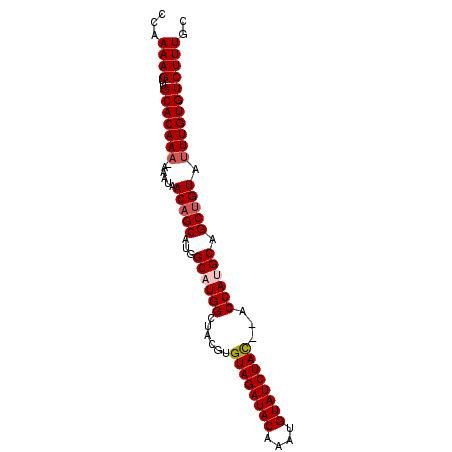
| Location | 9,669,365 – 9,669,456 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 91 |
| Reading direction | forward |
| Mean pairwise identity | 95.74 |
| Mean single sequence MFE | -32.00 |
| Consensus MFE | -27.94 |
| Energy contribution | -28.58 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.03 |
| Mean z-score | -5.20 |
| Structure conservation index | 0.87 |
| SVM decision value | 3.13 |
| SVM RNA-class probability | 0.998540 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 9669365 91 + 22407834 CCAAAAGUUGCACAAAAAAAUAACAGCACGGCAUGGCUACGUGUAGAUACAAAUGUAUCUAUCAGCCAUGCAGCUGUAUUUGUGUCUUUGC ...((((..(((((((......(((((...((((((((....((((((((....)))))))).)))))))).))))).))))))))))).. ( -34.70) >DroSec_CAF1 27640 88 + 1 CCAAAAGUUGCACAAA-AAAUAACAGCAUGGCAUGGCUACGUGUAGAUACAAAUGUAUCUAC--ACCAUGCAGCUGUAUUUGUGUCUUUGC ...((((..(((((((-.....(((((...((((((.....(((((((((....))))))))--))))))).))))).))))))))))).. ( -33.00) >DroSim_CAF1 28669 88 + 1 CCAAAAGUUGCACAAA-AAAUAACAGCAUGGCAUGGCUACGUGUAGAUACAAAUGUAUCUAC--ACCAUGCAGCUGUAUUUGUGUCUUUGC ...((((..(((((((-.....(((((...((((((.....(((((((((....))))))))--))))))).))))).))))))))))).. ( -33.00) >DroEre_CAF1 33230 88 + 1 CCAAAAGUUGCACAAA-AAAUAACUGCAUGGCAUGGCUACGUGUAGAUACAAAUGUAUCUAC--ACCAGGCAGCUGUAUUUGUGUCUUUGC ...((((..(((((((-..(((.((((.((((...)))).((((((((((....))))))))--))...)))).))).))))))))))).. ( -29.20) >DroYak_CAF1 27873 88 + 1 CCAAAAGUUGCACAAA-AAAUAACAGCAUGGCAUGGCUACGUGUAGAUACAAAUGUAUCUAC--ACCAGGCAGCUGUAGUUGUGUCUUUGC ................-........(((.((((..(((((((((((((((....))))))))--)).((....)))))))..))))..))) ( -30.10) >consensus CCAAAAGUUGCACAAA_AAAUAACAGCAUGGCAUGGCUACGUGUAGAUACAAAUGUAUCUAC__ACCAUGCAGCUGUAUUUGUGUCUUUGC ...((((..(((((((......(((((...((((((......((((((((....))))))))...)))))).))))).))))))))))).. (-27.94 = -28.58 + 0.64)

| Location | 9,669,365 – 9,669,456 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 91 |
| Reading direction | reverse |
| Mean pairwise identity | 95.74 |
| Mean single sequence MFE | -31.48 |
| Consensus MFE | -29.14 |
| Energy contribution | -29.98 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.03 |
| Mean z-score | -4.65 |
| Structure conservation index | 0.93 |
| SVM decision value | 4.24 |
| SVM RNA-class probability | 0.999845 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 9669365 91 - 22407834 GCAAAGACACAAAUACAGCUGCAUGGCUGAUAGAUACAUUUGUAUCUACACGUAGCCAUGCCGUGCUGUUAUUUUUUUGUGCAACUUUUGG .(((((.((((((.(((((.(((((((((.(((((((....)))))))....)))))))))...)))))......))))))....))))). ( -34.80) >DroSec_CAF1 27640 88 - 1 GCAAAGACACAAAUACAGCUGCAUGGU--GUAGAUACAUUUGUAUCUACACGUAGCCAUGCCAUGCUGUUAUUU-UUUGUGCAACUUUUGG .(((((.((((((.(((((.(((((((--((((((((....)))))))).....)))))))...))))).....-))))))....))))). ( -32.80) >DroSim_CAF1 28669 88 - 1 GCAAAGACACAAAUACAGCUGCAUGGU--GUAGAUACAUUUGUAUCUACACGUAGCCAUGCCAUGCUGUUAUUU-UUUGUGCAACUUUUGG .(((((.((((((.(((((.(((((((--((((((((....)))))))).....)))))))...))))).....-))))))....))))). ( -32.80) >DroEre_CAF1 33230 88 - 1 GCAAAGACACAAAUACAGCUGCCUGGU--GUAGAUACAUUUGUAUCUACACGUAGCCAUGCCAUGCAGUUAUUU-UUUGUGCAACUUUUGG .(((((.((((((...((((((.((((--((((((((....))))))))))(((....))))).))))))....-))))))....))))). ( -30.40) >DroYak_CAF1 27873 88 - 1 GCAAAGACACAACUACAGCUGCCUGGU--GUAGAUACAUUUGUAUCUACACGUAGCCAUGCCAUGCUGUUAUUU-UUUGUGCAACUUUUGG .(((((.(((((..(((((.((.((((--((((((((....)))))))).....)))).))...))))).....-.)))))....))))). ( -26.60) >consensus GCAAAGACACAAAUACAGCUGCAUGGU__GUAGAUACAUUUGUAUCUACACGUAGCCAUGCCAUGCUGUUAUUU_UUUGUGCAACUUUUGG .(((((.((((((.(((((.(((((((..((((((((....)))))))).....)))))))...)))))......))))))....))))). (-29.14 = -29.98 + 0.84)


| Location | 9,669,382 – 9,669,495 |
|---|---|
| Length | 113 |
| Sequences | 5 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 90.94 |
| Mean single sequence MFE | -36.38 |
| Consensus MFE | -29.34 |
| Energy contribution | -29.70 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.10 |
| Mean z-score | -3.99 |
| Structure conservation index | 0.81 |
| SVM decision value | 3.40 |
| SVM RNA-class probability | 0.999142 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 9669382 113 + 22407834 AAAUAACAGCACGGCAUGGCUACGUGUAGAUACAAAUGUAUCUAUCAGCCAUGCAGCUGUAUUUGUGUCUUUGCGAAUGUCUAGCUGCCUUUUGUGCACGAAAAAAAAAAACA ........(((((((((((((....((((((((....)))))))).))))))))((((((((((((......)))))))..)))))......)))))................ ( -38.20) >DroSec_CAF1 27656 107 + 1 AAAUAACAGCAUGGCAUGGCUACGUGUAGAUACAAAUGUAUCUAC--ACCAUGCAGCUGUAUUUGUGUCUUUGCGAAUGUCUAGCUGUCU--UGUGCACGAAAAAA--AAAGA ........((((.((((((.....(((((((((....))))))))--)))))))((((((((((((......)))))))..)))))....--.)))).........--..... ( -33.50) >DroSim_CAF1 28685 108 + 1 AAAUAACAGCAUGGCAUGGCUACGUGUAGAUACAAAUGUAUCUAC--ACCAUGCAGCUGUAUUUGUGUCUUUGCGAAUGUCUAGCUGUCU--UGUGCACGAAAAAA-AAAACA ........((((.((((((.....(((((((((....))))))))--)))))))((((((((((((......)))))))..)))))....--.)))).........-...... ( -33.50) >DroEre_CAF1 33246 111 + 1 AAAUAACUGCAUGGCAUGGCUACGUGUAGAUACAAAUGUAUCUAC--ACCAGGCAGCUGUAUUUGUGUCUUUGCGAGUGUCUAGCUGUCGCUUGUGCACGAACAAAAAUCACA .......((((((((........((((((((((....))))))))--))..(((((((((((((((......)))))))..)))))))))).))))))............... ( -38.40) >DroYak_CAF1 27889 111 + 1 AAAUAACAGCAUGGCAUGGCUACGUGUAGAUACAAAUGUAUCUAC--ACCAGGCAGCUGUAGUUGUGUCUUUGCGAAUGUCUAGCUGCCGCUUGAGCACGGAAAAAAAAGACA ........(((.((((..(((((((((((((((....))))))))--)).((....)))))))..))))..)))...(((((.....(((........))).......))))) ( -38.30) >consensus AAAUAACAGCAUGGCAUGGCUACGUGUAGAUACAAAUGUAUCUAC__ACCAUGCAGCUGUAUUUGUGUCUUUGCGAAUGUCUAGCUGUCU_UUGUGCACGAAAAAAAAAAACA ........((((.((((((......((((((((....))))))))...))))))((((((((((((......)))))))..))))).......))))................ (-29.34 = -29.70 + 0.36)

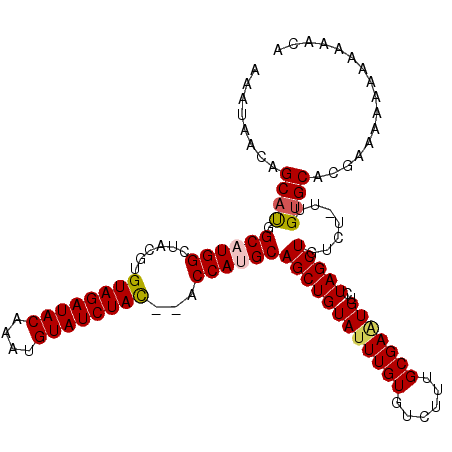
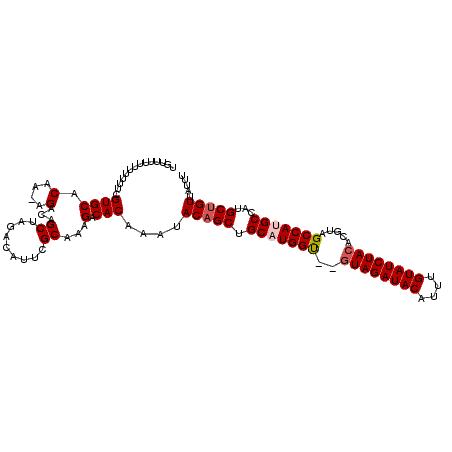
| Location | 9,669,382 – 9,669,495 |
|---|---|
| Length | 113 |
| Sequences | 5 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 90.94 |
| Mean single sequence MFE | -32.76 |
| Consensus MFE | -24.90 |
| Energy contribution | -25.54 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.04 |
| Mean z-score | -3.25 |
| Structure conservation index | 0.76 |
| SVM decision value | 2.13 |
| SVM RNA-class probability | 0.988668 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 9669382 113 - 22407834 UGUUUUUUUUUUUCGUGCACAAAAGGCAGCUAGACAUUCGCAAAGACACAAAUACAGCUGCAUGGCUGAUAGAUACAUUUGUAUCUACACGUAGCCAUGCCGUGCUGUUAUUU (((((((........(((.......)))((.........))))))))).(((((((((.(((((((((.(((((((....)))))))....)))))))))...)))).))))) ( -34.60) >DroSec_CAF1 27656 107 - 1 UCUUU--UUUUUUCGUGCACA--AGACAGCUAGACAUUCGCAAAGACACAAAUACAGCUGCAUGGU--GUAGAUACAUUUGUAUCUACACGUAGCCAUGCCAUGCUGUUAUUU (((((--.....((..((...--.....))..)).......)))))...(((((((((.(((((((--((((((((....)))))))).....)))))))...)))).))))) ( -28.40) >DroSim_CAF1 28685 108 - 1 UGUUUU-UUUUUUCGUGCACA--AGACAGCUAGACAUUCGCAAAGACACAAAUACAGCUGCAUGGU--GUAGAUACAUUUGUAUCUACACGUAGCCAUGCCAUGCUGUUAUUU ((((((-(....((..((...--.....))..)).......))))))).(((((((((.(((((((--((((((((....)))))))).....)))))))...)))).))))) ( -29.70) >DroEre_CAF1 33246 111 - 1 UGUGAUUUUUGUUCGUGCACAAGCGACAGCUAGACACUCGCAAAGACACAAAUACAGCUGCCUGGU--GUAGAUACAUUUGUAUCUACACGUAGCCAUGCCAUGCAGUUAUUU ((((.(((((((..(((..(.(((....))).).)))..))))))))))).....((((((.((((--((((((((....))))))))))(((....))))).)))))).... ( -38.60) >DroYak_CAF1 27889 111 - 1 UGUCUUUUUUUUCCGUGCUCAAGCGGCAGCUAGACAUUCGCAAAGACACAACUACAGCUGCCUGGU--GUAGAUACAUUUGUAUCUACACGUAGCCAUGCCAUGCUGUUAUUU (((((((.......(((.((.(((....))).)))))....))))))).....(((((.((.((((--((((((((....)))))))).....)))).))...)))))..... ( -32.50) >consensus UGUUUUUUUUUUUCGUGCACAA_AGACAGCUAGACAUUCGCAAAGACACAAAUACAGCUGCAUGGU__GUAGAUACAUUUGUAUCUACACGUAGCCAUGCCAUGCUGUUAUUU ..............((((.(....)...((.........))...).)))....(((((.(((((((..((((((((....)))))))).....)))))))...)))))..... (-24.90 = -25.54 + 0.64)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:50:10 2006