
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 9,291,493 – 9,291,631 |
| Length | 138 |
| Max. P | 0.920751 |

| Location | 9,291,493 – 9,291,594 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 75.60 |
| Mean single sequence MFE | -41.71 |
| Consensus MFE | -29.59 |
| Energy contribution | -30.12 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.71 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.920751 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
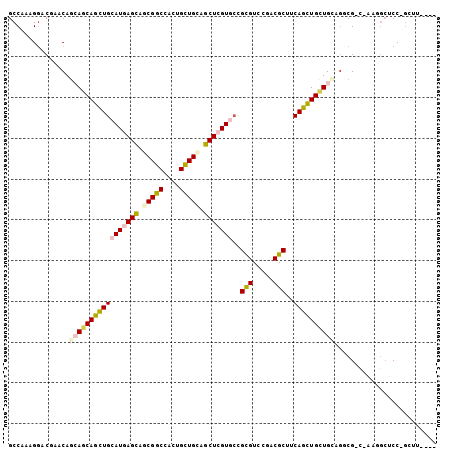
>2L_DroMel_CAF1 9291493 101 - 22407834 GCCAAAGGACGAACAGGAGCAGCUGCAUCAGUAGCGGCCAUUGCUGCAGCUCGUACCGCGUCCUUCGCACCAGUUGCUGCAGACGUUGAAUGCUCC-GCUUG--- ...............(((((((((((.......)))))((.(((((((((.......(((.....))).......))))))).)).))..))))))-.....--- ( -35.64) >DroVir_CAF1 182278 92 - 1 GCCACAGGACGAGCACCACCAGCUGCAUGAGCAGCGGCCACUGUUGUAGCUCGUGGCGUGUGCGACGCUUCAGUUGCUGGAGGCG---------CC-CCUUG--- ....((((..(.((.((.(((((..(((((((.(((((....))))).)))))))((((.....)))).......))))).)).)---------))-.))))--- ( -36.40) >DroPse_CAF1 143205 98 - 1 GCCAGAGGACCAGCAGCAGCAGCUGCAUGAGCAUCGGCCACUGCUGCAGCUCGUGCUGCGUGCGACGCUUCAGCUGUUGGAGGCGACUCGGGCGCC-GG------ (((.(((..((((((((.(((((.((((((((..((((....))))..)))))))).)).)))((....)).))))))))......))).)))...-..------ ( -46.90) >DroYak_CAF1 129067 101 - 1 GCCAAAGCACGAACAGGAGCAGCUGCAUCAGCAGCGGCCACUGCUGCAGCUCGUGCCGCGUCCUUCGCACCAGUUGCUGCAGUCGGCGAACGCUCC-GCUUU--- ...(((((.......(((((.(((((....))))).((((((((.((((((.((((..........)))).)))))).))))).)))....)))))-)))))--- ( -47.71) >DroAna_CAF1 121482 102 - 1 GCCACAGGACAAACAGCAGCAGUUGCAUGAGCAGCGGCCAUUGCUGCAGCUCGUACCGUGUCCUUCGCUUCAGCUGCUGCAACCG---ACUGCUCCCGCCUAUCC .....(((.......((((((((((.((((((.(((((....))))).))))))...(((.....)))..))))))))))...((---........))))).... ( -36.70) >DroPer_CAF1 143765 98 - 1 GCCAGAGGACCAGCAGCAGCAGCUGCAUGAGCAUCGGCCACUGCUGCAGCUCGUGCUGCGUGCGACGCUUCAGCUGUUGGAGGCGACUCGGGCGCC-GG------ (((.(((..((((((((.(((((.((((((((..((((....))))..)))))))).)).)))((....)).))))))))......))).)))...-..------ ( -46.90) >consensus GCCAAAGGACGAACAGCAGCAGCUGCAUGAGCAGCGGCCACUGCUGCAGCUCGUGCCGCGUCCGACGCUUCAGCUGCUGCAGGCG_C_AAGGCUCC_GCUU____ ...............(((((((((((((((((.(((((....))))).)))))))..(((.....)))..))))))))))......................... (-29.59 = -30.12 + 0.53)


| Location | 9,291,514 – 9,291,631 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.95 |
| Mean single sequence MFE | -44.63 |
| Consensus MFE | -23.80 |
| Energy contribution | -23.42 |
| Covariance contribution | -0.38 |
| Combinations/Pair | 1.46 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.599465 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
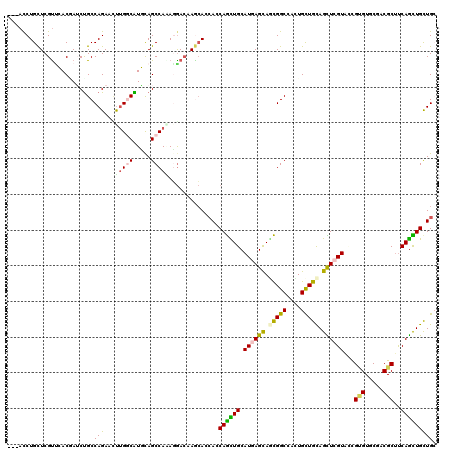
>2L_DroMel_CAF1 9291514 117 - 22407834 ---ACCUGUUCGUUGACCAGCUGCCAGAACUUGGCAUUUAGCCAAAGGACGAACAGGAGCAGCUGCAUCAGUAGCGGCCAUUGCUGCAGCUCGUACCGCGUCCUUCGCACCAGUUGCUGC ---.((((((((((.....((((((((...)))))....))).....))))))))))((((((((.((.(((.(((((....))))).))).))...(((.....)))..)))))))).. ( -46.40) >DroVir_CAF1 182290 120 - 1 AGGAUCUGCUCGUUCACGAUCUGCCAGAAUUUGCCAUGCAGCCACAGGACGAGCACCACCAGCUGCAUGAGCAGCGGCCACUGUUGUAGCUCGUGGCGUGUGCGACGCUUCAGUUGCUGG ......(((((((((.....((((.............)))).....)))))))))(((.(((((((((((((.(((((....))))).)))))))((((.....))))..)))))).))) ( -46.32) >DroWil_CAF1 225914 117 - 1 ---ACCUGCUCGUUCACGAUCUGCCAGAAUUUGCCAUGCAGCCAAAUAACAAGAACCACCAAUUGCAUUAGCAAUGGCCAUUGCUGCAGUUCGUAUUGGGUUCUUCGCUUCAAUUGCUGC ---..(((((((....)))...).)))..........(((((........(((((((((.(((((((...(((((....)))))))))))).))....)))))))..........))))) ( -27.47) >DroMoj_CAF1 223497 120 - 1 AGUAUCUGCUCGCGCACAAUCUGCCAGAACUUGGCGUGCAGCCAGACGAGCAGCACCACCAGCUGCAUGAGCAGCGGCCACUGUUGCAGUUCGUGACGUGUGCGACGCUUCAGCUGCUGC ((((.((((((((((((.............(((((.....)))))((((((.(((.((((.(((((....)))))))....)).))).))))))...)))))))).))...)).)))).. ( -45.80) >DroAna_CAF1 121504 117 - 1 ---ACCUGCUCGUUGACCAACUGCCAGAACUUGGCAUUCAGCCACAGGACAAACAGCAGCAGUUGCAUGAGCAGCGGCCAUUGCUGCAGCUCGUACCGUGUCCUUCGCUUCAGCUGCUGC ---.((((...(((((.....((((((...)))))).)))))..)))).......((((((((((.((((((.(((((....))))).))))))...(((.....)))..)))))))))) ( -47.70) >DroPer_CAF1 143783 117 - 1 ---ACCUGCUCGUCGACGAUCCGCCAGAAGCGGGCGUUCAGCCAGAGGACCAGCAGCAGCAGCUGCAUGAGCAUCGGCCACUGCUGCAGCUCGUGCUGCGUGCGACGCUUCAGCUGUUGG ---.(((.((.((.((((.(((((.....)))))))))..)).))))).((((((((.(((((.((((((((..((((....))))..)))))))).)).)))((....)).)))))))) ( -54.10) >consensus ___ACCUGCUCGUUCACGAUCUGCCAGAACUUGGCAUGCAGCCAAAGGACAAGCACCACCAGCUGCAUGAGCAGCGGCCACUGCUGCAGCUCGUACCGUGUGCGACGCUUCAGCUGCUGC ........................(((...(((((.....)))))..............((((((.((((((.(((((....))))).))))))...(((.....)))..))))))))). (-23.80 = -23.42 + -0.38)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:47:37 2006