
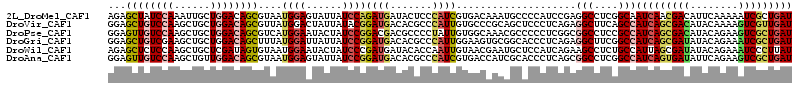
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 9,242,339 – 9,242,453 |
| Length | 114 |
| Max. P | 0.831901 |

| Location | 9,242,339 – 9,242,453 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 78.89 |
| Mean single sequence MFE | -37.48 |
| Consensus MFE | -22.62 |
| Energy contribution | -24.10 |
| Covariance contribution | 1.48 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.831901 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 9242339 114 + 22407834 AGAGCUAUCCAAAUUGCUGGACAGCGUAAUGGAGUAUUAUCCAGAUGAUACUCCCAUCGUGACAAAUGCCCCAUCCGAGGCCUCGGCAAUCAACGACAUUCAAAAAUCGCUGAU .......((((......))))(((((....((((((((((....))))))))))..((((......((((((......))....))))....))))...........))))).. ( -31.20) >DroVir_CAF1 116813 114 + 1 GGAGCUGUCCAAGCUGCUGGACAGCGUUAUGGACUAUUAUACGGAUGACACGCCCAUUGUGCCCGCAGCUCCCUCAGAGGCUUCAGCCAUCAGCGACAUACAAAAGUCGUUGAU (((((((((((.((((.....))))....))))).......(((..(.((((.....)))))))).))))))......(((....)))(((((((((........))))))))) ( -41.20) >DroPse_CAF1 87873 114 + 1 GGAGUUGUCCAAGCUGCUGGACAGCGUCAUGGAAUACUAUCCGGACGACGCCCCUAUUGUGGCAAACGCCCCCUCGGCGGCCUCCGCCAUCAGCGACAUACAGAAGUCGCUGAU ((.(((((((..((((.....)))).....(((......))))))))))(((.(....).)))........))..(((((...)))))(((((((((........))))))))) ( -47.80) >DroGri_CAF1 121734 114 + 1 GGAGCUGUCGAAGCUGCUGGACAGCUUUAUGGAUUAUUAUCCGGAUGACACGCCCAUUGGAAGUGCGGCACCCUCAGAGGCUUCGGCCAUCAGCGAUAUACAGAAAUCGCUGAU .(((.(((((((((((.....))))))).(((((....)))))...)))).((((((.....))).)))...)))...(((....)))(((((((((........))))))))) ( -44.30) >DroWil_CAF1 131421 114 + 1 AGAGCUCUCCAAGCUGCUCGAUAGUGUAAUGGAAUACUAUCCCGAUGAUACACCAAUUGUAACGAAUGCUCCAUCAGAAGCCUCUGCCAUUAGCGAUAUACAGAAAUCCCUUAU .((((..........))))((((((((......))))))))...............(((((...(.((((....((((....)))).....)))).).)))))........... ( -19.90) >DroAna_CAF1 72165 114 + 1 GGAGUUGUCCAAGCUGUUGGACAGCGUAAUGGAGUAUUAUCCGGAUGACACGCCCAUCGUGACCAUCGCACCCUCAGCGGCCUCGGCCAUCAGUGAUAUUCAGAAGUCGCUGAU ((.(((((((((....)))))))))((..((((......))))((((.((((.....))))..))))))..)).....(((....)))(((((((((........))))))))) ( -40.50) >consensus GGAGCUGUCCAAGCUGCUGGACAGCGUAAUGGAAUAUUAUCCGGAUGACACGCCCAUUGUGACAAACGCACCCUCAGAGGCCUCGGCCAUCAGCGACAUACAGAAAUCGCUGAU ...((((((((......))))))))....((((......))))((((.......))))....................(((....)))(((((((((........))))))))) (-22.62 = -24.10 + 1.48)
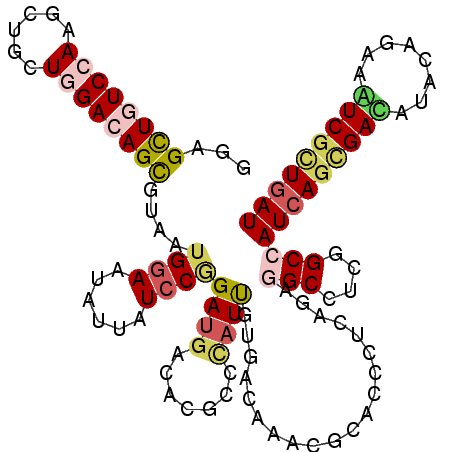
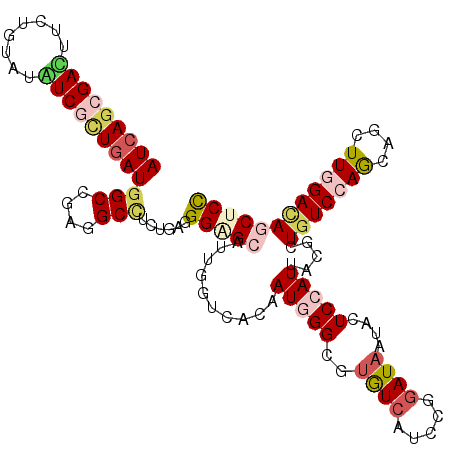
| Location | 9,242,339 – 9,242,453 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 78.89 |
| Mean single sequence MFE | -41.27 |
| Consensus MFE | -22.07 |
| Energy contribution | -23.55 |
| Covariance contribution | 1.48 |
| Combinations/Pair | 1.27 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.52 |
| SVM RNA-class probability | 0.768746 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 9242339 114 - 22407834 AUCAGCGAUUUUUGAAUGUCGUUGAUUGCCGAGGCCUCGGAUGGGGCAUUUGUCACGAUGGGAGUAUCAUCUGGAUAAUACUCCAUUACGCUGUCCAGCAAUUUGGAUAGCUCU (((((((((........)))))))))..((((....))))((((((...(((((..(((((.....)))))..)))))..))))))...(((((((((....)))))))))... ( -41.70) >DroVir_CAF1 116813 114 - 1 AUCAACGACUUUUGUAUGUCGCUGAUGGCUGAAGCCUCUGAGGGAGCUGCGGGCACAAUGGGCGUGUCAUCCGUAUAAUAGUCCAUAACGCUGUCCAGCAGCUUGGACAGCUCC ((((.((((........)))).))))(((....)))......((((((((((((((.......))))...))))......(((((....((((.....)))).))))))))))) ( -41.40) >DroPse_CAF1 87873 114 - 1 AUCAGCGACUUCUGUAUGUCGCUGAUGGCGGAGGCCGCCGAGGGGGCGUUUGCCACAAUAGGGGCGUCGUCCGGAUAGUAUUCCAUGACGCUGUCCAGCAGCUUGGACAACUCC (((((((((........)))))))))((((((.(((.(....).))).))))))......((((((((((..(((......))))))))))(((((((....))))))).))). ( -55.00) >DroGri_CAF1 121734 114 - 1 AUCAGCGAUUUCUGUAUAUCGCUGAUGGCCGAAGCCUCUGAGGGUGCCGCACUUCCAAUGGGCGUGUCAUCCGGAUAAUAAUCCAUAAAGCUGUCCAGCAGCUUCGACAGCUCC (((((((((........)))))))))(((((((((..(((.(((((((((.((......))))).).)))))((((....))))...........)))..))))))...))).. ( -41.40) >DroWil_CAF1 131421 114 - 1 AUAAGGGAUUUCUGUAUAUCGCUAAUGGCAGAGGCUUCUGAUGGAGCAUUCGUUACAAUUGGUGUAUCAUCGGGAUAGUAUUCCAUUACACUAUCGAGCAGCUUGGAGAGCUCU ...((((.(((((.......((.....))((..(((((((((((.((((..((....))..)))).)))))))((((((..........))))))))))..)).))))).)))) ( -29.00) >DroAna_CAF1 72165 114 - 1 AUCAGCGACUUCUGAAUAUCACUGAUGGCCGAGGCCGCUGAGGGUGCGAUGGUCACGAUGGGCGUGUCAUCCGGAUAAUACUCCAUUACGCUGUCCAACAGCUUGGACAACUCC .((((((.((((.(..((((...))))..))))).))))))(((((((((((.((((.....))))))))).(((......))).....))(((((((....))))))))))). ( -39.10) >consensus AUCAGCGACUUCUGUAUAUCGCUGAUGGCCGAGGCCUCUGAGGGAGCAUUGGUCACAAUGGGCGUGUCAUCCGGAUAAUACUCCAUUACGCUGUCCAGCAGCUUGGACAGCUCC (((((((((........)))))))))(((....)))......(((((..........(((((..((((.....))))....))))).....(((((((....)))))))))))) (-22.07 = -23.55 + 1.48)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:47:24 2006