
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 9,107,496 – 9,107,655 |
| Length | 159 |
| Max. P | 0.985366 |

| Location | 9,107,496 – 9,107,615 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.46 |
| Mean single sequence MFE | -34.60 |
| Consensus MFE | -29.42 |
| Energy contribution | -29.10 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.98 |
| SVM RNA-class probability | 0.984509 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 9107496 119 - 22407834 AAUUUGGCACAGCGGUGAGAUGGCGAAGUAGUCUCUUCAUGGCCAGUCAUUAAUGCAGAUGGGUGGCCAUCAAAUCAGCUCAACUCGACACCAACAAACAACUGCGUGUACAAG-CCCAU ..((((((((.((((((((.((((((((......))))(((((((.(((((......))))).))))))).......)).)).)))(....)........))))))))).))))-..... ( -37.50) >DroSec_CAF1 9669 107 - 1 AAUUUGGCACAGCGGUGAGAUGGCGAAGCAGACUCUUCAUGGCCAGUCAUUAAUGCAGAUGGGUGGCCAUCAAAUCUGCUCAACUCGACACCAACAAAC------------AAG-CCCAU ..((((((...))((((.((.(..((.(((((...((.(((((((.(((((......))))).))))))).)).)))))))..)))..))))..)))).------------...-..... ( -32.40) >DroSim_CAF1 9740 119 - 1 AAUUUGGCACAGCGGUGAGAUGGCGAAGCAGACUCUUCAUGGCCAGUCAUUAAUGCAGAUGGGUGGCCAUCAAAUCAGCUCAACUCGACACCAACAAACAACUGUGUGUACAAG-UCCAU .((((((((((.(((((((.((((((((......))))(((((((.(((((......))))).))))))).......)).)).)))(....)........))))))))).))))-).... ( -36.00) >DroEre_CAF1 9837 114 - 1 AAUUUGGCACAGCGGUGAGAUGGCGAAGCAGACUCUUCAUGGCCAGUCAUUAAUGCAGAUGGGUGGCCAUCAAAUCAGCUCAACUCGACACCAACAGGCAACUA------CACUUGCCAU ....(((((....((((((.((((((((......))))(((((((.(((((......))))).))))))).......)).)).)))...)))...((....)).------....))))). ( -37.40) >DroYak_CAF1 14497 111 - 1 AAUUUGGCACAGCGGUGAGAUGGCGAAGCAGACUCUUCAUGGCCAGUCAUUAAUGCAGAUGGUUGGCCAUCAAAUCAGCUCAACUCGACACCAACAAACAACUA------GAA---GCAC ..((((((...))((((((.((((((((......))))(((((((..((((......))))..))))))).......)).)).)))...)))..))))......------...---.... ( -29.70) >consensus AAUUUGGCACAGCGGUGAGAUGGCGAAGCAGACUCUUCAUGGCCAGUCAUUAAUGCAGAUGGGUGGCCAUCAAAUCAGCUCAACUCGACACCAACAAACAACUA______CAAG_CCCAU ..((((((...))((((((.((((((((......))))(((((((.(((((......))))).))))))).......)).)).)))...)))..))))...................... (-29.42 = -29.10 + -0.32)

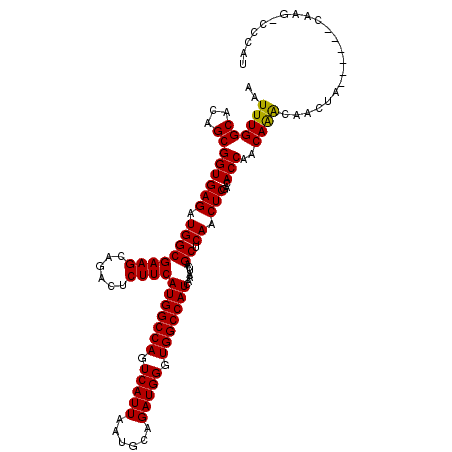
| Location | 9,107,535 – 9,107,655 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.67 |
| Mean single sequence MFE | -35.32 |
| Consensus MFE | -33.42 |
| Energy contribution | -33.62 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.67 |
| Structure conservation index | 0.95 |
| SVM decision value | 1.48 |
| SVM RNA-class probability | 0.957412 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 9107535 120 + 22407834 AGCUGAUUUGAUGGCCACCCAUCUGCAUUAAUGACUGGCCAUGAAGAGACUACUUCGCCAUCUCACCGCUGUGCCAAAUUAAAUGCAUAAUUACUUGGUAAUUGGAUUUGUGGCCGGCUC (((((.....(((((((..(((........)))..)))))))((((......))))(((((....(((...((((((((((......))))...))))))..)))....)))))))))). ( -34.20) >DroSec_CAF1 9696 120 + 1 AGCAGAUUUGAUGGCCACCCAUCUGCAUUAAUGACUGGCCAUGAAGAGUCUGCUUCGCCAUCUCACCGCUGUGCCAAAUUAAAUGCAUAAUUACUUGGUAAUUGGAUUUGUGGCCGGCUC (((((((((.(((((((..(((........)))..)))))))...))))))))).............((((.((((...(((((.((..(((((...))))))).))))))))))))).. ( -39.10) >DroSim_CAF1 9779 120 + 1 AGCUGAUUUGAUGGCCACCCAUCUGCAUUAAUGACUGGCCAUGAAGAGUCUGCUUCGCCAUCUCACCGCUGUGCCAAAUUAAAUGCAUAAUUACUUGGUAAUUGGAUUUGUGGCCGGCUC (((.(((((.(((((((..(((........)))..)))))))...))))).))).............((((.((((...(((((.((..(((((...))))))).))))))))))))).. ( -34.50) >DroEre_CAF1 9871 120 + 1 AGCUGAUUUGAUGGCCACCCAUCUGCAUUAAUGACUGGCCAUGAAGAGUCUGCUUCGCCAUCUCACCGCUGUGCCAAAUUAAAUGCAUAAUUACUUGGUAAUUGGAUUUGUGGCCGGCUC (((.(((((.(((((((..(((........)))..)))))))...))))).))).............((((.((((...(((((.((..(((((...))))))).))))))))))))).. ( -34.50) >DroYak_CAF1 14528 120 + 1 AGCUGAUUUGAUGGCCAACCAUCUGCAUUAAUGACUGGCCAUGAAGAGUCUGCUUCGCCAUCUCACCGCUGUGCCAAAUUAAAUGCAUAAUUACUUGGUAAUUGGAUUUGUGGCCGGCUC (((.(((((.(((((((..(((........)))..)))))))...))))).))).............((((.((((...(((((.((..(((((...))))))).))))))))))))).. ( -34.30) >consensus AGCUGAUUUGAUGGCCACCCAUCUGCAUUAAUGACUGGCCAUGAAGAGUCUGCUUCGCCAUCUCACCGCUGUGCCAAAUUAAAUGCAUAAUUACUUGGUAAUUGGAUUUGUGGCCGGCUC (((((.....(((((((..(((........)))..)))))))((((......))))(((((....(((...((((((((((......))))...))))))..)))....)))))))))). (-33.42 = -33.62 + 0.20)

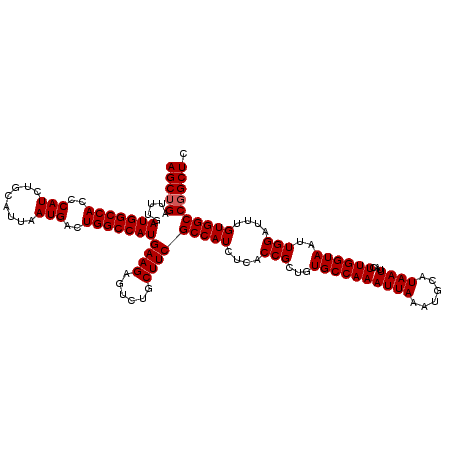
| Location | 9,107,535 – 9,107,655 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.67 |
| Mean single sequence MFE | -35.92 |
| Consensus MFE | -34.58 |
| Energy contribution | -34.78 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.00 |
| SVM RNA-class probability | 0.985366 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 9107535 120 - 22407834 GAGCCGGCCACAAAUCCAAUUACCAAGUAAUUAUGCAUUUAAUUUGGCACAGCGGUGAGAUGGCGAAGUAGUCUCUUCAUGGCCAGUCAUUAAUGCAGAUGGGUGGCCAUCAAAUCAGCU .....((((((..(((.((((((...)))))).((((((.....((((...((.(((((..(((......)))..))))).))..))))..)))))))))..))))))............ ( -38.40) >DroSec_CAF1 9696 120 - 1 GAGCCGGCCACAAAUCCAAUUACCAAGUAAUUAUGCAUUUAAUUUGGCACAGCGGUGAGAUGGCGAAGCAGACUCUUCAUGGCCAGUCAUUAAUGCAGAUGGGUGGCCAUCAAAUCUGCU .....((((((..(((.((((((...)))))).((((((....(((((.((..((.(((...((...))...))).)).))))))).....)))))))))..))))))............ ( -36.00) >DroSim_CAF1 9779 120 - 1 GAGCCGGCCACAAAUCCAAUUACCAAGUAAUUAUGCAUUUAAUUUGGCACAGCGGUGAGAUGGCGAAGCAGACUCUUCAUGGCCAGUCAUUAAUGCAGAUGGGUGGCCAUCAAAUCAGCU .....((((((..(((.((((((...)))))).((((((....(((((.((..((.(((...((...))...))).)).))))))).....)))))))))..))))))............ ( -36.00) >DroEre_CAF1 9871 120 - 1 GAGCCGGCCACAAAUCCAAUUACCAAGUAAUUAUGCAUUUAAUUUGGCACAGCGGUGAGAUGGCGAAGCAGACUCUUCAUGGCCAGUCAUUAAUGCAGAUGGGUGGCCAUCAAAUCAGCU .....((((((..(((.((((((...)))))).((((((....(((((.((..((.(((...((...))...))).)).))))))).....)))))))))..))))))............ ( -36.00) >DroYak_CAF1 14528 120 - 1 GAGCCGGCCACAAAUCCAAUUACCAAGUAAUUAUGCAUUUAAUUUGGCACAGCGGUGAGAUGGCGAAGCAGACUCUUCAUGGCCAGUCAUUAAUGCAGAUGGUUGGCCAUCAAAUCAGCU (.(((((((((......((((((...)))))).((((((....(((((.((..((.(((...((...))...))).)).))))))).....))))))).)))))))))............ ( -33.20) >consensus GAGCCGGCCACAAAUCCAAUUACCAAGUAAUUAUGCAUUUAAUUUGGCACAGCGGUGAGAUGGCGAAGCAGACUCUUCAUGGCCAGUCAUUAAUGCAGAUGGGUGGCCAUCAAAUCAGCU .....((((((..(((.((((((...)))))).((((((....(((((.((((.((...)).))((((......)))).))))))).....)))))))))..))))))............ (-34.58 = -34.78 + 0.20)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:46:25 2006