
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 8,928,180 – 8,928,333 |
| Length | 153 |
| Max. P | 0.734329 |

| Location | 8,928,180 – 8,928,300 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.22 |
| Mean single sequence MFE | -41.25 |
| Consensus MFE | -30.95 |
| Energy contribution | -30.82 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.628462 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 8928180 120 + 22407834 CACAUCGCUGAUUAUGAGAACCAUGCGAUCGGCCAGUGAAUUCGAUGAGGCCAUGCGUCGUCUGAGCAUAGCUUCGAUUGAGUCCGCCUUGGUUCAACCAACAAUAGUGGUGCCCACGCC ..((((((((....((.((((((.((.....))..(((.((((((((((((.((((.((....)))))).))))).))))))).)))..))))))...))....))))))))........ ( -35.90) >DroVir_CAF1 109203 120 + 1 CACAUCGCUUAUUAUGAAAACAAUGCGCUCGGCGAGCGAAUUCGAUGAGGCCAUGCGUCGCCUGAGCAUAGCCUCAAUCGAGUCCGCUUUGAUACAGCCCACAAUUGUGGUUCCCACGCC ......((.((((.(....).)))).))..((((((((.((((((((((((.((((.((....)))))).))))).))))))).))))..........((((....))))......)))) ( -41.80) >DroGri_CAF1 108894 120 + 1 CACAUCGCUCAUAAUGAAAACAAUGCGCUCCGCAAGCGAAUUCGAUGAGGCCAUGCGCCGCUUGAGCAUAGCCUCAAUCGAGUCCGCACUUAUCCAGCCCACAAUUGUGGUGCCCACGCC ......((.(((..(....)..))).))...((..(((.((((((((((((.((((.........)))).))))).))))))).))).........((((((....)))).))....)). ( -40.20) >DroWil_CAF1 202248 120 + 1 CACAUCAUUGAUAAUGAAAACCAUGCGAUCAGCAAGCGAAUUCGAUGAGGCCAUGCGUCGUCUGAGCAUAGCCUCAAUCGAGUCCGCAUUGAUUCAACCCACGAUACUAGUGCCUAGGCC ...(((.(((((.(((.....)))...)))))((((((.((((((((((((.((((.((....)))))).))))).))))))).))).)))...........))).((((...))))... ( -35.50) >DroMoj_CAF1 173059 120 + 1 GACAUCGUUGAUAAUGAAAACCAUGCGAUCGGCAAGUGAAUUCGAUGAGGCAAUGCGUCGAUUGAGCAUUGCCUCGAUCGAGUCCGCUCUGAUACAGCCCACGAUUGUGGUGCCGACGCC ....((((.....)))).......(((.((((((((((.(((((((((((((((((.((....)))))))))))).))))))).))))..........((((....))))))))))))). ( -49.40) >DroAna_CAF1 140197 120 + 1 CACGUCCCUUAUAAUGCGCACCAUGCGCUCGGCCAGCGAAUUCGAUGAGGCCAUGCGGCGCCUGAGCAUAGCUUCGAUUGAGUCCGCCCUGGUCCAGCCGACGAUCGUGGUGCCCACGCC ..((((.........((((.....))))..((((((((.((((((((((((.((((.........)))).))))).))))))).)))..))))).....))))..(((((...))))).. ( -44.70) >consensus CACAUCGCUGAUAAUGAAAACCAUGCGAUCGGCAAGCGAAUUCGAUGAGGCCAUGCGUCGCCUGAGCAUAGCCUCAAUCGAGUCCGCACUGAUACAGCCCACAAUUGUGGUGCCCACGCC ........................(((...(((..(((.((((((((((((.((((.........)))).)))))).)))))).))).........(((.((....))))))))..))). (-30.95 = -30.82 + -0.14)



| Location | 8,928,180 – 8,928,300 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.22 |
| Mean single sequence MFE | -47.02 |
| Consensus MFE | -35.47 |
| Energy contribution | -37.00 |
| Covariance contribution | 1.53 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.32 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.43 |
| SVM RNA-class probability | 0.734329 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 8928180 120 - 22407834 GGCGUGGGCACCACUAUUGUUGGUUGAACCAAGGCGGACUCAAUCGAAGCUAUGCUCAGACGACGCAUGGCCUCAUCGAAUUCACUGGCCGAUCGCAUGGUUCUCAUAAUCAGCGAUGUG ...((((...))))(((((((((((((((((..((((........((.(((((((((....)).))))))).)).(((...........))))))).))))))....))))))))))).. ( -38.20) >DroVir_CAF1 109203 120 - 1 GGCGUGGGAACCACAAUUGUGGGCUGUAUCAAAGCGGACUCGAUUGAGGCUAUGCUCAGGCGACGCAUGGCCUCAUCGAAUUCGCUCGCCGAGCGCAUUGUUUUCAUAAUAAGCGAUGUG ((((......((((....))))..........((((((.(((((.((((((((((((....)).))))))))))))))).))))))))))...((((((((((.......)))))))))) ( -54.40) >DroGri_CAF1 108894 120 - 1 GGCGUGGGCACCACAAUUGUGGGCUGGAUAAGUGCGGACUCGAUUGAGGCUAUGCUCAAGCGGCGCAUGGCCUCAUCGAAUUCGCUUGCGGAGCGCAUUGUUUUCAUUAUGAGCGAUGUG .((((.(((.((((....))))))).)......(((((.(((((.((((((((((.(.....).))))))))))))))).))))).)))....((((((((((.......)))))))))) ( -52.60) >DroWil_CAF1 202248 120 - 1 GGCCUAGGCACUAGUAUCGUGGGUUGAAUCAAUGCGGACUCGAUUGAGGCUAUGCUCAGACGACGCAUGGCCUCAUCGAAUUCGCUUGCUGAUCGCAUGGUUUUCAUUAUCAAUGAUGUG .((....)).....((((((.(((.((((((..(((((.(((((.((((((((((((....)).))))))))))))))).))))).(((.....))))))))).....))).)))))).. ( -44.80) >DroMoj_CAF1 173059 120 - 1 GGCGUCGGCACCACAAUCGUGGGCUGUAUCAGAGCGGACUCGAUCGAGGCAAUGCUCAAUCGACGCAUUGCCUCAUCGAAUUCACUUGCCGAUCGCAUGGUUUUCAUUAUCAACGAUGUC .((((((((.((((....)))).........(((.(((.(((((.((((((((((((....)).))))))))))))))).))).)))))))).))).((((.......))))........ ( -49.70) >DroAna_CAF1 140197 120 - 1 GGCGUGGGCACCACGAUCGUCGGCUGGACCAGGGCGGACUCAAUCGAAGCUAUGCUCAGGCGCCGCAUGGCCUCAUCGAAUUCGCUGGCCGAGCGCAUGGUGCGCAUUAUAAGGGACGUG ...(((.((((((....((((((((.......((((((.((.((.((.(((((((.........))))))).)))).)).)))))))))))).))..)))))).)))............. ( -42.41) >consensus GGCGUGGGCACCACAAUCGUGGGCUGAAUCAAAGCGGACUCGAUCGAGGCUAUGCUCAGGCGACGCAUGGCCUCAUCGAAUUCGCUUGCCGAGCGCAUGGUUUUCAUUAUCAGCGAUGUG (((...(((.((((....)))))))........(((((.(((((.((((((((((((....)).))))))))))))))).)))))..)))...(((((.(((.........))).))))) (-35.47 = -37.00 + 1.53)

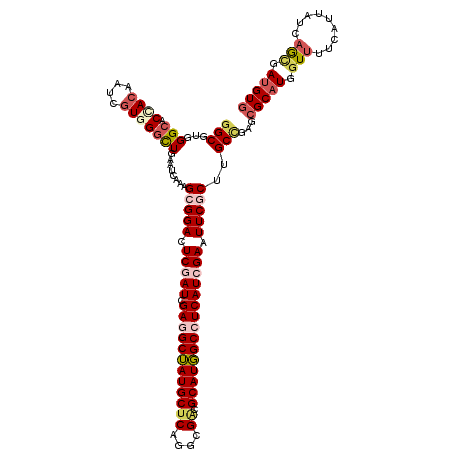
| Location | 8,928,220 – 8,928,333 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.18 |
| Mean single sequence MFE | -45.10 |
| Consensus MFE | -33.28 |
| Energy contribution | -33.87 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.36 |
| SVM RNA-class probability | 0.705722 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 8928220 113 - 22407834 -------CCCUCCUCCUGGUAGCUGUGCGAUCGCAGCAUGGGCGUGGGCACCACUAUUGUUGGUUGAACCAAGGCGGACUCAAUCGAAGCUAUGCUCAGACGACGCAUGGCCUCAUCGAA -------...(((.((((((.((((((....)))))).(((((....)).)))..............))).))).))).....((((.(((((((((....)).)))))))....)))). ( -37.30) >DroVir_CAF1 109243 113 - 1 -------CUCUCCUCCUGAUAGCUAUGCGAGCGCAACAUGGGCGUGGGAACCACAAUUGUGGGCUGUAUCAAAGCGGACUCGAUUGAGGCUAUGCUCAGGCGACGCAUGGCCUCAUCGAA -------...((((((((...(((.(((....))).....))).))))).((((....))))(((.......)))))).(((((.((((((((((((....)).))))))))))))))). ( -44.90) >DroGri_CAF1 108934 113 - 1 -------CUCUCCUCCUGAUAGCUGUGCGAGCGCAACAUUGGCGUGGGCACCACAAUUGUGGGCUGGAUAAGUGCGGACUCGAUUGAGGCUAUGCUCAAGCGGCGCAUGGCCUCAUCGAA -------...(((.(((..(((((((((..((((.......))))..))))(((....))))))))....)).).))).(((((.((((((((((.(.....).))))))))))))))). ( -47.20) >DroWil_CAF1 202288 120 - 1 UGUUCUAGUAUCCUCCUGGUAACUGUGCGAGCGAAUCAUUGGCCUAGGCACUAGUAUCGUGGGUUGAAUCAAUGCGGACUCGAUUGAGGCUAUGCUCAGACGACGCAUGGCCUCAUCGAA .((((((((..((....))..)))).....(((.....(..(((((.(.........).)))))..).....)))))))(((((.((((((((((((....)).))))))))))))))). ( -40.40) >DroMoj_CAF1 173099 113 - 1 -------CCCUCCUCCUGAUAGCUGUGCGAGCGCAACAUGGGCGUCGGCACCACAAUCGUGGGCUGUAUCAGAGCGGACUCGAUCGAGGCAAUGCUCAAUCGACGCAUUGCCUCAUCGAA -------...(((.((((((((((.(((....))).....)))..((((.((((....)))))))))))))).).))).(((((.((((((((((((....)).))))))))))))))). ( -50.20) >DroPer_CAF1 195620 113 - 1 -------GUCUCCUCCUGGUAGCUGUGGGAGCGCAGCAUCGGCGUGGGUACUACAAUGGAGGGCUGGAUCAGGGCGGACUCGAUCGAGGCAAUGCUCAGGCGUCGCAUUGCCUCAUCGAA -------((((.(.((((((.((((((....)))))).(((((.(.....(......)..).)))))))))))).))))(((((.((((((((((.........))))))))))))))). ( -50.60) >consensus _______CUCUCCUCCUGAUAGCUGUGCGAGCGCAACAUGGGCGUGGGCACCACAAUUGUGGGCUGAAUCAAAGCGGACUCGAUCGAGGCUAUGCUCAGGCGACGCAUGGCCUCAUCGAA ..........(((...((((((((((((..((((.......))))..))))(((....)))))))..))))....))).(((((.((((((((((((....)).))))))))))))))). (-33.28 = -33.87 + 0.59)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:45:15 2006