
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
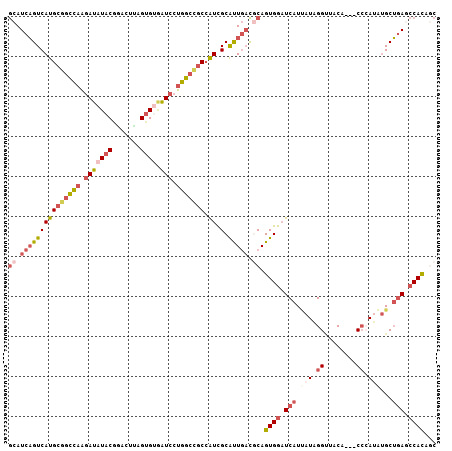
| Location | 8,866,936 – 8,867,040 |
| Length | 104 |
| Max. P | 0.886166 |

| Location | 8,866,936 – 8,867,040 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 78.48 |
| Mean single sequence MFE | -34.73 |
| Consensus MFE | -21.18 |
| Energy contribution | -23.27 |
| Covariance contribution | 2.09 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.87 |
| SVM RNA-class probability | 0.871895 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
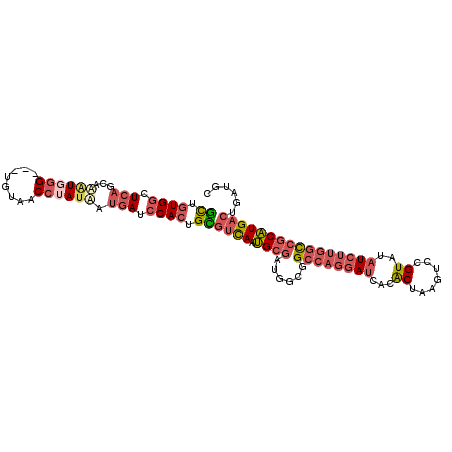
>2L_DroMel_CAF1 8866936 104 + 22407834 GCAUCAGUCAUGCGGCCAAGAUAUACGGACUUAGUGUGAUCCUGGCCGCCAUCGCAUUGACACAGUGGAUCAUUAUAGGUUACA---CCCAUAUGCUGAGCCACAGC ((....((((.(((((((.(((((((.......)))).))).)))))))........))))...((((.(((.((((((.....---.)).)))).))).)))).)) ( -34.40) >DroVir_CAF1 21938 107 + 1 ACAUACGUCACGGCAUGCGCAUCUACUUGUUGAGUGCGGUCUUUGCCGCAGUUGCGCUACUCCAGUGGAUCAUAAUUGGAUACUUCUCGGACUGGCGCAAGCAUAAA ......(((.(((...(((((.((((((...))))(((((....))))))).)))))...((((((........))))))......))))))..((....))..... ( -32.50) >DroSec_CAF1 77168 104 + 1 GCAUCAGUCAUGCGGCCAAGAUAUACGGACUUAGUGUAAUCCUGGCUGCCAUCGCAUUGACGCAGUGGAUCAUUAUAGGUAACA---CCCAUAUGCUGAGCCACAGC ((.(((((((((((((((.(((((((.......)))).))).)))))).))).).))))).)).((((.(((.((((((.....---.)).)))).))).))))... ( -35.10) >DroSim_CAF1 75667 104 + 1 GCAUCAGUCAUGCGGCCAAGAUAUACGGACUUAGUGUAAUCCUGGCUGCCAUCGCAUUGACGCAGUGGAUCAUUAUAGGUUACA---CCCAUAUGCUGAGCCACAGC ((.(((((((((((((((.(((((((.......)))).))).)))))).))).).))))).)).((((.(((.((((((.....---.)).)))).))).))))... ( -35.10) >DroEre_CAF1 81394 104 + 1 GCAUCAGUCAUGCGGCCAAGAUAUACGGACUCAGCGUGAUCCUGGCCGCCAUCGCAUUGACGCAGUGGAUCAUCAUAGGCUCCA---CCCAUUUGCUGAGCCACAGC ((.(((((((((((((((.(((.((((.......))))))).)))))).))).).))))).)).((((.(((.((..((.....---.))...)).))).))))... ( -35.90) >DroYak_CAF1 79402 104 + 1 GCAUCAGUCAUGCGGCCAAGAUAUACGGACUAAGUGCCAUCCUGGCCGCCAUCGCAUUGACGCAGUGGCUCAUCAUUGGCUCCA---CCGAGUUGCUGACCCACAAC ((.(((((((((((((((.(((.(((.......)))..))).)))))).))).).))))).)).((((.(((.((...((((..---..)))))).))).))))... ( -35.40) >consensus GCAUCAGUCAUGCGGCCAAGAUAUACGGACUUAGUGUGAUCCUGGCCGCCAUCGCAUUGACGCAGUGGAUCAUUAUAGGUUACA___CCCAUAUGCUGAGCCACAGC ((.(((((((((((((((.(((((((.......)))).))).))))))).)).).))))).)).((((.(((.(((.((........)).)))...))).))))... (-21.18 = -23.27 + 2.09)


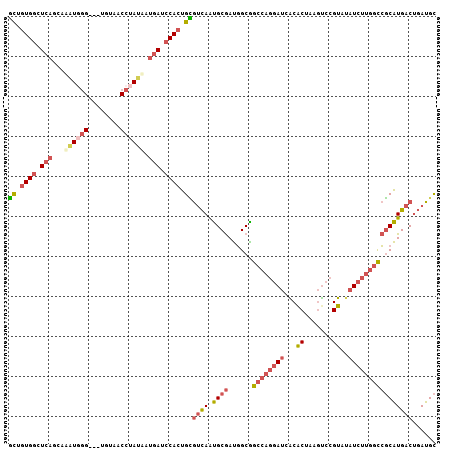
| Location | 8,866,936 – 8,867,040 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 107 |
| Reading direction | reverse |
| Mean pairwise identity | 78.48 |
| Mean single sequence MFE | -37.83 |
| Consensus MFE | -22.68 |
| Energy contribution | -24.43 |
| Covariance contribution | 1.76 |
| Combinations/Pair | 1.27 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.94 |
| SVM RNA-class probability | 0.886166 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 8866936 104 - 22407834 GCUGUGGCUCAGCAUAUGGG---UGUAACCUAUAAUGAUCCACUGUGUCAAUGCGAUGGCGGCCAGGAUCACACUAAGUCCGUAUAUCUUGGCCGCAUGACUGAUGC ...((((.(((...((((((---.....)))))).))).)))).((((((...((...(((((((((((...((.......))..))))))))))).))..)))))) ( -37.70) >DroVir_CAF1 21938 107 - 1 UUUAUGCUUGCGCCAGUCCGAGAAGUAUCCAAUUAUGAUCCACUGGAGUAGCGCAACUGCGGCAAAGACCGCACUCAACAAGUAGAUGCGCAUGCCGUGACGUAUGU ..((((((..(((((((..((...(((......)))..)).))))).((((((((.((((((......))))(((.....))))).))))).)))))..).))))). ( -30.40) >DroSec_CAF1 77168 104 - 1 GCUGUGGCUCAGCAUAUGGG---UGUUACCUAUAAUGAUCCACUGCGUCAAUGCGAUGGCAGCCAGGAUUACACUAAGUCCGUAUAUCUUGGCCGCAUGACUGAUGC ((.((((.(((...((((((---.....)))))).))).)))).))((((.((((......(((((((((((.........))).))))))))))))))))...... ( -37.50) >DroSim_CAF1 75667 104 - 1 GCUGUGGCUCAGCAUAUGGG---UGUAACCUAUAAUGAUCCACUGCGUCAAUGCGAUGGCAGCCAGGAUUACACUAAGUCCGUAUAUCUUGGCCGCAUGACUGAUGC ((.((((.(((...((((((---.....)))))).))).)))).))((((.((((......(((((((((((.........))).))))))))))))))))...... ( -37.50) >DroEre_CAF1 81394 104 - 1 GCUGUGGCUCAGCAAAUGGG---UGGAGCCUAUGAUGAUCCACUGCGUCAAUGCGAUGGCGGCCAGGAUCACGCUGAGUCCGUAUAUCUUGGCCGCAUGACUGAUGC ...((((.(((....(((((---.....)))))..))).)))).((((((...((...(((((((((((.(((.......)))..))))))))))).))..)))))) ( -41.80) >DroYak_CAF1 79402 104 - 1 GUUGUGGGUCAGCAACUCGG---UGGAGCCAAUGAUGAGCCACUGCGUCAAUGCGAUGGCGGCCAGGAUGGCACUUAGUCCGUAUAUCUUGGCCGCAUGACUGAUGC ...((.((((((((((.(((---(((..(.......)..)))))).))...)))....((((((((((((..((.......)).)))))))))))).))))).)).. ( -42.10) >consensus GCUGUGGCUCAGCAAAUGGG___UGUAACCUAUAAUGAUCCACUGCGUCAAUGCGAUGGCGGCCAGGAUCACACUAAGUCCGUAUAUCUUGGCCGCAUGACUGAUGC ((.((((.(((...((((((........)))))).))).)))).))((((.((((......((((((((...((.......))..))))))))))))))))...... (-22.68 = -24.43 + 1.76)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:44:45 2006