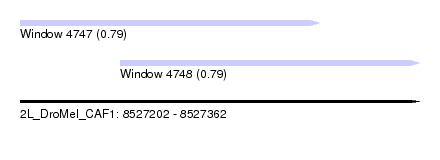
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 8,527,202 – 8,527,362 |
| Length | 160 |
| Max. P | 0.794359 |

| Location | 8,527,202 – 8,527,322 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.67 |
| Mean single sequence MFE | -52.90 |
| Consensus MFE | -35.72 |
| Energy contribution | -37.90 |
| Covariance contribution | 2.18 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.52 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.57 |
| SVM RNA-class probability | 0.786923 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 8527202 120 + 22407834 CUCCUGAUGUGGAGCUAGUUGAGGCGUGGGAAGUGGUCGAGCCAGUGGCCAGUUUUGUGGGCAGUCCUCCGCUGGCUGCCGGCGAGGCCAUGGCCCUGGAAUUCACAACUCCCGCACUCC ((((......))))......(((..((((((.((((.....((((.((((((((((((.(((((.((......))))))).)))))))..)))))))))...))))...)))))).))). ( -58.60) >DroPse_CAF1 3983 99 + 1 UCCCCGAUGUCGAGCUGGUUGACGCAUGGGAAGUCGUCGAACCCGUUGCCAGUUUGGUGGGCAGCCCCCCACUAGCCGCCGGCGAGGCCAUGGUCCUGG--------------------- ...(((..(((..((((((....(((((((..(....)...)))).)))..(.((((((((......)))))))).)))))))..)))..)))......--------------------- ( -33.20) >DroSim_CAF1 35695 120 + 1 CUCCUGAUGUGGAGCUAGUUGAGGCGUGGGAUGUGGUCGACCCAGUGGCCAGUUUGGUGGGCAGUCCCCCGCUGGCUGCCGGCGAGGCCAUGGCCCUUGAGUUCACAACUCCCGCACUUC ((((......))))......((((.((((((.((.((.(((.(((.(((((((((.((.(((((((.......))))))).)).))))..))))).))).))).)).)))))))).)))) ( -54.60) >DroEre_CAF1 36291 120 + 1 CUCCUGAAGUGGAGCUAGUUGAGGCGUGGGAAGUGGUCGACCCAGUGGCCAGUUUGGUUGGCAGUCCCCCACUGGCUGCCGGCGAGGCCAUGGCCCUGGAGUUCACAAUCCCCGCACUCC ((((......))))......((((((.((((....((.(((((((.(((((((((.((((((((((.......)))))))))).))))..))))))))).))).))..))))))).))). ( -59.40) >DroYak_CAF1 37048 120 + 1 CUCCUGAAGUGGAGCUAGUUGAGGCGUGGGAAGUGGUCGACCCAGUGGCCAGUUUGGUGGGCAGUCCACCGCUGGCUGCCGGCGAUGCCAUGGCCCUGGAAUUCACAACACCCGCACUCC ((((......))))......((((((.(((..((((.....((((.(((((((.(.((.(((((.(((....)))))))).)).).))..)))))))))...))))..).))))).))). ( -51.30) >DroAna_CAF1 19977 120 + 1 UUCCAGAAUUGGAGCUGGUCGAAGCGUGAGAAGUAGUCGAUCCCGUGGCUAGCUUCGUAGGUAGUCCUCCGCUGGCUGCCGGCGAAGCUAUGGUACGGGAUCCUGCCGCUCCAGGGCUCG ....((..(((((((.(((.(...(....)........(((((((((.((((((((((.(((((.((......))))))).))))))))).).)))))))))).))))))))))..)).. ( -60.30) >consensus CUCCUGAAGUGGAGCUAGUUGAGGCGUGGGAAGUGGUCGACCCAGUGGCCAGUUUGGUGGGCAGUCCCCCGCUGGCUGCCGGCGAGGCCAUGGCCCUGGAAUUCACAACUCCCGCACUCC ((((......))))......((((((.(((..((((.....((((.(((((((((.((.(((((((.......))))))).)).))))..)))))))))...))))..))).))).))). (-35.72 = -37.90 + 2.18)

| Location | 8,527,242 – 8,527,362 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.50 |
| Mean single sequence MFE | -51.16 |
| Consensus MFE | -42.79 |
| Energy contribution | -44.10 |
| Covariance contribution | 1.31 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.60 |
| SVM RNA-class probability | 0.794359 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 8527242 120 + 22407834 GCCAGUGGCCAGUUUUGUGGGCAGUCCUCCGCUGGCUGCCGGCGAGGCCAUGGCCCUGGAAUUCACAACUCCCGCACUCCCAGCUCCGGCAGUAUCUGUCAUCUUAGGCAAGGCUACUGG .(((((((((.(((((((.(((((.((......))))))).)))))))....(((..(((.............((.......))...(((((...))))).)))..)))..))))))))) ( -51.00) >DroPse_CAF1 4023 96 + 1 ACCCGUUGCCAGUUUGGUGGGCAGCCCCCCACUAGCCGCCGGCGAGGCCAUGGUCCUGG-----------------------GCACCGGC-GCGUCUAUCAGCUUCGGGCCCACCACGGG .(((((.........(((((((.((((...((((...(((.....)))..))))...))-----------------------)).((((.-((........)).)))))))))))))))) ( -44.90) >DroSec_CAF1 36431 120 + 1 CCCAGUGGCCAGUUUGGUGGGCAGUCCCCCGCUGGCUGCCGGCGAGGCCAUAGCCCUGGAGUUCACAACUCCCGCACUUCCAGCACCGGCAGCAUCUGUCAGCUUAGGCAAGGCUACUGG .(((((((((.....((((((......)))))).((((((((.(.(((....)))..(((((.....)))))...........).))))))))...((((......)))).))))))))) ( -55.00) >DroSim_CAF1 35735 120 + 1 CCCAGUGGCCAGUUUGGUGGGCAGUCCCCCGCUGGCUGCCGGCGAGGCCAUGGCCCUUGAGUUCACAACUCCCGCACUUCCAGCACCGGCAGCAUCUGUCAGCUUAGGCAAGGCUACUGG .(((((((((.....((((((......)))))).((((((((.(.(((....)))...((((.....))))............).))))))))...((((......)))).))))))))) ( -53.60) >DroEre_CAF1 36331 120 + 1 CCCAGUGGCCAGUUUGGUUGGCAGUCCCCCACUGGCUGCCGGCGAGGCCAUGGCCCUGGAGUUCACAAUCCCCGCACUCCCAGCUCCGGCAGCAUCUGUCAUCUUAGGCAAGGCUACUGG .(((((((((.((((((.((((((..........((((((((.(.(((....)))(((((((.(.........).))).))))).))))))))..))))))..))))))..))))))))) ( -52.50) >DroYak_CAF1 37088 120 + 1 CCCAGUGGCCAGUUUGGUGGGCAGUCCACCGCUGGCUGCCGGCGAUGCCAUGGCCCUGGAAUUCACAACACCCGCACUCCCAGCUCCGGCAGCAUCUGUCAUCUUAGGCAAGGCUACUGG .((((((((((((..(((((.....)))))))).((((((((.(..((....)).((((....................))))).))))))))...((((......)))).))))))))) ( -49.95) >consensus CCCAGUGGCCAGUUUGGUGGGCAGUCCCCCGCUGGCUGCCGGCGAGGCCAUGGCCCUGGAGUUCACAACUCCCGCACUCCCAGCACCGGCAGCAUCUGUCAGCUUAGGCAAGGCUACUGG .(((((((((....(((((((......)))))))((((((((.(.(((....)))((((....................))))).))))))))...((((......)))).))))))))) (-42.79 = -44.10 + 1.31)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:41:25 2006