
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 815,556 – 815,692 |
| Length | 136 |
| Max. P | 0.940521 |

| Location | 815,556 – 815,668 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 79.64 |
| Mean single sequence MFE | -46.40 |
| Consensus MFE | -27.81 |
| Energy contribution | -27.23 |
| Covariance contribution | -0.58 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.26 |
| SVM RNA-class probability | 0.659018 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
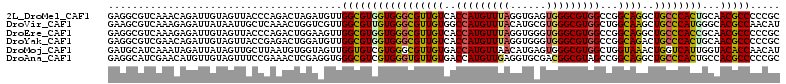
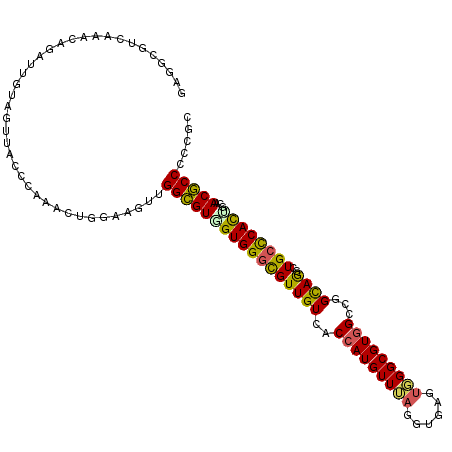
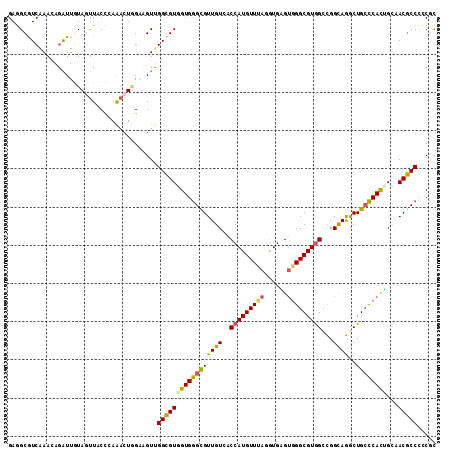
>2L_DroMel_CAF1 815556 112 + 22407834 GAGGCGUCAAACAGAUUGUAGUUACCCAGACUAGAUGUUGGCGUGGUGGGCGUUGUCACCAUGUUUAGGUGAGUGGGCGUGGCCGGCAGGCUGCCCACUGCAACGCCCCCGC ...(((((((.((...(.(((((.....))))).))))))))))((.(((((((((((((.......))))(((((((..((((....)))))))))))))))))))))).. ( -54.30) >DroVir_CAF1 2268 112 + 1 GAAGCGUCAAAGAGAUUAUAAUUGCUCAAACUGGUCGUUGGCGUUGUGGGCGUUGUGGCCAUGUUUACAUGCGUGGGCGUGGCUGGCAAGCUGCCCAUGGGCACGCCAACAU ((.(((................))))).........((((((((((((((((((((((((((((((((....)))))))))))).))))..))))))))...)))))))).. ( -44.09) >DroEre_CAF1 2103 112 + 1 GAGGCGUCAAAGAGAUUGUAGUUACCCAGACUGGAAGUUGGCGUGGUGGGCGUUGUCACCAUGUUUAGGUGGGUGGGCGUGGCCGGCAGGCUGCCCACCGCAACGCCCCCGC ...(((((((.....((.(((((.....))))).)).)))))))((.(((((((((((((.......))))(((((((..((((....)))))))))))))))))))))).. ( -58.00) >DroYak_CAF1 2101 112 + 1 GAGGCGUCGAACAGAUUGUAGUUACCGAGACUGGAUGUUGGCGUGGUGGGCGUUGUCACCAUGUUUAGGUGGGUGGGCGUGGCCGGCAGACUGCCCACUGCAACGCCCCCGC ...(((((.((((...(.(((((.....))))).))))))))))((.(((((((((((((.......))))(((((((((.(....)..)).)))))))))))))))))).. ( -48.10) >DroMoj_CAF1 2192 112 + 1 GAUGCAUCAAAUAGAUUAUAGUUGCUUAAUGUGGUAGUUGGUGUCGUGGGCGUUGUGACCAUGUUAACAUGAGUGGGCGUGGCUGGUAAACUGGUCAUUGGUACACCAACAU ......................((((......))))(((((((((....((((..(.((((((....)))).)).)))))(((..(....)..)))....).)))))))).. ( -28.20) >DroAna_CAF1 2162 112 + 1 GAGGCAUCGAACAUGUUGUAGUUUCCGAAACUCGAGGUGGGCGUCGUGGGUGUUGUGACCAUGUUGAGGUGCGACGGCGUAGCCGGCAGGCUGCCCACUGCCACGCCCCCGC (((...((((((........)))..)))..)))..((.((((((.(..(..(((((.(((.......))))))))((.((((((....)))))))).)..).)))))))).. ( -45.70) >consensus GAGGCGUCAAACAGAUUGUAGUUACCCAAACUGGAAGUUGGCGUGGUGGGCGUUGUCACCAUGUUUAGGUGAGUGGGCGUGGCCGGCAGGCUGCCCACUGCAACGCCCCCGC .......................................(((((((((((((((((..(((((((((......)))))))))...))))..))))))))...)))))..... (-27.81 = -27.23 + -0.58)
| Location | 815,556 – 815,668 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 79.64 |
| Mean single sequence MFE | -37.20 |
| Consensus MFE | -20.86 |
| Energy contribution | -21.62 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.86 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.622737 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 815556 112 - 22407834 GCGGGGGCGUUGCAGUGGGCAGCCUGCCGGCCACGCCCACUCACCUAAACAUGGUGACAACGCCCACCACGCCAACAUCUAGUCUGGGUAACUACAAUCUGUUUGACGCCUC ((((((((((((..((((((.(((....)))...))))))(((((.......))))))))))))).)).......((..(((..((........))..)))..))..))... ( -45.30) >DroVir_CAF1 2268 112 - 1 AUGUUGGCGUGCCCAUGGGCAGCUUGCCAGCCACGCCCACGCAUGUAAACAUGGCCACAACGCCCACAACGCCAACGACCAGUUUGAGCAAUUAUAAUCUCUUUGACGCUUC .(((((((((.....(((((.(.(((...(((..((....))(((....))))))..)))))))))..)))))))))....(((.(((..........)))...)))..... ( -30.70) >DroEre_CAF1 2103 112 - 1 GCGGGGGCGUUGCGGUGGGCAGCCUGCCGGCCACGCCCACCCACCUAAACAUGGUGACAACGCCCACCACGCCAACUUCCAGUCUGGGUAACUACAAUCUCUUUGACGCCUC ((((((((((((.(((((((.(((....)))...)))))))((((.......)))).)))))))).)).............(((.(((..........)))...)))))... ( -47.70) >DroYak_CAF1 2101 112 - 1 GCGGGGGCGUUGCAGUGGGCAGUCUGCCGGCCACGCCCACCCACCUAAACAUGGUGACAACGCCCACCACGCCAACAUCCAGUCUCGGUAACUACAAUCUGUUCGACGCCUC ((((((((((((..((((((.(((....)))...)))))).((((.......)))).)))))))).)).............(((.(((..........)))...)))))... ( -40.80) >DroMoj_CAF1 2192 112 - 1 AUGUUGGUGUACCAAUGACCAGUUUACCAGCCACGCCCACUCAUGUUAACAUGGUCACAACGCCCACGACACCAACUACCACAUUAAGCAACUAUAAUCUAUUUGAUGCAUC ..((((((((.....(((((((((.((.................)).))).))))))...((....))))))))))...........(((.(............).)))... ( -21.23) >DroAna_CAF1 2162 112 - 1 GCGGGGGCGUGGCAGUGGGCAGCCUGCCGGCUACGCCGUCGCACCUCAACAUGGUCACAACACCCACGACGCCCACCUCGAGUUUCGGAAACUACAACAUGUUCGAUGCCUC ((((.((((((((....(((.....))).)))))))).))))...(((((((((((...........)))................(....).....)))))).))...... ( -37.50) >consensus GCGGGGGCGUUGCAGUGGGCAGCCUGCCGGCCACGCCCACCCACCUAAACAUGGUCACAACGCCCACCACGCCAACAUCCAGUCUGGGUAACUACAAUCUGUUUGACGCCUC (((.((((((...(((((((.(((....)))...)))))))..........((....))))))))....)))........................................ (-20.86 = -21.62 + 0.76)

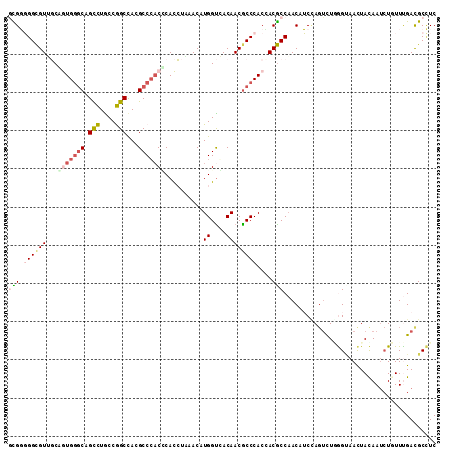
| Location | 815,593 – 815,692 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | forward |
| Mean pairwise identity | 85.32 |
| Mean single sequence MFE | -50.18 |
| Consensus MFE | -38.79 |
| Energy contribution | -40.35 |
| Covariance contribution | 1.56 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.82 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.829372 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
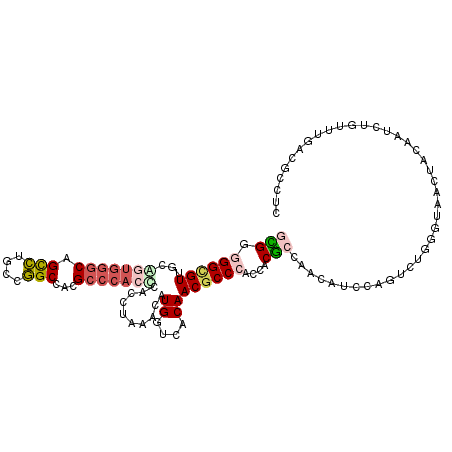
>2L_DroMel_CAF1 815593 99 + 22407834 UUGGCGUGGUGGGCGUUGUCACCAUGUUUAGGUGAGUGGGCGUGGCCGGCAGGCUGCCCACUGCAACGCCCCCGCUCACUGCGGAUGCGAUGGAGGAUG ...((((...(((((((((((((.......))))(((((((..((((....))))))))))))))))))))((((.....))))))))........... ( -50.40) >DroSec_CAF1 2138 99 + 1 UUGGCGUGGUGGGCGUUGUCACCAUGUUUAGGUGAGUGGGCGUGGCCGGCAGGCUGCCCACUGCAAUGCCCCCGCUCACUGCGGAUGCGGUGGAGGAUG .....((((.(((((((((((((.......))))(((((((..((((....))))))))))))))))))))))))(((((((....)))))))...... ( -53.80) >DroSim_CAF1 2138 99 + 1 UUGGCGUGGUGGGCGUUGUCACCAUGUUUAGGUGAGUGGGCGUGGCCGGCAGGCUGCCCACUGCAAUGCCCCCGCUCACUGCGGAUGCGGUGGAGGAUG .....((((.(((((((((((((.......))))(((((((..((((....))))))))))))))))))))))))(((((((....)))))))...... ( -53.80) >DroEre_CAF1 2140 99 + 1 UUGGCGUGGUGGGCGUUGUCACCAUGUUUAGGUGGGUGGGCGUGGCCGGCAGGCUGCCCACCGCAACGCCCCCGCUCACUGCGGAUGCGGAAGAGGAUG .(((.((((.(((((((((((((.......))))(((((((..((((....)))))))))))))))))))))))))))((((....))))......... ( -54.20) >DroYak_CAF1 2138 99 + 1 UUGGCGUGGUGGGCGUUGUCACCAUGUUUAGGUGGGUGGGCGUGGCCGGCAGACUGCCCACUGCAACGCCCCCGCUGACUACGGAAACGGAGGAGGAUG .....((((.(((((((((((((.......))))(((((((((.(....)..)).))))))))))))))))))))...((.((....)).))....... ( -46.10) >DroPer_CAF1 2203 92 + 1 UCGGUGUUGUGGGUGUCGUUACCAAAUGUACAUGGGUGGGCGUGGCGGGCAGACUGCCCACCCCCACUCCUACGCCCACAGCCGAUCCCGGG------- (((((..(((((((((((((....)))).....(((((((.(((((((.....))).)))).)))))))..)))))))))))))).......------- ( -42.80) >consensus UUGGCGUGGUGGGCGUUGUCACCAUGUUUAGGUGAGUGGGCGUGGCCGGCAGGCUGCCCACUGCAACGCCCCCGCUCACUGCGGAUGCGGUGGAGGAUG ..((.((((.(((((((((((((.......))))(((((((..((((....)))))))))))))))))))))))))).((((....))))......... (-38.79 = -40.35 + 1.56)

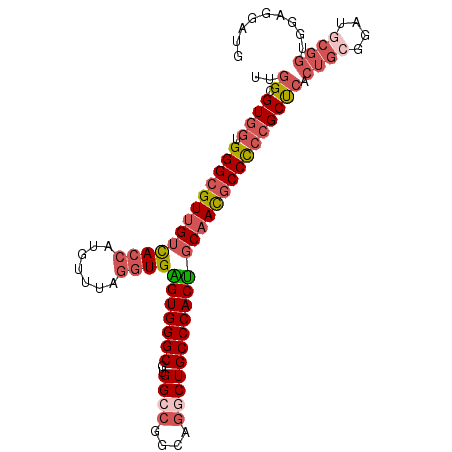
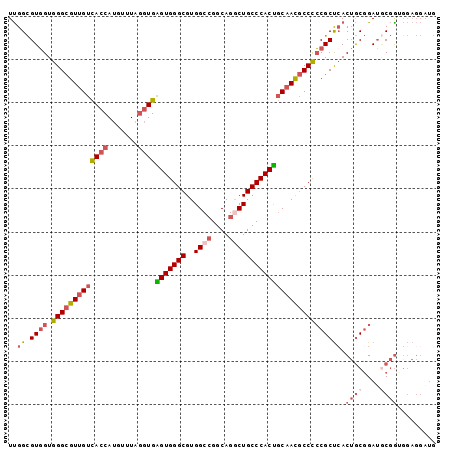
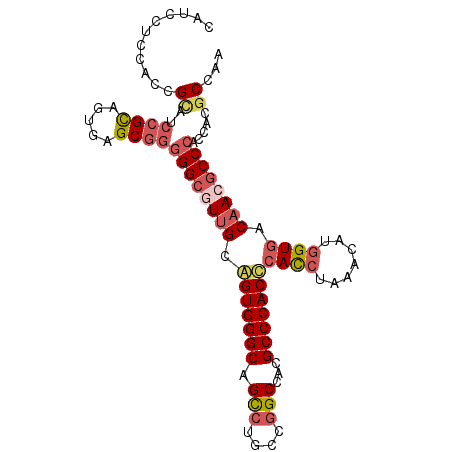
| Location | 815,593 – 815,692 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 85.32 |
| Mean single sequence MFE | -42.23 |
| Consensus MFE | -34.38 |
| Energy contribution | -35.43 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.18 |
| Mean z-score | -3.10 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.29 |
| SVM RNA-class probability | 0.940521 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 815593 99 - 22407834 CAUCCUCCAUCGCAUCCGCAGUGAGCGGGGGCGUUGCAGUGGGCAGCCUGCCGGCCACGCCCACUCACCUAAACAUGGUGACAACGCCCACCACGCCAA ...........((..((((.....))))((((((((..((((((.(((....)))...))))))(((((.......))))))))))))).....))... ( -45.60) >DroSec_CAF1 2138 99 - 1 CAUCCUCCACCGCAUCCGCAGUGAGCGGGGGCAUUGCAGUGGGCAGCCUGCCGGCCACGCCCACUCACCUAAACAUGGUGACAACGCCCACCACGCCAA .......(((.((....)).))).(((.((((.(((..((((((.(((....)))...))))))(((((.......)))))))).))))....)))... ( -40.20) >DroSim_CAF1 2138 99 - 1 CAUCCUCCACCGCAUCCGCAGUGAGCGGGGGCAUUGCAGUGGGCAGCCUGCCGGCCACGCCCACUCACCUAAACAUGGUGACAACGCCCACCACGCCAA .......(((.((....)).))).(((.((((.(((..((((((.(((....)))...))))))(((((.......)))))))).))))....)))... ( -40.20) >DroEre_CAF1 2140 99 - 1 CAUCCUCUUCCGCAUCCGCAGUGAGCGGGGGCGUUGCGGUGGGCAGCCUGCCGGCCACGCCCACCCACCUAAACAUGGUGACAACGCCCACCACGCCAA ...........((..((((.....))))((((((((.(((((((.(((....)))...)))))))((((.......)))).)))))))).....))... ( -47.50) >DroYak_CAF1 2138 99 - 1 CAUCCUCCUCCGUUUCCGUAGUCAGCGGGGGCGUUGCAGUGGGCAGUCUGCCGGCCACGCCCACCCACCUAAACAUGGUGACAACGCCCACCACGCCAA ..........(((..((((.....))))((((((((..((((((.(((....)))...)))))).((((.......)))).))))))))...))).... ( -38.20) >DroPer_CAF1 2203 92 - 1 -------CCCGGGAUCGGCUGUGGGCGUAGGAGUGGGGGUGGGCAGUCUGCCCGCCACGCCCACCCAUGUACAUUUGGUAACGACACCCACAACACCGA -------.......((((.((((((.((.((.((((((((((((.....)))))))...)))))))...(((.....)))...)).))))))...)))) ( -41.70) >consensus CAUCCUCCACCGCAUCCGCAGUGAGCGGGGGCGUUGCAGUGGGCAGCCUGCCGGCCACGCCCACCCACCUAAACAUGGUGACAACGCCCACCACGCCAA ...........((..((((.....))))((((((((.(((((((.(((....)))...)))))))((((.......)))).)))))))).....))... (-34.38 = -35.43 + 1.06)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:32:05 2006