
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
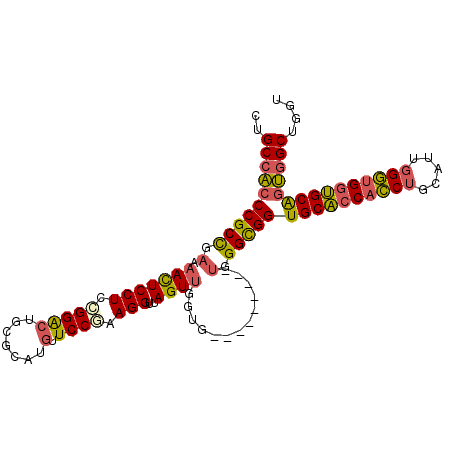
| Location | 7,895,852 – 7,895,948 |
| Length | 96 |
| Max. P | 0.897416 |

| Location | 7,895,852 – 7,895,948 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 85.19 |
| Mean single sequence MFE | -47.42 |
| Consensus MFE | -36.74 |
| Energy contribution | -36.49 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.27 |
| Mean z-score | -2.40 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.742013 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 7895852 96 + 22407834 CUGCCACCCGCUGAAAACUCCUCCGGGCUGCGCAUGUUCCGAAGGUUAGUUGGUG---------UGGGCGGUGCACCACCUGCAUUGGGUGGUGCGGUGGCUGGU ..((((((((((.(.(((((((.(((((.......).)))).)))..))))....---------).))))..(((((((((.....))))))))))))))).... ( -45.10) >DroSec_CAF1 68429 96 + 1 CUGCCACCCGCUGAAAACUCCUCGGGGCUGCGCAUGUUCCGAAGGUUAGUUGGUG---------UGGGCGGUGCACCACCUGCAUUGGGUGGUGCAGUGGCUGGU ..((((((((((.(.(((((((((((((.......)))))).)))..))))....---------).)))))((((((((((.....))))))))))))))).... ( -49.20) >DroSim_CAF1 69720 96 + 1 CUGCCACCCGCUGAAAACUCCUCGGGGCUGCGCAUGUUCCGAAGGUUAGUUGGUG---------UGGGCGGUGCACCACCUGCAUUGGGUGGUGCAGUGGCUGGU ..((((((((((.(.(((((((((((((.......)))))).)))..))))....---------).)))))((((((((((.....))))))))))))))).... ( -49.20) >DroEre_CAF1 68669 96 + 1 CUGCCACCCGCCGAAAACUCCUCCGGACUGCGCAUGUUCCGAAGGUUAGUUGGUG---------UGGGCGGUGCACCACCUGCAGUGGGUGGUGCACUGGCUGGC ..((((((((((((.....(((.(((((.......).)))).)))....))))))---------.)).(((((((((((((.....)))))))))))))..)))) ( -49.00) >DroYak_CAF1 70069 96 + 1 CUGCCACCCGCCGAGAACUCCUCCGGACUGCGCAUAUUCCGAAGGCUAGUUGGUG---------UGGGCGGUGCACCACCUGCAUGGGGUGGUGCAGUGGCUGGU ..((((((((((.(.(((((((.((((.((....)).)))).)))..)))...).---------).)))))((((((((((.....))))))))))))))).... ( -44.30) >DroAna_CAF1 68225 102 + 1 CUGCCGCCCGCCGAAAAUUCCUCAGGACUGCGUAUGUUCCUCAGGCUAGUGGGUGCCACCGUGGCGGGUGGUGCCCCAGCAGC---GGUCGCGGCAGCAGCGGGU (((((((((((.........((.(((((.......).)))).))(((.(.(((..(((((......)))))..))))))).))---))..)))))))........ ( -47.70) >consensus CUGCCACCCGCCGAAAACUCCUCCGGACUGCGCAUGUUCCGAAGGUUAGUUGGUG_________UGGGCGGUGCACCACCUGCAUUGGGUGGUGCAGUGGCUGGU ..((((((((((.(.(((((((.(((((.......).)))).)))..)))).............).)))))((((((((((.....))))))))))))))).... (-36.74 = -36.49 + -0.25)


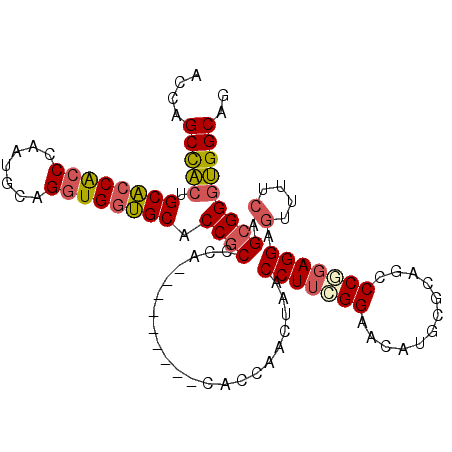
| Location | 7,895,852 – 7,895,948 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 85.19 |
| Mean single sequence MFE | -43.27 |
| Consensus MFE | -34.08 |
| Energy contribution | -34.42 |
| Covariance contribution | 0.34 |
| Combinations/Pair | 1.20 |
| Mean z-score | -3.04 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.00 |
| SVM RNA-class probability | 0.897416 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 7895852 96 - 22407834 ACCAGCCACCGCACCACCCAAUGCAGGUGGUGCACCGCCCA---------CACCAACUAACCUUCGGAACAUGCGCAGCCCGGAGGAGUUUUCAGCGGGUGGCAG ....((((((((((((((.......))))))))..(((...---------....((((..(((((((............)))))))))))....))))))))).. ( -42.20) >DroSec_CAF1 68429 96 - 1 ACCAGCCACUGCACCACCCAAUGCAGGUGGUGCACCGCCCA---------CACCAACUAACCUUCGGAACAUGCGCAGCCCCGAGGAGUUUUCAGCGGGUGGCAG ....((((((((((((((.......)))))))))((((...---------....((((...((((((.............))))))))))....))))))))).. ( -38.52) >DroSim_CAF1 69720 96 - 1 ACCAGCCACUGCACCACCCAAUGCAGGUGGUGCACCGCCCA---------CACCAACUAACCUUCGGAACAUGCGCAGCCCCGAGGAGUUUUCAGCGGGUGGCAG ....((((((((((((((.......)))))))))((((...---------....((((...((((((.............))))))))))....))))))))).. ( -38.52) >DroEre_CAF1 68669 96 - 1 GCCAGCCAGUGCACCACCCACUGCAGGUGGUGCACCGCCCA---------CACCAACUAACCUUCGGAACAUGCGCAGUCCGGAGGAGUUUUCGGCGGGUGGCAG ((((.((.((((((((((.......)))))))))).(((..---------....((((..((((((((.(.......)))))))))))))...))))).)))).. ( -47.50) >DroYak_CAF1 70069 96 - 1 ACCAGCCACUGCACCACCCCAUGCAGGUGGUGCACCGCCCA---------CACCAACUAGCCUUCGGAAUAUGCGCAGUCCGGAGGAGUUCUCGGCGGGUGGCAG ....((((((((((((((.......)))))))))(((((..---------....((((..((((((((..........))))))))))))...)))))))))).. ( -47.00) >DroAna_CAF1 68225 102 - 1 ACCCGCUGCUGCCGCGACC---GCUGCUGGGGCACCACCCGCCACGGUGGCACCCACUAGCCUGAGGAACAUACGCAGUCCUGAGGAAUUUUCGGCGGGCGGCAG .....((((((((.((.((---((((.((((...(((((......)))))..)))).)))(((.((((.(.......))))).)))......))))))))))))) ( -45.90) >consensus ACCAGCCACUGCACCACCCAAUGCAGGUGGUGCACCGCCCA_________CACCAACUAACCUUCGGAACAUGCGCAGCCCGGAGGAGUUUUCAGCGGGUGGCAG ....(((((.((((((((.......)))))))).((((......................(((((((............))))))).(....).))))))))).. (-34.08 = -34.42 + 0.34)

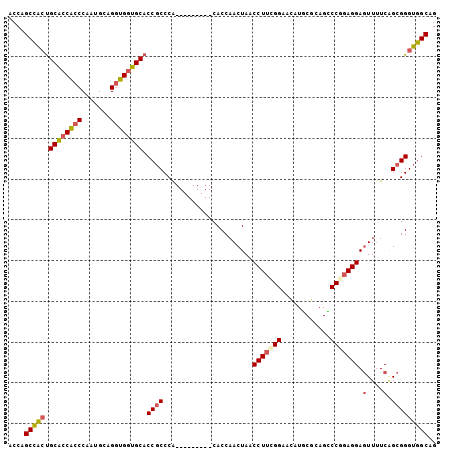
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:36:13 2006