
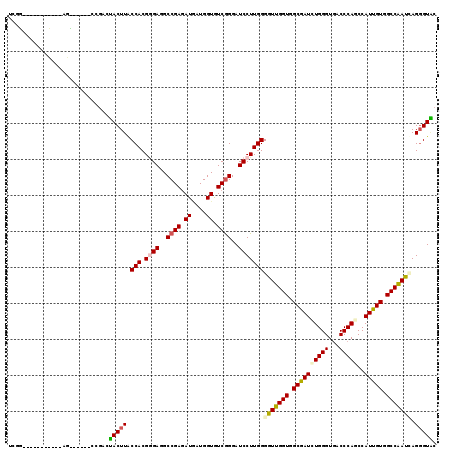
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 7,827,048 – 7,827,180 |
| Length | 132 |
| Max. P | 0.919022 |

| Location | 7,827,048 – 7,827,144 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 76.70 |
| Mean single sequence MFE | -41.20 |
| Consensus MFE | -31.25 |
| Energy contribution | -31.42 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.46 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.12 |
| SVM RNA-class probability | 0.919022 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 7827048 96 + 22407834 -----------------------GCGAUUACUUACCACGGGAGGCCGAAAUUAUGGUGUCGGGAUCCUUGGGGUUGGUGGCGAUCUGGGUGACCCAUCCAUUGUGGCCAAUGAGGGUAC -----------------------.....(((((.(((.((((..((((.((....)).))))..))))))).((((((.((((..((((((...)))))))))).))))))..))))). ( -33.60) >DroPse_CAF1 11514 118 + 1 UCGGCAUU-CGCGAAAGCGGCUUCCGAGUACUUGCCACGGGAGGCCGAGAUGAUGGUGUCGGGAUCCUUGGGGUUGGUGGCGAUUUGGGUGACCCAGCCAUUGUGGCCGAUCAGGGUAC ((((.(..-(((....)))..).))))((((((.(((.((((..((((.((....)).))))..)))))))(((((((.((((((((((...)))))..))))).))))))).)))))) ( -52.20) >DroGri_CAF1 4017 112 + 1 UCAAUUUAACCUCAAAGU-------GAAUACUUGCCACGCGACGCCGAGAUGAUGGUGUCGGGAUCCUUGGGAUUGGUCGCAAUCUGGGUAACCCAGCCAUUGUGGCCGAUCAGCGUGC ..............((((-------....))))..((((((((((((......)))))))...........((((((((((((((((((...)))))..))))))))))))).))))). ( -43.00) >DroWil_CAF1 5934 96 + 1 -----------------------UUCACAACUUACCACGAGAUGCCGAGAUAAUGGUAUCGGGAUCCUUGGGAUUGGUAGCGAUUUGGGUGACCCAGCCAUUGUGGCCAAUUAGGGUUC -----------------------.....(((((.(((.(.(((.((((.((....)).)))).)))).)))(((((((.((((((((((...)))))..))))).))))))).))))). ( -30.60) >DroAna_CAF1 5030 103 + 1 UGGG-----------AGU-----GGGUCUACUUACCACGGGAGGCCGAGAUGAUGGUGUCGGGAUCCUUGGGGUUGGUGGCGAUCUGGGUGACCCAUCCAUUGUGGCCAAUGAGGGUAC ..((-----------(.(-----(((((.((((((((.((((..((((.((....)).))))..)))))))(((((....)))))))))))))))))))...(((.((.....)).))) ( -39.50) >DroPer_CAF1 11494 118 + 1 UCGGCAUU-CGCGAAAGCGGCUUCCGAGUACUUGCCACGGGAGGCCGAGAUGAUGGUGUCAGGAUCCUUGGGGUUGGUGGCGAUUUGGGUGACCCAGCCAUUGUGGCCGAUCAGGGUAC ((((.(..-(((....)))..).))))((((((.(((.((((..((..(((......))).)).)))))))(((((((.((((((((((...)))))..))))).))))))).)))))) ( -48.30) >consensus UCGG___________AG______CCGACUACUUACCACGGGAGGCCGAGAUGAUGGUGUCGGGAUCCUUGGGGUUGGUGGCGAUCUGGGUGACCCAGCCAUUGUGGCCAAUCAGGGUAC ............................(((((.(((.((((..((((.((....)).))))..)))))))(((((((.((((((((((...)))))..))))).))))))).))))). (-31.25 = -31.42 + 0.17)


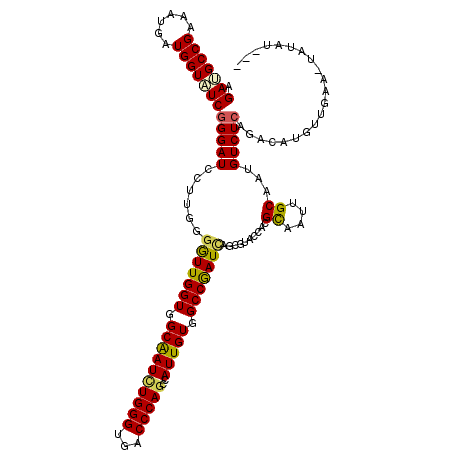
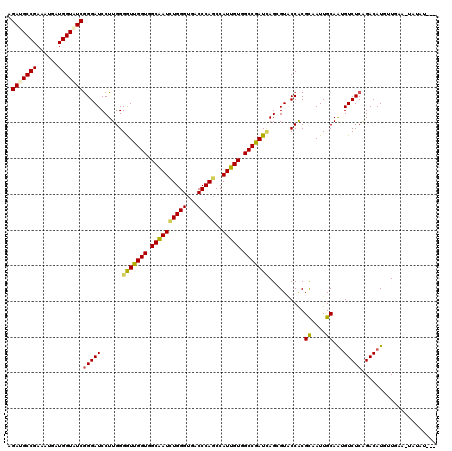
| Location | 7,827,064 – 7,827,180 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.45 |
| Mean single sequence MFE | -40.68 |
| Consensus MFE | -32.08 |
| Energy contribution | -31.63 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.589830 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 7827064 116 + 22407834 GGAGGCCGAAAUUAUGGUGUCGGGAUCCUUGGGGUUGGUGGCGAUCUGGGUGACCCAUCCAUUGUGGCCAAUGAGGGUACCGCGCAAUUGCAGGGUCUCGGACAUCUUGAG-UUUAU--- ........(((((..((((((((((((((.(.(((((((((..((((((....))....(((((....))))).)))).)))).))))).))))))))).))))))...))-)))..--- ( -42.00) >DroVir_CAF1 4020 119 + 1 CGAUGCCGAAAUGAUGGUAUCGGGAUCCUUGGGGUUGGUGGCAAUCUGGGUGACCCAGCCAUUGUGGCCGAUCAGCGUACCACGCAAUUGCAAUGUCUCAGACAUGUUUUAAUAUAACU- .(((((((......)))))))(((((..(((.(((((((.((((((((((...)))))..))))).))))))).(((.....))).....))).)))))....................- ( -39.60) >DroGri_CAF1 4049 118 + 1 CGACGCCGAGAUGAUGGUGUCGGGAUCCUUGGGAUUGGUCGCAAUCUGGGUAACCCAGCCAUUGUGGCCGAUCAGCGUGCCACGCAAUUGCAAUGUCUCAGACAUUUU--AAUAUGUUUU .(((((((......)))))))(((((..(((.((((((((((((((((((...)))))..))))))))))))).(((.....))).....))).)))))((((((...--...)))))). ( -46.80) >DroWil_CAF1 5950 116 + 1 AGAUGCCGAGAUAAUGGUAUCGGGAUCCUUGGGAUUGGUAGCGAUUUGGGUGACCCAGCCAUUGUGGCCAAUUAGGGUUCCACGUAAUUGCAAUGUCUCAGACAUGGUUAA-UAAAU--- .(((((((......)))))))((((((((...(((((((.((((((((((...)))))..))))).)))))))))))))))........((.(((((...))))).))...-.....--- ( -39.10) >DroMoj_CAF1 4354 119 + 1 AGAUGCCGAAAUAAUGGUAUCGGGAUCCUUGGGGUUGGUAGCAAUCUGGGUGACCCAGCCAUUGUGGCCGAUCAGCGUACCACGCAAUUGCAAUGUCUCAGACAUGUUGAAAUAUUACU- .(((((((......)))))))(((((..(((.(((((((.((((((((((...)))))..))))).))))))).(((.....))).....))).)))))....................- ( -37.40) >DroPer_CAF1 11532 116 + 1 GGAGGCCGAGAUGAUGGUGUCAGGAUCCUUGGGGUUGGUGGCGAUUUGGGUGACCCAGCCAUUGUGGCCGAUCAGGGUACCACGCAAUUGCAAUGUCUCAGACAUUUUCAG-UCAAU--- .......((..(((..(((((.(((((((...(((((((.((((((((((...)))))..))))).)))))))))))).))..((....)).........)))))..))).-))...--- ( -39.20) >consensus AGAUGCCGAAAUGAUGGUAUCGGGAUCCUUGGGGUUGGUGGCAAUCUGGGUGACCCAGCCAUUGUGGCCGAUCAGCGUACCACGCAAUUGCAAUGUCUCAGACAUGUUGAA_UAUAU___ .(((((((......)))))))(((((......(((((((.((((((((((...)))))..))))).)))))))..........((....))...)))))..................... (-32.08 = -31.63 + -0.44)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:35:42 2006