
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 7,353,920 – 7,354,055 |
| Length | 135 |
| Max. P | 0.971212 |

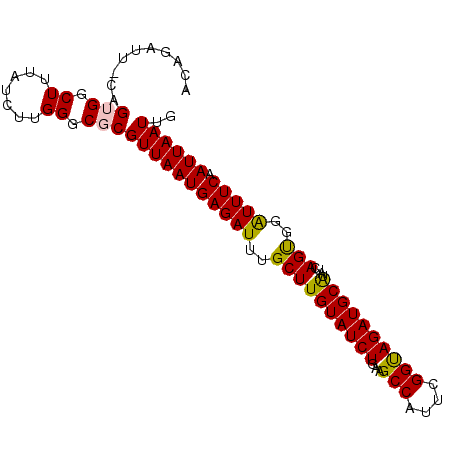
| Location | 7,353,920 – 7,354,015 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 95 |
| Reading direction | forward |
| Mean pairwise identity | 95.43 |
| Mean single sequence MFE | -25.58 |
| Consensus MFE | -24.18 |
| Energy contribution | -23.68 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.40 |
| Structure conservation index | 0.95 |
| SVM decision value | 1.67 |
| SVM RNA-class probability | 0.971212 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 7353920 95 + 22407834 CCAGAUUCCAGUGGCUUUAUCUUGGGCGCGUUAAUGAGAUUUGCUUGUAUCUGAAGCCAUUCGGCAGAUGCGUAUCAGUGGAUUUCAAUUAAUUG ..........(((.((.......)).)))((((((((((((..(((((((((...(((....))))))))))....))..)))))).)))))).. ( -26.20) >DroVir_CAF1 191008 94 + 1 ACAGAUU-CAGUGGCUUUAUCUUGGGCGCGUUAAUGAGAUUUGCUUGUAUCUGGAGCCAUUCGGUAGAUGCAUAUCAGUGGGUUUCAAUUAAUUG .......-..(((.((.......)).)))((((((((((((..(((((((((...(((....))))))))))....))..)))))).)))))).. ( -25.00) >DroPse_CAF1 172288 95 + 1 CCAGAUUCCAGUGGCUUUAUCUUGGGCUCGUUAAUGAGAUUUGCUUGUAUCUGAAGCCAUUCGGUAGAUGCAUAUCAGCGGAUUUCAAUUAAUUG ...((..((((.((.....))))))..))(((((((((((((((((((((((...(((....))))))))))....)))))))))).)))))).. ( -26.50) >DroGri_CAF1 161020 94 + 1 ACAGAUU-CAGUGGCUUUAUCUUGGGCGCGUUAAUGAGAUUUGCUUGUAUCUGGAGCCAUUCGGUAGAUGCAUAUCAGUGGGUUUCAAUUAAUUG .......-..(((.((.......)).)))((((((((((((..(((((((((...(((....))))))))))....))..)))))).)))))).. ( -25.00) >DroMoj_CAF1 202788 94 + 1 ACAGAUU-CAGUGGCUUUAUCUUGGGCGCGUUAAUGAGAUUUGCUUGUAUCUGGAGCCAUUCGGUAGAUGCGUAUCAGUGGGUUUCAAUUAAUUG .......-..(((.((.......)).)))((((((((((((..(((((((((...(((....))))))))))....))..)))))).)))))).. ( -24.30) >DroPer_CAF1 174220 95 + 1 CCAGAUUCCAGUGGCUUUAUCUUGGGCUCGUUAAUGAGAUUUGCUUGUAUCUGAAGCCAUUCGGUAGAUGCAUAUCAGCGGAUUUCAAUUAAUUG ...((..((((.((.....))))))..))(((((((((((((((((((((((...(((....))))))))))....)))))))))).)))))).. ( -26.50) >consensus ACAGAUU_CAGUGGCUUUAUCUUGGGCGCGUUAAUGAGAUUUGCUUGUAUCUGAAGCCAUUCGGUAGAUGCAUAUCAGUGGAUUUCAAUUAAUUG ..........(((.((.......)).)))(((((((((((..((((((((((...(((....))))))))))....)))..))))).)))))).. (-24.18 = -23.68 + -0.50)

| Location | 7,353,935 – 7,354,055 |
|---|---|
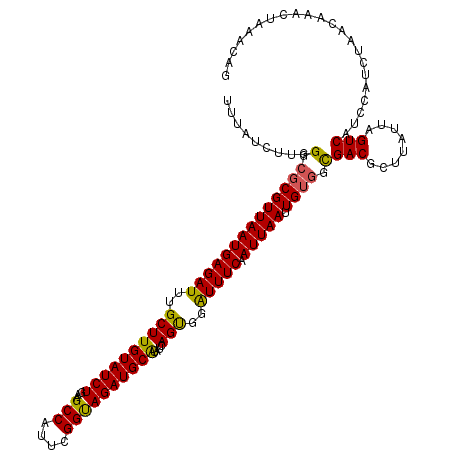
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.39 |
| Mean single sequence MFE | -30.23 |
| Consensus MFE | -27.19 |
| Energy contribution | -26.63 |
| Covariance contribution | -0.55 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.25 |
| Structure conservation index | 0.90 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.708711 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 7353935 120 + 22407834 UUUAUCUUGGGCGCGUUAAUGAGAUUUGCUUGUAUCUGAAGCCAUUCGGCAGAUGCGUAUCAGUGGAUUUCAAUUAAUUGUGGCGACGCUUAUUAGUCAUCCAUCUAACAAACUAAACAC ((((..((((.(((((((((((((((..(((((((((...(((....))))))))))....))..)))))).))))).)))).)(((........)))..........)))..))))... ( -30.90) >DroVir_CAF1 191022 120 + 1 UUUAUCUUGGGCGCGUUAAUGAGAUUUGCUUGUAUCUGGAGCCAUUCGGUAGAUGCAUAUCAGUGGGUUUCAAUUAAUUGUGGCGACGCUUAUUAGUCAUCCAUCUAACAAACUAAACUG ((((..((((.(((((((((((((((..(((((((((...(((....))))))))))....))..)))))).))))).)))).)(((........)))..........)))..))))... ( -29.70) >DroGri_CAF1 161034 120 + 1 UUUAUCUUGGGCGCGUUAAUGAGAUUUGCUUGUAUCUGGAGCCAUUCGGUAGAUGCAUAUCAGUGGGUUUCAAUUAAUUGUGGUGACGCUUAUUAGUCAUCCAUCUAACAAACUAAACUG .......(((.(((((((((((((((..(((((((((...(((....))))))))))....))..)))))).))))).))))(((((........))))))))................. ( -30.40) >DroWil_CAF1 205091 120 + 1 UUUAUCUGGGCAGCGUUAAUGAGAUUUGCUUGUAUCUGAAGCCAUUCGGUAGAUGCAUAUCAGUGGAUUUCAAUUAAUUGUGGCGACGCUUAUUAGUCAUCCAUCUAAUGAACAAAAAAA .(((..(((((.((((((((((((((..(((((((((...(((....))))))))))....))..)))))).))))).))).))(((........)))..)))..)))............ ( -30.50) >DroMoj_CAF1 202802 120 + 1 UUUAUCUUGGGCGCGUUAAUGAGAUUUGCUUGUAUCUGGAGCCAUUCGGUAGAUGCGUAUCAGUGGGUUUCAAUUAAUUGUGGCGACGCUUAUUAGUCAUCCAUCUAACAAACUAAACUG ((((..((((.(((((((((((((((..(((((((((...(((....))))))))))....))..)))))).))))).)))).)(((........)))..........)))..))))... ( -29.00) >DroPer_CAF1 174235 120 + 1 UUUAUCUUGGGCUCGUUAAUGAGAUUUGCUUGUAUCUGAAGCCAUUCGGUAGAUGCAUAUCAGCGGAUUUCAAUUAAUUGUGGCGACGCUUAUUAGUCAUCCAUCUAACAAACUAAACAC .......((((((.(((((((((((((((((((((((...(((....))))))))))....)))))))))).))))))...)))(((........)))..)))................. ( -30.90) >consensus UUUAUCUUGGGCGCGUUAAUGAGAUUUGCUUGUAUCUGAAGCCAUUCGGUAGAUGCAUAUCAGUGGAUUUCAAUUAAUUGUGGCGACGCUUAUUAGUCAUCCAUCUAACAAACUAAACAG .........(.((((((((((((((..((((((((((...(((....))))))))))....)))..))))).))))).)))).)(((........)))...................... (-27.19 = -26.63 + -0.55)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:32:20 2006