
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 7,135,467 – 7,135,558 |
| Length | 91 |
| Max. P | 0.972880 |

| Location | 7,135,467 – 7,135,558 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 80.47 |
| Mean single sequence MFE | -33.37 |
| Consensus MFE | -22.10 |
| Energy contribution | -22.02 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.15 |
| Mean z-score | -3.60 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.70 |
| SVM RNA-class probability | 0.972880 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
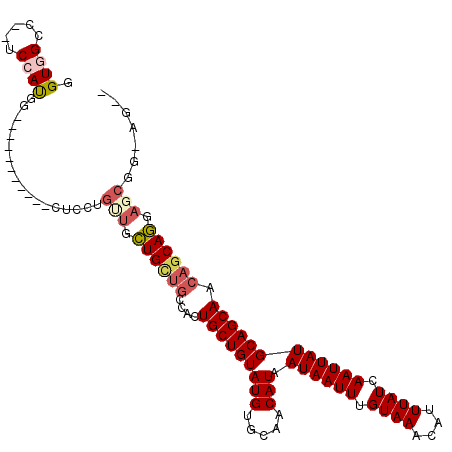
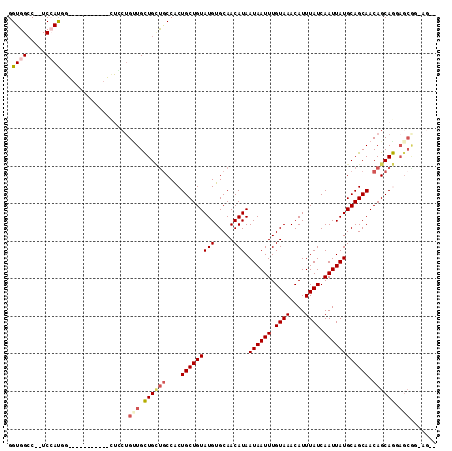
>2L_DroMel_CAF1 7135467 91 + 22407834 --CU-ACGCUCCUGCUGUUGCUGCAUAAUUGAUAAAUGUUUACAAAUUAUUAUGUUGCACAUACAGCAGUGGCAGCAGCAAC------------------AUGGA--GCCCACC --..-..((((((((((((((..(((((((..(((....)))..))))))..(((((......))))))..)))))))))..------------------..)))--))..... ( -32.40) >DroPse_CAF1 102253 101 + 1 UGCUGUUGCUGUUGCUGUUGCUGCAUAAUUGAUAAAUGUUUACAAAUUAUUAUGUUGCACAUACAGCAGUGGCAACAACAACAGGAA-----------CCAUCGA--GGCCACC ((.((((((..((((((((((.(((((((.(((...((....)).)))))))))).)))...)))))))..)))))).))...((..-----------((.....--))))... ( -32.30) >DroSim_CAF1 79586 109 + 1 --CU-CCGCUCCUGCUGUUGCUGCAUAAUUGAUAAAUGUUUACAAAUUAUUAUGUUGCACAUACAGCAGUGGCAGCAGCACCAGCAGCAGCCACAUCGCGAUGGA--GCCCACC --..-..((((((((((((((.(((((((.(((...((....)).)))))))))).)))...))))))(((((.((.((....)).)).)))))........)))--))..... ( -37.70) >DroYak_CAF1 84157 105 + 1 --CU-CCGCUCCUGCUGUUGCUGCAUAAUUGAUAAAUGUUUACAAAUUAUUAUGUUGCACAUACAGCAGUGGCAGCAGCAGCAGUACC------AUCGCCAUGGAAUGGCCACC --..-......(((((((((((((...((((....((((..(((........)))...))))....)))).)))))))))))))....------...(((((...))))).... ( -36.10) >DroAna_CAF1 82407 110 + 1 --CUGCCGCUCCUGCUGUUGCUGCAUAAUUGAUAAAUGUUUACAAAUUAUUAUGUUGCACAUACAGCAGCGGUGCCAGCAACAGCGGCAGCAACAACACAGUGGA--GGCCACC --((((((((..(((((..(((((((((((..(((....)))..))))))..(((((......))))))))))..)))))..))))))))..........((((.--..)))). ( -39.70) >DroPer_CAF1 93163 90 + 1 -----------UUGCUGUUGCUGCAUAAUUGAUAAAUGUUUACAAAUUAUUAUGUUGCACAUACAGCAGUGGCAACAACAACAGGAA-----------CCAUCGA--GGCCACC -----------.(((((((((.(((((((.(((...((....)).)))))))))).)))...))))))(((((..(...........-----------.....).--.))))). ( -21.99) >consensus __CU_CCGCUCCUGCUGUUGCUGCAUAAUUGAUAAAUGUUUACAAAUUAUUAUGUUGCACAUACAGCAGUGGCAGCAGCAACAGCAA___________CCAUGGA__GGCCACC ............((((((((((((((((((..(((....)))..))))))..(((((......))))))))))))))))).................................. (-22.10 = -22.02 + -0.08)



| Location | 7,135,467 – 7,135,558 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 80.47 |
| Mean single sequence MFE | -35.12 |
| Consensus MFE | -19.47 |
| Energy contribution | -20.13 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.19 |
| Structure conservation index | 0.55 |
| SVM decision value | 1.45 |
| SVM RNA-class probability | 0.954966 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 7135467 91 - 22407834 GGUGGGC--UCCAU------------------GUUGCUGCUGCCACUGCUGUAUGUGCAACAUAAUAAUUUGUAAACAUUUAUCAAUUAUGCAGCAACAGCAGGAGCGU-AG-- ..((.((--(((.(------------------((((.((((((...(((.......))).....((((((.((((....)))).)))))))))))).)))))))))).)-).-- ( -31.90) >DroPse_CAF1 102253 101 - 1 GGUGGCC--UCGAUGG-----------UUCCUGUUGUUGUUGCCACUGCUGUAUGUGCAACAUAAUAAUUUGUAAACAUUUAUCAAUUAUGCAGCAACAGCAACAGCAACAGCA ((.((((--.....))-----------))))((((((((((((...(((((((((.....))).((((((.((((....)))).))))))))))))...))))))))))))... ( -34.00) >DroSim_CAF1 79586 109 - 1 GGUGGGC--UCCAUCGCGAUGUGGCUGCUGCUGGUGCUGCUGCCACUGCUGUAUGUGCAACAUAAUAAUUUGUAAACAUUUAUCAAUUAUGCAGCAACAGCAGGAGCGG-AG-- ......(--(((..(((...)))(((.((((((.(((((((((.((........))))).....((((((.((((....)))).)))))))))))).)))))).)))))-))-- ( -38.80) >DroYak_CAF1 84157 105 - 1 GGUGGCCAUUCCAUGGCGAU------GGUACUGCUGCUGCUGCCACUGCUGUAUGUGCAACAUAAUAAUUUGUAAACAUUUAUCAAUUAUGCAGCAACAGCAGGAGCGG-AG-- .((.(((((...))))).))------....(((((.((((((....(((((((((.....))).((((((.((((....)))).)))))))))))).)))))).)))))-..-- ( -36.40) >DroAna_CAF1 82407 110 - 1 GGUGGCC--UCCACUGUGUUGUUGCUGCCGCUGUUGCUGGCACCGCUGCUGUAUGUGCAACAUAAUAAUUUGUAAACAUUUAUCAAUUAUGCAGCAACAGCAGGAGCGGCAG-- (((((..--.))))).........((((((((.((((((.....(((((..((((.....))))((((((.((((....)))).)))))))))))..)))))).))))))))-- ( -42.60) >DroPer_CAF1 93163 90 - 1 GGUGGCC--UCGAUGG-----------UUCCUGUUGUUGUUGCCACUGCUGUAUGUGCAACAUAAUAAUUUGUAAACAUUUAUCAAUUAUGCAGCAACAGCAA----------- ((.((((--.....))-----------))))((((((((((((...(((.......))).....((((((.((((....)))).)))))))))))))))))).----------- ( -27.00) >consensus GGUGGCC__UCCAUGG___________CUCCUGUUGCUGCUGCCACUGCUGUAUGUGCAACAUAAUAAUUUGUAAACAUUUAUCAAUUAUGCAGCAACAGCAGGAGCGG_AG__ .((((.....))))..................(((.((((((....(((((((((.....))).((((((.((((....)))).)))))))))))).)))))).)))....... (-19.47 = -20.13 + 0.67)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:31:04 2006