
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 6,529,560 – 6,529,705 |
| Length | 145 |
| Max. P | 0.815642 |

| Location | 6,529,560 – 6,529,665 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.84 |
| Mean single sequence MFE | -42.60 |
| Consensus MFE | -21.59 |
| Energy contribution | -21.45 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.66 |
| SVM RNA-class probability | 0.815642 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
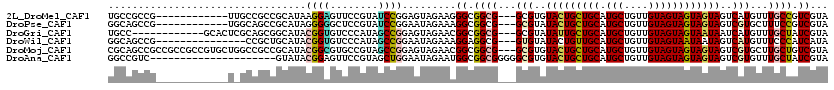
>2L_DroMel_CAF1 6529560 105 + 22407834 UGCCGCCG------------UUGCCGCCGCAUAAGGAGUUCCGUAUCCGGAGUAGAAGGGCGGCG---GCGUGUACUGCUGCAUGCUGUUGUAGUAGUAGUAGUCAUGUUUGCCGUCGUA .((((((.------------((((..(((.(((.((....)).))).))).))))...))))))(---(((((.((((((((.(((((...))))))))))))))))....)))...... ( -42.20) >DroPse_CAF1 1594 105 + 1 GGCAGCCG------------UGGCAGCCGCAUAGGGGGCUCCGUAUCCGGAAUAGAAAGGCGGCG---GCGUAUACUGCUGCAUGCUGUUGUAGUAGUAGUAGUCGUGCUUUCCGUCGUA (((.((((------------(.((.(((.(....).)))((((....))))........)).)))---))((((((((((((.(((((...))))))))))))).)))).....)))... ( -43.30) >DroGri_CAF1 2197 105 + 1 UGCC------------GCACUCGCAGCGGCAUACGGUGUCCCAUAGCCGGAGUAGAACGGCGGCG---GCGUAUAUUGCUGCAUGCUGUUGUAGUAGUAAUAAUCAUGUUUGCUAUCGUA ((((------------((.......))))))..((((........))))...((.((((((((((---(((.....)))))).))))))).))(((((((.........))))))).... ( -38.40) >DroWil_CAF1 3481 102 + 1 GGCAGCCG---------------CCGCUGCAUACGGUGUCCCAUAGCCGGAAUAGAAAGGAGGCG---GUGUAUACUGUUGCAUGCUGUUGUAGUAAUAAUAGUCAUGUUUCCCAUCAUA .(((((..---------------..)))))...((((........)))).....((..(((((((---((....))))))((((((((((((....))))))).))))).)))..))... ( -30.90) >DroMoj_CAF1 105255 117 + 1 CGCAGCCGCCGCCGCCGUGCUGGCCGCCGCAUACGGCGUGCCGUAGCCGGAGUAGAACGGCGGCG---GCGUGUACUGCUGCAUGCUGUUGUAGUAGUAGUAGUCGUGCUUGCUGUCGUA ((((((((((((((((((.(((.(((..((.(((((....))))))))))..))).)))))))))---))).((((.(((((.(((((......)))))))))).))))..)))).)).. ( -64.00) >DroAna_CAF1 2020 99 + 1 GGCCGUC---------------------GUAUACGGAGUUCCGUAGCUGGAAUAGAAUGGCGGCGGGGGCGUGUACUGCUGCAUGCUGUUGUAGUAGUAGUAGUCGUGUUUGCUAUCGUA ..((((.---------------------....)))).((((((....))))))...(((..((((((.(((..(((((((((.(((....))))))))))))..))).))))))..))). ( -36.80) >consensus GGCAGCCG____________U_GCCGCCGCAUACGGAGUUCCGUAGCCGGAAUAGAAAGGCGGCG___GCGUAUACUGCUGCAUGCUGUUGUAGUAGUAGUAGUCAUGUUUGCCAUCGUA .................................((((........)))).........((.((((...(((..(((((((((.(((....))))))))))))..)))...)))).))... (-21.59 = -21.45 + -0.14)


| Location | 6,529,588 – 6,529,705 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 82.79 |
| Mean single sequence MFE | -50.80 |
| Consensus MFE | -33.74 |
| Energy contribution | -33.77 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.64 |
| SVM RNA-class probability | 0.808111 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
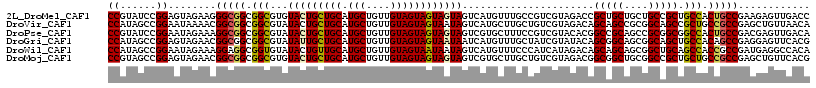
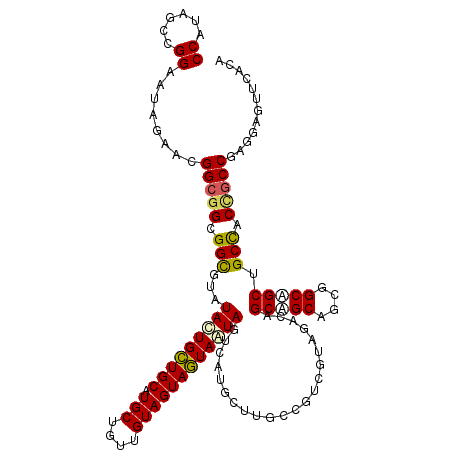
>2L_DroMel_CAF1 6529588 117 + 22407834 CCGUAUCCGGAGUAGAAGGGCGGCGGCGUGUACUGCUGCAUGCUGUUGUAGUAGUAGUAGUCAUGUUUGCCGUCGUAGACCGCUGCUGCUGCCGCUGCCACUGCCGAAGAGUUGACC (((....))).((((...((((((((((.(((((((((((......)))))))))((((((...((((((....)))))).)))))))))))))))))).))))............. ( -49.60) >DroVir_CAF1 82792 117 + 1 CCAUAGCCGGAAUAAAACGGCGGCGGCGUAUACUGCUGCAUGCUGUUGUAGUAGUAAUAGUCAUGCUUGCUGUCGUAGACAGCAGCCGCGGCAGCCGCUGCCGCCGAGCUGUUAACA ..((((((((.......((((((((((...((((((((((......))))))))))...(((.((.((((((((...)))))))).)).))).))))))))))))).)))))..... ( -58.21) >DroPse_CAF1 1622 117 + 1 CCGUAUCCGGAAUAGAAAGGCGGCGGCGUAUACUGCUGCAUGCUGUUGUAGUAGUAGUAGUCGUGCUUUCCGUCGUACACGGCCGCAGCCGCGGCGGCCACUGCCGACGAGUUGACA (((....)))....(.((((((.((((...((((((((((......))))))))))...)))))))))).)((((.((.(((((((....)))))(((....)))..)).)))))). ( -49.40) >DroGri_CAF1 2225 117 + 1 CCAUAGCCGGAGUAGAACGGCGGCGGCGUAUAUUGCUGCAUGCUGUUGUAGUAGUAAUAAUCAUGUUUGCUAUCGUAUACAGCGGCAGCGGCAGCUGCCACAGCCGAGGAGUUCACG ((...((((........))))((((((......((((((.(((((((((((((((((.........)))))))....))))))))))))))))))))))........))........ ( -47.90) >DroWil_CAF1 3506 117 + 1 CCAUAGCCGGAAUAGAAAGGAGGCGGUGUAUACUGUUGCAUGCUGUUGUAGUAAUAAUAGUCAUGUUUCCCAUCAUAGACAGCAGCAGCGGCUGCAGCCACCGCCGAUGAGGCCACA .....(((...((........(((((((....((((.((.((((((((((....)....((((((........))).))).))))))))))).)))).))))))).))..))).... ( -38.70) >DroMoj_CAF1 105295 117 + 1 CCGUAGCCGGAGUAGAACGGCGGCGGCGUGUACUGCUGCAUGCUGUUGUAGUAGUAGUAGUCGUGCUUGCUGUCGUAGACGGCGGCUGCGGCCGCUGCUGCCGCCGAGCUGUUCACG (((....))).((.(((((((((((((...((((((((((......))))))))))((((.((.(((.(((((((....)))))))...))))))))).))))))..))))))))). ( -61.00) >consensus CCAUAGCCGGAAUAGAACGGCGGCGGCGUAUACUGCUGCAUGCUGUUGUAGUAGUAAUAGUCAUGCUUGCCGUCGUAGACAGCAGCAGCGGCAGCUGCCACCGCCGAGGAGUUCACA ((......))........(((((.(((...(((((((((.(((....))))))))))))......................(((((....))))).))).)))))............ (-33.74 = -33.77 + 0.03)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:27:05 2006