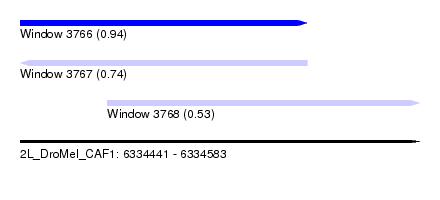
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 6,334,441 – 6,334,583 |
| Length | 142 |
| Max. P | 0.939947 |

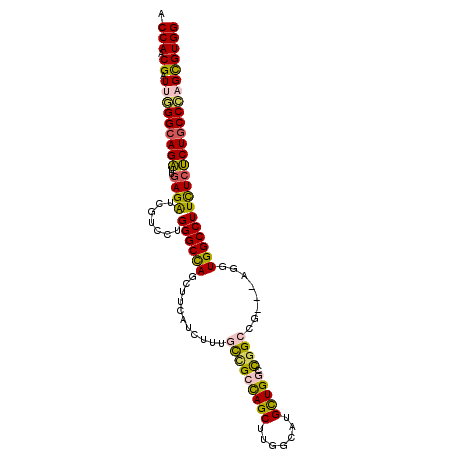
| Location | 6,334,441 – 6,334,543 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 81.49 |
| Mean single sequence MFE | -46.71 |
| Consensus MFE | -35.53 |
| Energy contribution | -34.95 |
| Covariance contribution | -0.58 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.88 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.28 |
| SVM RNA-class probability | 0.939947 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
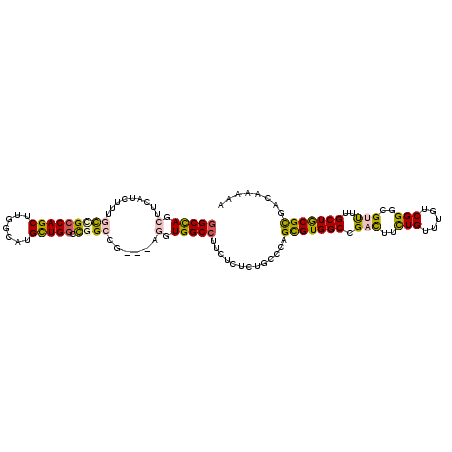
>2L_DroMel_CAF1 6334441 102 + 22407834 ACCAACGAUUUGGCAGGUUGAGAUCGUCUUGGGCUAGCUUUAGCUUUGCCGCCAGCUUGGAGUGCUGACCUGCCG---UUGUGGCCUUUUCCCUGCCCAGUGUGG .(((((.....(((((((.............(((.(((....)))..)))..((((.......))))))))))).---.((.(((.........))))))).))) ( -32.30) >DroVir_CAF1 44534 102 + 1 ACCAACGAUGCGGCAGAUUGAGAUCGUCCUGGGCCAGCUUCAUUUUCGUUGCCAGCUUGGCAUGUUGGGCGGCUG---UGCUGGCCUUCUCUCUGCCGAGCGUGG .(((.((...(((((((..((((.......((((((((..((........(((.(((..(....)..))))))))---.)))))))))))))))))))..))))) ( -45.31) >DroPse_CAF1 64475 102 + 1 ACCAUCGAUUGGGCAGAUUGAGAUCGUCUUGGGCCAGCUUCAUCUUUGCCGCCAGCUUUGCAUGCUGGUUGGAGG---AGGUGGCCUUCUCUCUGCCCAGCGUGG .(((.((.(((((((((..((((.......((((((.((((.(((.....((((((.......)))))).)))))---)).)))))))))))))))))))))))) ( -50.41) >DroGri_CAF1 19173 105 + 1 ACCAGCGAUGCGGCAGAUUUAGAUCGUCCUGGGCCAGCUUCAUUUUGGUCGCCAGCUUGGCUUGCUGGGCGGCCGAGGAGCUGGCCUUCUCUCUGCCGAGCGUGG .(((.((...(((((((...(((.......(((((((((((...((((((((((((.......))).))))))))))))))))))))))).)))))))..))))) ( -57.01) >DroAna_CAF1 26392 99 + 1 ACCAACGAUUGGGGAGGUUGAGGUCAUCCUGGGCCAGUUUCAGCUUCGCCGCUAGCUUGGCGUGUUGCCCGGCC------GUGGCCUUCUCCCUGCCCAGCGUGG .(((.((.(((((.(((..((((((((.((((((.......(((..(((((......))))).)))))))))..------)))))).))..))).)))))))))) ( -44.81) >DroPer_CAF1 62559 102 + 1 ACCAUCGAUUGGGCAGAUUGAGAUCGUCUUGGGCCAGCUUCAUCUUUGCCGCCAGCUUUGCAUGCUGGUUGGAGG---AGGUGGCCUUCUCUCUGCCCAGCGUGG .(((.((.(((((((((..((((.......((((((.((((.(((.....((((((.......)))))).)))))---)).)))))))))))))))))))))))) ( -50.41) >consensus ACCAACGAUUGGGCAGAUUGAGAUCGUCCUGGGCCAGCUUCAUCUUUGCCGCCAGCUUGGCAUGCUGGCCGGCCG___AGGUGGCCUUCUCUCUGCCCAGCGUGG .(((.((.(((((((((..((((.......((((((...........(((((((((.......))))).))))........)))))))))))))))))))))))) (-35.53 = -34.95 + -0.58)

| Location | 6,334,441 – 6,334,543 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 81.49 |
| Mean single sequence MFE | -37.15 |
| Consensus MFE | -24.67 |
| Energy contribution | -25.28 |
| Covariance contribution | 0.61 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.742668 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 6334441 102 - 22407834 CCACACUGGGCAGGGAAAAGGCCACAA---CGGCAGGUCAGCACUCCAAGCUGGCGGCAAAGCUAAAGCUAGCCCAAGACGAUCUCAACCUGCCAAAUCGUUGGU (((.((..((((((......(((....---.)))..((((((.......))))))(((..(((....))).)))..............)))))).....))))). ( -33.10) >DroVir_CAF1 44534 102 - 1 CCACGCUCGGCAGAGAGAAGGCCAGCA---CAGCCGCCCAACAUGCCAAGCUGGCAACGAAAAUGAAGCUGGCCCAGGACGAUCUCAAUCUGCCGCAUCGUUGGU (((((..(((((((((((.(((((((.---.....(.....).((((.....))))...........)))))))(.....).))))..)))))))...)).))). ( -36.10) >DroPse_CAF1 64475 102 - 1 CCACGCUGGGCAGAGAGAAGGCCACCU---CCUCCAACCAGCAUGCAAAGCUGGCGGCAAAGAUGAAGCUGGCCCAAGACGAUCUCAAUCUGCCCAAUCGAUGGU (((((.((((((((((((.((((((.(---(.(((..(((((.......)))))..)....)).)).).)))))(.....).))))..))))))))..)).))). ( -37.10) >DroGri_CAF1 19173 105 - 1 CCACGCUCGGCAGAGAGAAGGCCAGCUCCUCGGCCGCCCAGCAAGCCAAGCUGGCGACCAAAAUGAAGCUGGCCCAGGACGAUCUAAAUCUGCCGCAUCGCUGGU (((((..(((((((.(((.((((((((.(..((.((((.(((.......))))))).)).....).))))))))(.....).)))...)))))))...)).))). ( -44.10) >DroAna_CAF1 26392 99 - 1 CCACGCUGGGCAGGGAGAAGGCCAC------GGCCGGGCAACACGCCAAGCUAGCGGCGAAGCUGAAACUGGCCCAGGAUGACCUCAACCUCCCCAAUCGUUGGU (((((.((((.(((..((.((((..------((((((.((.(.((((........))))..).))...))))))..))....))))..))).))))..)).))). ( -35.40) >DroPer_CAF1 62559 102 - 1 CCACGCUGGGCAGAGAGAAGGCCACCU---CCUCCAACCAGCAUGCAAAGCUGGCGGCAAAGAUGAAGCUGGCCCAAGACGAUCUCAAUCUGCCCAAUCGAUGGU (((((.((((((((((((.((((((.(---(.(((..(((((.......)))))..)....)).)).).)))))(.....).))))..))))))))..)).))). ( -37.10) >consensus CCACGCUGGGCAGAGAGAAGGCCACCU___CGGCCGACCAGCAUGCCAAGCUGGCGGCAAAGAUGAAGCUGGCCCAAGACGAUCUCAAUCUGCCCAAUCGUUGGU (((((..(((((((((((.(((..........)))..............((((((............)))))).........))))..)))))))...)).))). (-24.67 = -25.28 + 0.61)

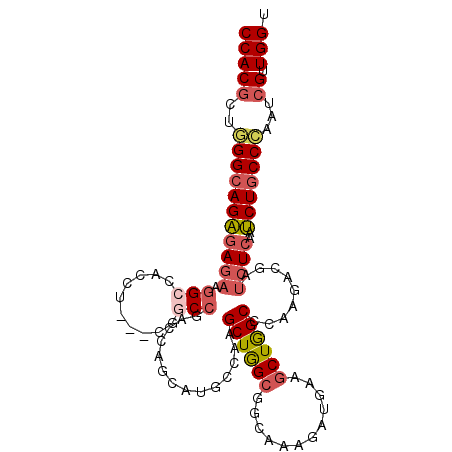

| Location | 6,334,472 – 6,334,583 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 81.07 |
| Mean single sequence MFE | -44.60 |
| Consensus MFE | -28.09 |
| Energy contribution | -27.71 |
| Covariance contribution | -0.38 |
| Combinations/Pair | 1.39 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.63 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.526443 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 6334472 111 + 22407834 GGCUAGCUUUAGCUUUGCCGCCAGCUUGGAGUGCUGACCUGCCG---UUGUGGCCUUUUCCCUGCCCAGUGUGGCGGAUUUCUGUUUGUCGGGUGUUUUGCUGCGUGACAAAAA (((.(((....)))..)))..((((.......))))..((((((---(((.(((.........))))))..)))))).......(((((((.((........)).))))))).. ( -34.60) >DroVir_CAF1 44565 111 + 1 GGCCAGCUUCAUUUUCGUUGCCAGCUUGGCAUGUUGGGCGGCUG---UGCUGGCCUUCUCUCUGCCGAGCGUGGCAGACUUUUGUUUAUCGGGCGUUUUGCUGCGCGACAGAAU (((((((..((........(((.(((..(....)..))))))))---.))))))).....((((....(((..((((((.((((.....)))).).)))))..)))..)))).. ( -42.40) >DroPse_CAF1 64506 111 + 1 GGCCAGCUUCAUCUUUGCCGCCAGCUUUGCAUGCUGGUUGGAGG---AGGUGGCCUUCUCUCUGCCCAGCGUGGCCGCCUUCUGUUUGUCGGGGGUCUUGCUGCGCGCCAAAAA (((((.((((.(((.....((((((.......)))))).)))))---)).)))))........((.(((((.((((.((..(.....)..)).)))).))))).))........ ( -47.60) >DroGri_CAF1 19204 114 + 1 GGCCAGCUUCAUUUUGGUCGCCAGCUUGGCUUGCUGGGCGGCCGAGGAGCUGGCCUUCUCUCUGCCGAGCGUGGCCGACUUUUGUUUGUCGGGCGUUUUGCUGCGCGACAAAAU ((((((((((...((((((((((((.......))).))))))))))))))))))).....((.((((....)))).))......(((((((.(((......))).))))))).. ( -56.70) >DroEre_CAF1 26074 111 + 1 AGCUAGCUUCAGCUUCGCCGCCAGCUUGGAGUGCUGGCCUGCCG---UCGUGGCCUUUUCCCUGCCCAGCGUGGCGGAUUUCUGUUUGUCGGGCGUUUUGCUACGUGAUAAGAA .((((((........((((((((((.......))))))((((((---.((((((.........)))..)))))))))..............))))....)))).))........ ( -36.90) >DroPer_CAF1 62590 111 + 1 GGCCAGCUUCAUCUUUGCCGCCAGCUUUGCAUGCUGGUUGGAGG---AGGUGGCCUUCUCUCUGCCCAGCGUGGCCGCCUUCUGUUUGUCGGGGGUCUUGCUGCGCGCCAGAAA (((((.((((.(((.....((((((.......)))))).)))))---)).)))))((((...(((.(((((.((((.((..(.....)..)).)))).))))).)))..)))). ( -49.40) >consensus GGCCAGCUUCAUCUUUGCCGCCAGCUUGGCAUGCUGGCCGGCCG___AGGUGGCCUUCUCUCUGCCCAGCGUGGCCGACUUCUGUUUGUCGGGCGUUUUGCUGCGCGACAAAAA (((((.(.........(((((((((.......))))).))))......).))))).............(((((((.(((..(((.....)))..)))..)))))))........ (-28.09 = -27.71 + -0.38)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:25:36 2006