
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 675,324 – 675,433 |
| Length | 109 |
| Max. P | 0.999939 |

| Location | 675,324 – 675,419 |
|---|---|
| Length | 95 |
| Sequences | 5 |
| Columns | 95 |
| Reading direction | forward |
| Mean pairwise identity | 96.42 |
| Mean single sequence MFE | -35.14 |
| Consensus MFE | -32.66 |
| Energy contribution | -33.26 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.40 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.03 |
| SVM RNA-class probability | 0.902782 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 675324 95 + 22407834 GGGGCGGUGCAAAAGGCCACGACGAUGGCGUCCCCGUCCCAAGUGAGCCAACUGAACCACCACAGGCGGUUCAAUCAGCAGCCCACCGGCACCAC (((.(((((.....((((((...(((((.....)))))....))).)))...((((((.((...)).))))))..........))))).).)).. ( -36.40) >DroSec_CAF1 63560 95 + 1 GGGGCGGUGCAAGAAGCCACGACGAUGGCGUCCCCGUCCCAAGUGAGCCAACUGAACCACCACAGGCGGUUCAAUCAGCAGCCCACCGGCACCAC (((((((.(...((.((((......)))).))))))))))..(((.((....((((((.((...)).))))))....)).(((....)))..))) ( -35.50) >DroSim_CAF1 68319 95 + 1 GGGGCGGUGCAAGAAGCCACGACGAUGGCGUCCCCGUCCCAAGUGAGCCAACUGAACCACCACAGGCGGUUCAAUCAGCAGCCCACCGGCACCAC (((((((.(...((.((((......)))).))))))))))..(((.((....((((((.((...)).))))))....)).(((....)))..))) ( -35.50) >DroEre_CAF1 72733 95 + 1 GGGGCGGUGCAAGAAGCCACGAUGAUGGCGUCCCCGUCCCAAGUGAGCCAACUGAACCACCACAGGCGGUUCAAUCAGCAGCCCACCGGCACCAC (((((((.(...((.((((......)))).))))))))))..(((.((....((((((.((...)).))))))....)).(((....)))..))) ( -35.50) >DroYak_CAF1 68584 95 + 1 GGGGCGGUGCAAGAAGCCACGAUGAUGGCGUCCGCGUCCCAAGCGAGCCAACUGAACCACCACGGGCGGUUCAUACAGCAGCCCACCGGCACCAC (((.(((((...((.((((......)))).))(((.......))).((....((((((.((...)).))))))....))....))))).).)).. ( -32.80) >consensus GGGGCGGUGCAAGAAGCCACGACGAUGGCGUCCCCGUCCCAAGUGAGCCAACUGAACCACCACAGGCGGUUCAAUCAGCAGCCCACCGGCACCAC (((((((.....((.((((......)))).)).)))))))..(((.((....((((((.((...)).))))))....)).(((....)))..))) (-32.66 = -33.26 + 0.60)



| Location | 675,324 – 675,419 |
|---|---|
| Length | 95 |
| Sequences | 5 |
| Columns | 95 |
| Reading direction | reverse |
| Mean pairwise identity | 96.42 |
| Mean single sequence MFE | -47.62 |
| Consensus MFE | -46.12 |
| Energy contribution | -46.40 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.65 |
| Structure conservation index | 0.97 |
| SVM decision value | 4.69 |
| SVM RNA-class probability | 0.999939 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 675324 95 - 22407834 GUGGUGCCGGUGGGCUGCUGAUUGAACCGCCUGUGGUGGUUCAGUUGGCUCACUUGGGACGGGGACGCCAUCGUCGUGGCCUUUUGCACCGCCCC (((((((....(((((((..(((((((((((...)))))))))))..)).......((.((....))))........)))))...)))))))... ( -49.00) >DroSec_CAF1 63560 95 - 1 GUGGUGCCGGUGGGCUGCUGAUUGAACCGCCUGUGGUGGUUCAGUUGGCUCACUUGGGACGGGGACGCCAUCGUCGUGGCUUCUUGCACCGCCCC ((((((((((((((((...((((((((((((...))))))))))))))))))).)))..((((((.(((((....)))))))))))))))))... ( -48.40) >DroSim_CAF1 68319 95 - 1 GUGGUGCCGGUGGGCUGCUGAUUGAACCGCCUGUGGUGGUUCAGUUGGCUCACUUGGGACGGGGACGCCAUCGUCGUGGCUUCUUGCACCGCCCC ((((((((((((((((...((((((((((((...))))))))))))))))))).)))..((((((.(((((....)))))))))))))))))... ( -48.40) >DroEre_CAF1 72733 95 - 1 GUGGUGCCGGUGGGCUGCUGAUUGAACCGCCUGUGGUGGUUCAGUUGGCUCACUUGGGACGGGGACGCCAUCAUCGUGGCUUCUUGCACCGCCCC ((((((((((((((((...((((((((((((...))))))))))))))))))).)))..((((((.(((((....)))))))))))))))))... ( -48.40) >DroYak_CAF1 68584 95 - 1 GUGGUGCCGGUGGGCUGCUGUAUGAACCGCCCGUGGUGGUUCAGUUGGCUCGCUUGGGACGCGGACGCCAUCAUCGUGGCUUCUUGCACCGCCCC (((((((.((.((((..(....(((((((((...)))))))))(.((((((((.......))))..)))).)...)..)))))).)))))))... ( -43.90) >consensus GUGGUGCCGGUGGGCUGCUGAUUGAACCGCCUGUGGUGGUUCAGUUGGCUCACUUGGGACGGGGACGCCAUCGUCGUGGCUUCUUGCACCGCCCC (((((((.((((((((...((((((((((((...)))))))))))))))))))).......((((.(((((....))))).)))))))))))... (-46.12 = -46.40 + 0.28)


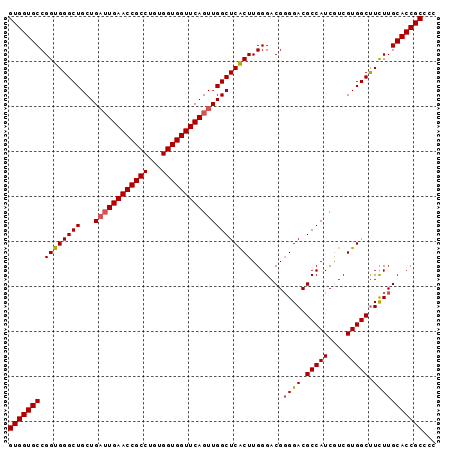
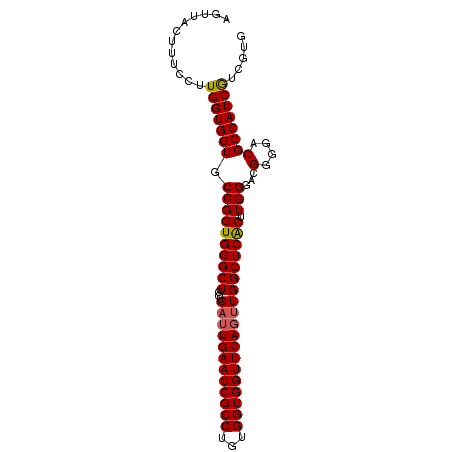
| Location | 675,341 – 675,433 |
|---|---|
| Length | 92 |
| Sequences | 5 |
| Columns | 92 |
| Reading direction | reverse |
| Mean pairwise identity | 96.74 |
| Mean single sequence MFE | -40.60 |
| Consensus MFE | -38.28 |
| Energy contribution | -38.28 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.53 |
| Structure conservation index | 0.94 |
| SVM decision value | 4.10 |
| SVM RNA-class probability | 0.999795 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 675341 92 - 22407834 AGUUACUUUCCUUGGUGGUGCCGGUGGGCUGCUGAUUGAACCGCCUGUGGUGGUUCAGUUGGCUCACUUGGGACGGGGACGCCAUCGUCGUG .....((.((((.(((((.(((....))).((..(((((((((((...)))))))))))..))))))).)))).)).((((....))))... ( -41.80) >DroSec_CAF1 63577 92 - 1 AGUUACUUUCCUUGGUGGUGCCGGUGGGCUGCUGAUUGAACCGCCUGUGGUGGUUCAGUUGGCUCACUUGGGACGGGGACGCCAUCGUCGUG .....((.((((.(((((.(((....))).((..(((((((((((...)))))))))))..))))))).)))).)).((((....))))... ( -41.80) >DroSim_CAF1 68336 92 - 1 AGUUACUUUCCUUGGUGGUGCCGGUGGGCUGCUGAUUGAACCGCCUGUGGUGGUUCAGUUGGCUCACUUGGGACGGGGACGCCAUCGUCGUG .....((.((((.(((((.(((....))).((..(((((((((((...)))))))))))..))))))).)))).)).((((....))))... ( -41.80) >DroEre_CAF1 72750 92 - 1 ACUUACUUUCCUUGGUGGUGCCGGUGGGCUGCUGAUUGAACCGCCUGUGGUGGUUCAGUUGGCUCACUUGGGACGGGGACGCCAUCAUCGUG ............(((((((.((((((((((...((((((((((((...))))))))))))))))))).)))...(....))))))))..... ( -40.30) >DroYak_CAF1 68601 92 - 1 AGUUACUUUCCUUGGUGGUGCCGGUGGGCUGCUGUAUGAACCGCCCGUGGUGGUUCAGUUGGCUCGCUUGGGACGCGGACGCCAUCAUCGUG ............(((((((((((((((((.(((...(((((((((...)))))))))...)))..))))...)).))).))))))))..... ( -37.30) >consensus AGUUACUUUCCUUGGUGGUGCCGGUGGGCUGCUGAUUGAACCGCCUGUGGUGGUUCAGUUGGCUCACUUGGGACGGGGACGCCAUCGUCGUG ............(((((((.((((((((((...((((((((((((...))))))))))))))))))).)))...(....))))))))..... (-38.28 = -38.28 + 0.00)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:30:43 2006