
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 6,302,288 – 6,302,422 |
| Length | 134 |
| Max. P | 0.996879 |

| Location | 6,302,288 – 6,302,404 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.76 |
| Mean single sequence MFE | -32.47 |
| Consensus MFE | -26.25 |
| Energy contribution | -28.67 |
| Covariance contribution | 2.42 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.37 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.50 |
| SVM RNA-class probability | 0.959249 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
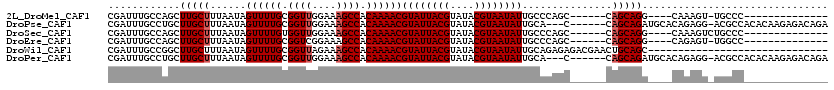
>2L_DroMel_CAF1 6302288 116 + 22407834 CU-UCUGGCACAUUUUAAUACGUUACGUUACGUAACAAUUCGAUUUGCCAGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGC--- ..-.((((((.(((.......((((((...)))))).....))).))))))...((.......((((((.((((....)))).))))))((((((((....)))))))).)).....--- ( -33.70) >DroPse_CAF1 247454 113 + 1 CU-CCUGGCACAUUUUAAUAUGUUACUCAACGUAACAAUUCGAUUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCA---C--- ..-...((((.(((..(((.((((((.....))))))))).))).))))....(((.......((((((.((((....)))).))))))((((((((....)))))))).)))---.--- ( -32.70) >DroSec_CAF1 138283 116 + 1 CU-UCUGGCACAUUUUAAUACGUUACGUUACGUAACAAUUCGAUUUGCCAGCUUGCUUUAAUAGUUUUGUGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGC--- ..-.((((((.(((.......((((((...)))))).....))).))))))...((.......(((((((((((....)))))))))))((((((((....)))))))).)).....--- ( -37.60) >DroEre_CAF1 138271 116 + 1 CU-UCUGGCACAUUUUAAUACGUUACGUUACGUAACAAUUCGAUUUGCCAGCUUGCUUUAAUAGUUUUGCGGUCGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGC--- ..-.((((((.(((.......((((((...)))))).....))).))))))...((.......((((((.(((......))).))))))((((((((....)))))))).)).....--- ( -30.60) >DroWil_CAF1 251918 116 + 1 CUGUGUGGACAAUUUUGAUA----ACUAAACGUUACAAUUCGAUUUGCCGGCUUGCUUUAAUAGUUUUGCGGUUAGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCAGAGAGAC (((.(..((.....(((.((----((.....))))))).....))..)))).((((.......((((((.((((....)))).))))))((((((((....)))))))).))))...... ( -27.50) >DroPer_CAF1 246509 113 + 1 CU-CCUGGCACAUUUUAAUAUGUUACUCAACGUAACAAUUCGAUUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCA---C--- ..-...((((.(((..(((.((((((.....))))))))).))).))))....(((.......((((((.((((....)))).))))))((((((((....)))))))).)))---.--- ( -32.70) >consensus CU_UCUGGCACAUUUUAAUACGUUACGUAACGUAACAAUUCGAUUUGCCAGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCACAGC___ ....((((((.(((.......(((((.....))))).....))).))))))...((.......((((((.((((....)))).))))))((((((((....)))))))).))........ (-26.25 = -28.67 + 2.42)

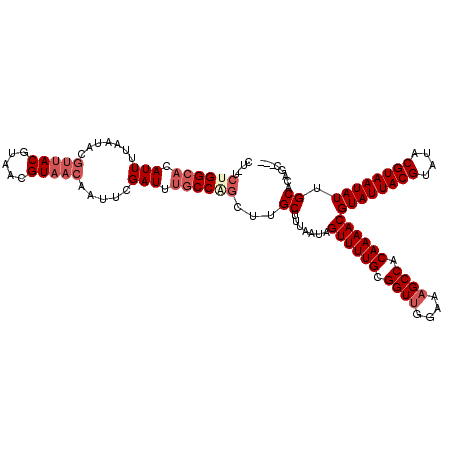
| Location | 6,302,288 – 6,302,404 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.76 |
| Mean single sequence MFE | -33.55 |
| Consensus MFE | -29.06 |
| Energy contribution | -29.75 |
| Covariance contribution | 0.69 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.53 |
| Structure conservation index | 0.87 |
| SVM decision value | 2.76 |
| SVM RNA-class probability | 0.996879 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 6302288 116 - 22407834 ---GCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCUGGCAAAUCGAAUUGUUACGUAACGUAACGUAUUAAAAUGUGCCAGA-AG ---(((..((..(((((((....))))))).(((((((((........))))))))).......)).)))(((((.....((((((((((...))))))).)))......)))))..-.. ( -36.40) >DroPse_CAF1 247454 113 - 1 ---G---UGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCAGGCAAAUCGAAUUGUUACGUUGAGUAACAUAUUAAAAUGUGCCAGG-AG ---.---(((..(((((((....))))))).(((((((((........))))))))).......)))....((((.....(((((((((.....)))))).)))......))))...-.. ( -33.10) >DroSec_CAF1 138283 116 - 1 ---GCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCACAAAACUAUUAAAGCAAGCUGGCAAAUCGAAUUGUUACGUAACGUAACGUAUUAAAAUGUGCCAGA-AG ---(((..((..(((((((....))))))).(((((((((........))))))))).......)).)))(((((.....((((((((((...))))))).)))......)))))..-.. ( -36.80) >DroEre_CAF1 138271 116 - 1 ---GCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCGACCGCAAAACUAUUAAAGCAAGCUGGCAAAUCGAAUUGUUACGUAACGUAACGUAUUAAAAUGUGCCAGA-AG ---(((..((..(((((((....))))))).(((((((((........))))))))).......)).)))(((((.....((((((((((...))))))).)))......)))))..-.. ( -36.40) >DroWil_CAF1 251918 116 - 1 GUCUCUCUGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCUAACCGCAAAACUAUUAAAGCAAGCCGGCAAAUCGAAUUGUAACGUUUAGU----UAUCAAAAUUGUCCACACAG (((.((.(((..(((((((....))))))).(((((((((........))))))))).......)))))..)))........(((((((.....))----)).))).............. ( -25.50) >DroPer_CAF1 246509 113 - 1 ---G---UGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCAGGCAAAUCGAAUUGUUACGUUGAGUAACAUAUUAAAAUGUGCCAGG-AG ---.---(((..(((((((....))))))).(((((((((........))))))))).......)))....((((.....(((((((((.....)))))).)))......))))...-.. ( -33.10) >consensus ___GCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCUGGCAAAUCGAAUUGUUACGUAAAGUAACGUAUUAAAAUGUGCCAGA_AG .......(((..(((((((....))))))).(((((((((........)))))))))...........)))((((.....(((((((((.....)))))).)))......))))...... (-29.06 = -29.75 + 0.69)

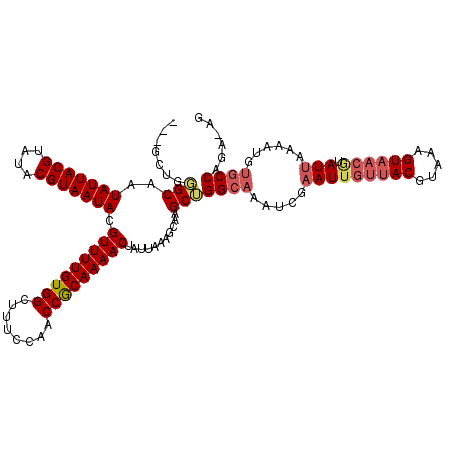
| Location | 6,302,327 – 6,302,422 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.57 |
| Mean single sequence MFE | -32.45 |
| Consensus MFE | -20.78 |
| Energy contribution | -22.12 |
| Covariance contribution | 1.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.13 |
| Structure conservation index | 0.64 |
| SVM decision value | 2.03 |
| SVM RNA-class probability | 0.985973 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 6302327 95 + 22407834 CGAUUUGCCAGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGC------CAGCAGG----CAAAGU-UGCCC-------------- ...((((((.(((.(((......((((((.((((....)))).))))))((((((((....)))))))).....)))------.))).))----))))..-.....-------------- ( -35.10) >DroPse_CAF1 247493 110 + 1 CGAUUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCA---C------CAGCAGAUGCACAGAGG-ACGCCACACAAGAGACAGA .....(((((((((((.......((((((.((((....)))).))))))((((((((....)))))))).)))---.------.)))))..)))......-................... ( -30.70) >DroSec_CAF1 138322 96 + 1 CGAUUUGCCAGCUUGCUUUAAUAGUUUUGUGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGC------CAGCAGG----CAAAGUCUGCCC-------------- .((((((((.(((.(((......(((((((((((....)))))))))))((((((((....)))))))).....)))------.))).))----).))))).....-------------- ( -39.60) >DroEre_CAF1 138310 95 + 1 CGAUUUGCCAGCUUGCUUUAAUAGUUUUGCGGUCGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCCCAGC------CAGCAGG----CAGAGU-UGGCC-------------- ......((((((((((.......((((((.(((......))).))))))((((((((....)))))))).))...((------(....))----).))))-)))).-------------- ( -34.00) >DroWil_CAF1 251954 90 + 1 CGAUUUGCCGGCUUGCUUUAAUAGUUUUGCGGUUAGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCAGAGAGACGAACUGCAGC------------------------------ ....((((.((.(((((((....((((((.((((....)))).))))))((((((((....)))))))).....)))).))).)))))).------------------------------ ( -24.60) >DroPer_CAF1 246548 110 + 1 CGAUUUGCCUGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCA---C------CAGCAGAUGCACAGAGG-ACGCCACACAAGAGACAGA .....(((((((((((.......((((((.((((....)))).))))))((((((((....)))))))).)))---.------.)))))..)))......-................... ( -30.70) >consensus CGAUUUGCCAGCUUGCUUUAAUAGUUUUGCGGUUGGAAAGCCACAAAACGUAUUACGUAUACGUAAUAUUGCACAGC______CAGCAGG____CAGAGU_UGGCC______________ ............(((((......((((((.((((....)))).))))))((((((((....))))))))...............)))))............................... (-20.78 = -22.12 + 1.33)
| Location | 6,302,327 – 6,302,422 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.57 |
| Mean single sequence MFE | -34.22 |
| Consensus MFE | -21.31 |
| Energy contribution | -21.17 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.68 |
| Structure conservation index | 0.62 |
| SVM decision value | 2.24 |
| SVM RNA-class probability | 0.990975 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
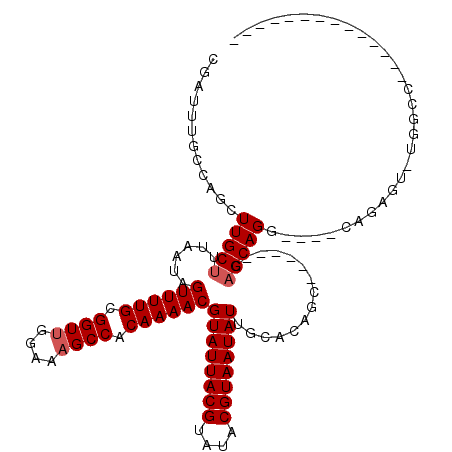
>2L_DroMel_CAF1 6302327 95 - 22407834 --------------GGGCA-ACUUUG----CCUGCUG------GCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCUGGCAAAUCG --------------(((((-....))----)))((..------(((..((..(((((((....))))))).(((((((((........))))))))).......)).)))..))...... ( -37.10) >DroPse_CAF1 247493 110 - 1 UCUGUCUCUUGUGUGGCGU-CCUCUGUGCAUCUGCUG------G---UGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCAGGCAAAUCG ..(((((((((((((((((-......((((((....)------)---)))).....)))).))).(((((.(((((((((........))))))))))))))..))))).)))))..... ( -36.00) >DroSec_CAF1 138322 96 - 1 --------------GGGCAGACUUUG----CCUGCUG------GCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCACAAAACUAUUAAAGCAAGCUGGCAAAUCG --------------((((((...)))----)))((..------(((..((..(((((((....))))))).(((((((((........))))))))).......)).)))..))...... ( -37.20) >DroEre_CAF1 138310 95 - 1 --------------GGCCA-ACUCUG----CCUGCUG------GCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCGACCGCAAAACUAUUAAAGCAAGCUGGCAAAUCG --------------(((..-.....)----))(((..------(((..((..(((((((....))))))).(((((((((........))))))))).......)).)))..)))..... ( -34.90) >DroWil_CAF1 251954 90 - 1 ------------------------------GCUGCAGUUCGUCUCUCUGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCUAACCGCAAAACUAUUAAAGCAAGCCGGCAAAUCG ------------------------------(((((((.........))))..(((((((....))))))).(((((((((........)))))))))......))).............. ( -24.10) >DroPer_CAF1 246548 110 - 1 UCUGUCUCUUGUGUGGCGU-CCUCUGUGCAUCUGCUG------G---UGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCAGGCAAAUCG ..(((((((((((((((((-......((((((....)------)---)))).....)))).))).(((((.(((((((((........))))))))))))))..))))).)))))..... ( -36.00) >consensus ______________GGCCA_ACUCUG____CCUGCUG______GCUGGGCAAUAUUACGUAUACGUAAUACGUUUUGUGGCUUUCCAACCGCAAAACUAUUAAAGCAAGCUGGCAAAUCG ................................((((...........(((..(((((((....))))))).(((((((((........)))))))))...........)))))))..... (-21.31 = -21.17 + -0.14)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:25:14 2006