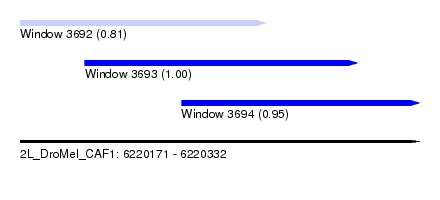
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 6,220,171 – 6,220,332 |
| Length | 161 |
| Max. P | 0.998196 |

| Location | 6,220,171 – 6,220,270 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 76.09 |
| Mean single sequence MFE | -24.00 |
| Consensus MFE | -8.29 |
| Energy contribution | -9.23 |
| Covariance contribution | 0.95 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.52 |
| Structure conservation index | 0.35 |
| SVM decision value | 0.66 |
| SVM RNA-class probability | 0.813930 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 6220171 99 + 22407834 AAGGCAAAAGCAAACAGCAAAACCAAAGUGAAGCAUGAGCAAUCUGCAAUGAAUUGCACAAAUUCUAUAUGC-----ACA-UAUAUGUGCAUGAUGGUUGGUUUU .........((.....))((((((((......(((.((....)))))...(((((.....)))))..(((((-----(((-....))))))))....)))))))) ( -24.40) >DroSec_CAF1 56787 104 + 1 AAGGCAAAAGCAAACAGCAAAACCAAAGUGAAGCAUGAGCAAUCUGCAAUGAAUUGCACAAAUGCUAUAUGCUGUAUGUA-UAUAUGUGCAUGAUGGUUGGUUUU .........((.....))((((((((.....((((((((((...(((((....)))))....)))).)))))).((((((-(....)))))))....)))))))) ( -27.00) >DroSim_CAF1 68410 104 + 1 AAGGCAAAAGCAAACAGCAAAACCAAAGUGAAGCAUGAGCAAUCUGCAAUGAAUUGCACAAAUGCUCUAUGCUAUAUGUA-UGUAUGUGCAUGAUGGUUGGUUUU .........((.....))((((((((.....((((((((((...(((((....)))))....))))).))))).((((((-(....)))))))....)))))))) ( -29.50) >DroEre_CAF1 55434 97 + 1 AAGGCAAAAGCAAACAGCAAAACCAAAGUGAAGCAUGAGCAAUCUGCAAUGCAUUGCACAAAUGCCA-------UGUGUA-UGUAUUUGCAUGAUGGUUGGUUUU .........((.....))(((((((..(..(.((((..((.....)).)))).)..)......((((-------((((((-......))))).)))))))))))) ( -25.00) >DroYak_CAF1 68168 89 + 1 AAGGCAAAAGCAAACAGCAAAACCAAAGUGAAGCAUGAGCAAUG--CACUAU-------AUGUACAA-------UGUGUACUAUAUGUGCAUGAUGGUUGGUUUU .........((.....))((((((((......((....)).(((--((((((-------(.((((..-------...)))))))).)))))).....)))))))) ( -22.90) >DroAna_CAF1 27788 82 + 1 AUGGCGAAAGUAAACAGCACAACCAAACUGGAGCCAAAGCUAUUUGCGAUGCAUGGCACAAAUGCUAUA-----------------------AAUUCUUGUUUUU ...((....))(((((((((..((.....)).......((.....))).)))((((((....)))))).-----------------------......))))).. ( -15.20) >consensus AAGGCAAAAGCAAACAGCAAAACCAAAGUGAAGCAUGAGCAAUCUGCAAUGAAUUGCACAAAUGCUAUA_____U_UGUA_UAUAUGUGCAUGAUGGUUGGUUUU ...((....)).......((((((((......((....))....(((((....))))).......................................)))))))) ( -8.29 = -9.23 + 0.95)

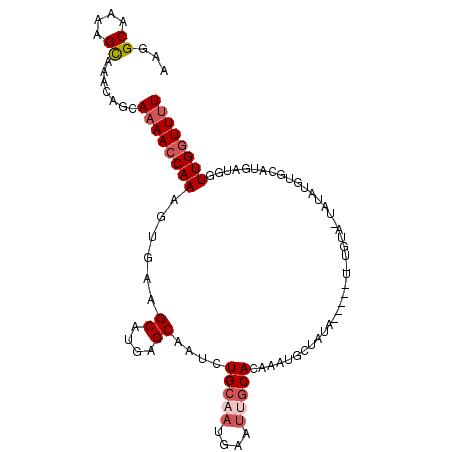

| Location | 6,220,197 – 6,220,307 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 75.53 |
| Mean single sequence MFE | -30.97 |
| Consensus MFE | -14.39 |
| Energy contribution | -16.37 |
| Covariance contribution | 1.98 |
| Combinations/Pair | 1.21 |
| Mean z-score | -3.51 |
| Structure conservation index | 0.46 |
| SVM decision value | 3.03 |
| SVM RNA-class probability | 0.998196 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 6220197 110 + 22407834 AGUGAAGCAUGAGCAAUCUGCAAUGAAUUGCACAAAUUCUAUAUGC-----ACA-UAUAUGUGCAUGAUGGUUGGUUUUGGCCAAAAUAAAUAUGAUCAAAGCUGAUUAUUUACAG .((((((((.((....)))))...(((((.....)))))..(((((-----(((-....)))))))).((((..((((((..((.........))..))))))..))))))))).. ( -30.60) >DroSec_CAF1 56813 115 + 1 AGUGAAGCAUGAGCAAUCUGCAAUGAAUUGCACAAAUGCUAUAUGCUGUAUGUA-UAUAUGUGCAUGAUGGUUGGUUUUGGCCAAAAUAAAUUUGAUCAAAGCUGAUUAUUUACAG .((((((((((((((...(((((....)))))....)))).)))))).((((((-(....)))))))(((((..((((((..((((.....))))..))))))..))))))))).. ( -35.50) >DroSim_CAF1 68436 115 + 1 AGUGAAGCAUGAGCAAUCUGCAAUGAAUUGCACAAAUGCUCUAUGCUAUAUGUA-UGUAUGUGCAUGAUGGUUGGUUUUGGCCAAAAUGAAUUUGAUCAAAGCUGAUUAUUUACAG .((((((((((((((...(((((....)))))....))))).))))).((((((-(....)))))))(((((..((((((..((((.....))))..))))))..))))))))).. ( -38.00) >DroEre_CAF1 55460 108 + 1 AGUGAAGCAUGAGCAAUCUGCAAUGCAUUGCACAAAUGCCA-------UGUGUA-UGUAUUUGCAUGAUGGUUGGUUUUGGCCAAAAUAAAUUUGAUCAAAGCUGAUUAUUUACAG .(((((((....)).(((((((((((((.(((((.......-------))))))-)))).))))).)))(((..((((((..((((.....))))..))))))..))).))))).. ( -32.40) >DroYak_CAF1 68194 100 + 1 AGUGAAGCAUGAGCAAUG--CACUAU-------AUGUACAA-------UGUGUACUAUAUGUGCAUGAUGGUUGGUUUUGGCCAAAAUAAAUUUGAUCAAAGCUGAUUAUUUACAG .(((((((....)).(((--((((((-------(.((((..-------...)))))))).))))))..((((..((((((..((((.....))))..))))))..))))))))).. ( -31.70) >DroAna_CAF1 27814 89 + 1 ACUGGAGCCAAAGCUAUUUGCGAUGCAUGGCACAAAUGCUAUA-----------------------AAUUCUUGUUUUUGGACAAA----AAUUGAUCAAAGCAUUUUGUUUGCAC ............((.....))..((((.((((.(((((((...-----------------------((((.(((((....))))).----))))......))))))))))))))). ( -17.60) >consensus AGUGAAGCAUGAGCAAUCUGCAAUGAAUUGCACAAAUGCUAUA_____U_UGUA_UAUAUGUGCAUGAUGGUUGGUUUUGGCCAAAAUAAAUUUGAUCAAAGCUGAUUAUUUACAG .(((((((((..((((((......).)))))....)))).............................((((..((((((..((((.....))))..))))))..))))))))).. (-14.39 = -16.37 + 1.98)

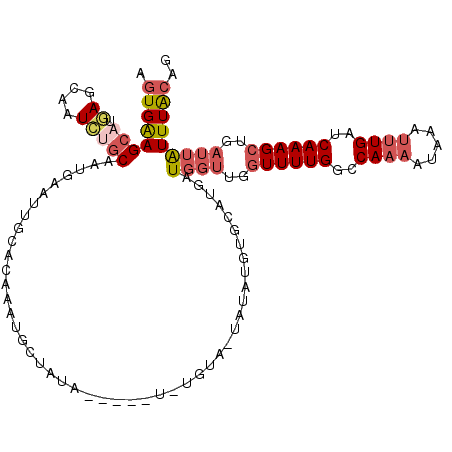

| Location | 6,220,236 – 6,220,332 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 80.38 |
| Mean single sequence MFE | -28.40 |
| Consensus MFE | -17.52 |
| Energy contribution | -19.05 |
| Covariance contribution | 1.53 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.87 |
| Structure conservation index | 0.62 |
| SVM decision value | 1.36 |
| SVM RNA-class probability | 0.946102 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 6220236 96 + 22407834 UAUAUGC-----ACA-UAUAUGUGCAUGAUGGUUGGUUUUGGCCAAAAUAAAUAUGAUCAAAGCUGAUUAUUUACAGAGGGCAGCC-----GGAGUUGUUGACCCAA ..(((((-----(((-....))))))))(((((..((((((..((.........))..))))))..))))).......(((((((.-----......)))).))).. ( -29.60) >DroVir_CAF1 45451 90 + 1 ------------AUAACAUAUGG-C--AAUGUUUUG--UUGGUCAAAAUAGAUUUGAUCUAAGCUGAUUAUUUGCAUUGGCCAGCCAGGGCAAAGCAUUUGACCCAA ------------.........((-(--((....)))--))(((((((.......(((((......)))))(((((.((((....)))).)))))...)))))))... ( -22.80) >DroSec_CAF1 56852 101 + 1 UAUAUGCUGUAUGUA-UAUAUGUGCAUGAUGGUUGGUUUUGGCCAAAAUAAAUUUGAUCAAAGCUGAUUAUUUACAGAGGGCAGCC-----GGAGUUGUUGACCCAA ......(((((((((-(....))))).((((((..((((((..((((.....))))..))))))..))))))))))).(((((((.-----......)))).))).. ( -31.00) >DroSim_CAF1 68475 101 + 1 UCUAUGCUAUAUGUA-UGUAUGUGCAUGAUGGUUGGUUUUGGCCAAAAUGAAUUUGAUCAAAGCUGAUUAUUUACAGAGGGCAGCC-----GGAGUUGUUGACCCAA ((((((((((((...-.))))).))))((((((..((((((..((((.....))))..))))))..))))))...)))(((((((.-----......)))).))).. ( -29.90) >DroEre_CAF1 55499 94 + 1 CA-------UGUGUA-UGUAUUUGCAUGAUGGUUGGUUUUGGCCAAAAUAAAUUUGAUCAAAGCUGAUUAUUUACAGAGGGCAGCC-----AGAGCUGUUGACCCAA ..-------.(((((-......)))))((((((..((((((..((((.....))))..))))))..))))))......(((((((.-----......)))).))).. ( -27.10) >DroYak_CAF1 68224 95 + 1 AA-------UGUGUACUAUAUGUGCAUGAUGGUUGGUUUUGGCCAAAAUAAAUUUGAUCAAAGCUGAUUAUUUACAGAGGGCAGCC-----AGAGUUGUUGACCCAA ..-------.((((((.....))))))((((((..((((((..((((.....))))..))))))..))))))......(((((((.-----......)))).))).. ( -30.00) >consensus UA_______U_UGUA_UAUAUGUGCAUGAUGGUUGGUUUUGGCCAAAAUAAAUUUGAUCAAAGCUGAUUAUUUACAGAGGGCAGCC_____AGAGUUGUUGACCCAA ....................((((...((((((..((((((..((((.....))))..))))))..))))))))))..(((((((............)))).))).. (-17.52 = -19.05 + 1.53)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:24:27 2006