
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 5,580,573 – 5,580,718 |
| Length | 145 |
| Max. P | 0.982979 |

| Location | 5,580,573 – 5,580,686 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 76.35 |
| Mean single sequence MFE | -47.57 |
| Consensus MFE | -35.24 |
| Energy contribution | -35.47 |
| Covariance contribution | 0.22 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.37 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.99 |
| SVM RNA-class probability | 0.895783 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
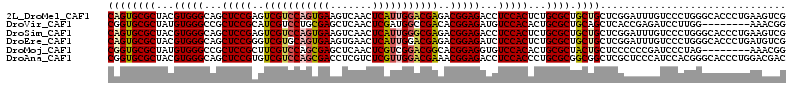
>2L_DroMel_CAF1 5580573 113 + 22407834 CGACUUCAGGGUGCCCAGGGACAAAUCCGAGCAGCAGCGCAGAGUGGAGGUCUCCGUCUCGUCCAAUGAGUUGACUUCACUGGACGACUCGGAGCUGCCCACGUAGCGCACUG ......((((.(((...(((.....)))..))).).((((...((((.((.(((((..(((((((.((((.....)))).)))))))..)))))))..))))...)))).))) ( -47.90) >DroVir_CAF1 35749 105 + 1 CCGUUU--------CCAAGGAUCUCGGUGAGCUGCAGCGCAGUGUGGACAUCUCCGUGUCGGCCAUCGAGUUGAGCUCGCAGGACGAUGCGGAGCGGCCCACAUAGCGCACCG (((..(--------(....))...))).........((((.((((((.(..((((((((((.((..((((.....))))..)).))))))))))..).)))))).)))).... ( -47.10) >DroSim_CAF1 26300 113 + 1 CGACUUCAGGGUGCCCAGGGACAAAUCCGAGCAGCAGCGCAGAGUGGAGGUCUCCGUCUCGCCCAAUGAGUUGACUUCACUGGACGACUCGGAGCUGCCCACGUAGCGCACUG ......((((.(((...(((.....)))..))).).((((...((((.((.(((((..(((.(((.((((.....)))).))).)))..)))))))..))))...)))).))) ( -42.90) >DroEre_CAF1 24661 113 + 1 CGACAUCAGGGUGCCCAGGGACAAAUCCGAGCAGCAGCGCAGAGUGGAGAUCUCCGUCUCGUCCAAUGAGUUCACUUCACUGCACGACCCGGAGCUGCCCACGUAGCGCACUG ......((((.(((...(((.....)))..))).).((((...((((.(..(((((..((((.((.((((.....)))).)).))))..)))))...)))))...)))).))) ( -39.30) >DroMoj_CAF1 46062 105 + 1 CCGUUU--------CUAGGGAUCGGGGGGAGCAGUAGCGCAGUGUGGACACCUCCGUGCCGUCCGACGAGUUGAGCUCGCUGGACGAAGCGGAGCGGCCCACAUAGCGCACCG ..((((--------((.(....)...))))))....((((.((((((.(..((((((..((((((.((((.....)))).))))))..))))))..).)))))).)))).... ( -51.70) >DroAna_CAF1 26475 113 + 1 GUCGUCCAGGGUGCCCGUGGAUGGGAGCGAGCCGCCGCGCAGGGUGGAGGUCUCCGUUUCGUCCAACGAGACGAGGUCGCUGGACGACACGGAGCUGCCCACGUAGCGCACCG ..((((((((....)).))))))((.(((...))).((((.(.((((.((.((((((.(((((((.(((.......))).))))))).))))))))..)))).).)))).)). ( -56.50) >consensus CGACUUCAGGGUGCCCAGGGACAAAUCCGAGCAGCAGCGCAGAGUGGAGAUCUCCGUCUCGUCCAACGAGUUGACUUCACUGGACGACGCGGAGCUGCCCACGUAGCGCACCG ....................................((((...((((....(((((..(((((((.((((.....)))).)))))))..)))))....))))...)))).... (-35.24 = -35.47 + 0.22)



| Location | 5,580,573 – 5,580,686 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 76.35 |
| Mean single sequence MFE | -50.68 |
| Consensus MFE | -40.74 |
| Energy contribution | -40.93 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.80 |
| SVM decision value | 1.93 |
| SVM RNA-class probability | 0.982979 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5580573 113 - 22407834 CAGUGCGCUACGUGGGCAGCUCCGAGUCGUCCAGUGAAGUCAACUCAUUGGACGAGACGGAGACCUCCACUCUGCGCUGCUGCUCGGAUUUGUCCCUGGGCACCCUGAAGUCG (((.((((...(((((...(((((..(((((((((((.......)))))))))))..)))))...)))))...))))...(((((((........)))))))..)))...... ( -51.50) >DroVir_CAF1 35749 105 - 1 CGGUGCGCUAUGUGGGCCGCUCCGCAUCGUCCUGCGAGCUCAACUCGAUGGCCGACACGGAGAUGUCCACACUGCGCUGCAGCUCACCGAGAUCCUUGG--------AAACGG ((((((((..(((((((..(((((..(((.((..((((.....))))..)).)))..)))))..)))))))..)))).))......(((((...)))))--------...)). ( -45.00) >DroSim_CAF1 26300 113 - 1 CAGUGCGCUACGUGGGCAGCUCCGAGUCGUCCAGUGAAGUCAACUCAUUGGGCGAGACGGAGACCUCCACUCUGCGCUGCUGCUCGGAUUUGUCCCUGGGCACCCUGAAGUCG (((.((((...(((((...(((((..(((((((((((.......)))))))))))..)))))...)))))...))))...(((((((........)))))))..)))...... ( -50.90) >DroEre_CAF1 24661 113 - 1 CAGUGCGCUACGUGGGCAGCUCCGGGUCGUGCAGUGAAGUGAACUCAUUGGACGAGACGGAGAUCUCCACUCUGCGCUGCUGCUCGGAUUUGUCCCUGGGCACCCUGAUGUCG (((.((((...(((((...(((((..((((.((((((.(....))))))).))))..)))))...)))))...))))...(((((((........)))))))..)))...... ( -50.30) >DroMoj_CAF1 46062 105 - 1 CGGUGCGCUAUGUGGGCCGCUCCGCUUCGUCCAGCGAGCUCAACUCGUCGGACGGCACGGAGGUGUCCACACUGCGCUACUGCUCCCCCCGAUCCCUAG--------AAACGG ((((((((..((((((((.(((((..((((((.(((((.....))))).))))))..)))))).)))))))..)))).))))......(((.((....)--------)..))) ( -50.20) >DroAna_CAF1 26475 113 - 1 CGGUGCGCUACGUGGGCAGCUCCGUGUCGUCCAGCGACCUCGUCUCGUUGGACGAAACGGAGACCUCCACCCUGCGCGGCGGCUCGCUCCCAUCCACGGGCACCCUGGACGAC ((.(((((...(((((...((((((.(((((((((((.......))))))))))).))))))...)))))...))))).))...........((((.((....)))))).... ( -56.20) >consensus CAGUGCGCUACGUGGGCAGCUCCGAGUCGUCCAGCGAACUCAACUCAUUGGACGAGACGGAGACCUCCACUCUGCGCUGCUGCUCGCAUUUAUCCCUGGGCACCCUGAAGUCG ((((((((...(((((...(((((..(((((((((((.......)))))))))))..)))))...)))))...)))).))))............................... (-40.74 = -40.93 + 0.20)

| Location | 5,580,606 – 5,580,718 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 83.67 |
| Mean single sequence MFE | -51.88 |
| Consensus MFE | -41.70 |
| Energy contribution | -43.20 |
| Covariance contribution | 1.50 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.75 |
| SVM RNA-class probability | 0.840465 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5580606 112 + 22407834 GCAGCGCAGAGUGGAGGUCUCCGUCUCGUCCAAUGAGUUGACUUCACUGGACGACUCGGAGCUGCCCACGUAGCGCACUGUGCUGGGGCUACCCAUAGAGCGCACGAGG--CUG ((.((((...((((.((.(((((..(((((((.((((.....)))).)))))))..)))))))..))))...))))..(((((((((....)))....))))))....)--).. ( -53.00) >DroVir_CAF1 35774 108 + 1 GCAGCGCAGUGUGGACAUCUCCGUGUCGGCCAUCGAGUUGAGCUCGCAGGACGAUGCGGAGCGGCCCACAUAGCGCACCGUGCUAGGACUA------GAGAGCAAGUGGCGCGG ...((((.((((((.(..((((((((((.((..((((.....))))..)).))))))))))..).)))))).)))).((((((((.(.((.------...)))...)))))))) ( -53.70) >DroSim_CAF1 26333 112 + 1 GCAGCGCAGAGUGGAGGUCUCCGUCUCGCCCAAUGAGUUGACUUCACUGGACGACUCGGAGCUGCCCACGUAGCGCACUGUGCUGGGGCUACCCAUAGAGCGCACUAUG--CUG (((((((...((((.((.(((((..(((.(((.((((.....)))).))).)))..)))))))..))))...))))..(((((((((....)))....))))))...))--).. ( -49.00) >DroEre_CAF1 24694 112 + 1 GCAGCGCAGAGUGGAGAUCUCCGUCUCGUCCAAUGAGUUCACUUCACUGCACGACCCGGAGCUGCCCACGUAGCGCACUGUGCUGGGGCUUCCCAUGCAGCGCACUAGG--CUG .((((((...((((.(..(((((..((((.((.((((.....)))).)).))))..)))))...)))))...))((.(((((.((((....))))))))).)).....)--))) ( -45.10) >DroYak_CAF1 24568 112 + 1 GCAGCGCAGAGUGGAGGUCUCCGUCUCGUCCAAUGAGUUGACUUCACUGGACGACCCGGAGCUGCCCACGUAGCGCACUGUGCUGGGGCUUCCCAUAGAGCGCACUAGG--CUG ((.((((...((((.((.(((((..(((((((.((((.....)))).)))))))..)))))))..))))...))))..(((((((((....)))....))))))....)--).. ( -52.00) >DroAna_CAF1 26508 112 + 1 GCCGCGCAGGGUGGAGGUCUCCGUUUCGUCCAACGAGACGAGGUCGCUGGACGACACGGAGCUGCCCACGUAGCGCACCGUGCUGGGACUGCCCAUGGCCGGCAGGAGG--CUC (((((((.(.((((.((.((((((.(((((((.(((.......))).))))))).))))))))..)))).).)))).((.((((((..(.......).)))))))).))--).. ( -58.50) >consensus GCAGCGCAGAGUGGAGGUCUCCGUCUCGUCCAAUGAGUUGACUUCACUGGACGACCCGGAGCUGCCCACGUAGCGCACUGUGCUGGGGCUACCCAUAGAGCGCACGAGG__CUG ...((((...((((.((.(((((..(((((((.((((.....)))).)))))))..)))))))..))))...))))...((((((((....)))....)))))........... (-41.70 = -43.20 + 1.50)


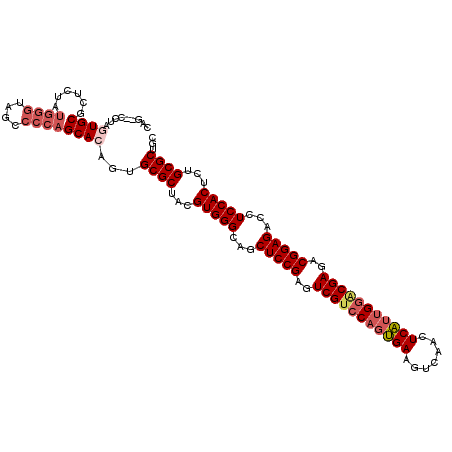
| Location | 5,580,606 – 5,580,718 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 83.67 |
| Mean single sequence MFE | -55.57 |
| Consensus MFE | -44.40 |
| Energy contribution | -45.52 |
| Covariance contribution | 1.11 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.70 |
| Structure conservation index | 0.80 |
| SVM decision value | 1.51 |
| SVM RNA-class probability | 0.959994 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5580606 112 - 22407834 CAG--CCUCGUGCGCUCUAUGGGUAGCCCCAGCACAGUGCGCUACGUGGGCAGCUCCGAGUCGUCCAGUGAAGUCAACUCAUUGGACGAGACGGAGACCUCCACUCUGCGCUGC (((--(...(((((((...((((....))))....)))))))...(((((...(((((..(((((((((((.......)))))))))))..)))))...))))).....)))). ( -54.10) >DroVir_CAF1 35774 108 - 1 CCGCGCCACUUGCUCUC------UAGUCCUAGCACGGUGCGCUAUGUGGGCCGCUCCGCAUCGUCCUGCGAGCUCAACUCGAUGGCCGACACGGAGAUGUCCACACUGCGCUGC (((.((.(((.......------.)))....)).))).((((..(((((((..(((((..(((.((..((((.....))))..)).)))..)))))..)))))))..))))... ( -45.10) >DroSim_CAF1 26333 112 - 1 CAG--CAUAGUGCGCUCUAUGGGUAGCCCCAGCACAGUGCGCUACGUGGGCAGCUCCGAGUCGUCCAGUGAAGUCAACUCAUUGGGCGAGACGGAGACCUCCACUCUGCGCUGC (((--(.(((((((((...((((....))))....))))))))).(((((...(((((..(((((((((((.......)))))))))))..)))))...))))).....)))). ( -56.60) >DroEre_CAF1 24694 112 - 1 CAG--CCUAGUGCGCUGCAUGGGAAGCCCCAGCACAGUGCGCUACGUGGGCAGCUCCGGGUCGUGCAGUGAAGUGAACUCAUUGGACGAGACGGAGAUCUCCACUCUGCGCUGC (((--(.((((((((((..((((....))))...)))))))))).(((((...(((((..((((.((((((.(....))))))).))))..)))))...))))).....)))). ( -58.60) >DroYak_CAF1 24568 112 - 1 CAG--CCUAGUGCGCUCUAUGGGAAGCCCCAGCACAGUGCGCUACGUGGGCAGCUCCGGGUCGUCCAGUGAAGUCAACUCAUUGGACGAGACGGAGACCUCCACUCUGCGCUGC (((--(.(((((((((...((((....))))....))))))))).(((((...(((((..(((((((((((.......)))))))))))..)))))...))))).....)))). ( -58.60) >DroAna_CAF1 26508 112 - 1 GAG--CCUCCUGCCGGCCAUGGGCAGUCCCAGCACGGUGCGCUACGUGGGCAGCUCCGUGUCGUCCAGCGACCUCGUCUCGUUGGACGAAACGGAGACCUCCACCCUGCGCGGC ..(--((....((((((..((((....)))))).))))((((...(((((...((((((.(((((((((((.......))))))))))).))))))...)))))...))))))) ( -60.40) >consensus CAG__CCUAGUGCGCUCUAUGGGUAGCCCCAGCACAGUGCGCUACGUGGGCAGCUCCGAGUCGUCCAGUGAAGUCAACUCAUUGGACGAGACGGAGACCUCCACUCUGCGCUGC .........((((......((((....))))))))...((((...(((((...(((((..(((((((((((.......)))))))))))..)))))...)))))...))))... (-44.40 = -45.52 + 1.11)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:19:04 2006