
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 5,492,145 – 5,492,279 |
| Length | 134 |
| Max. P | 0.810533 |

| Location | 5,492,145 – 5,492,240 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 96 |
| Reading direction | forward |
| Mean pairwise identity | 83.16 |
| Mean single sequence MFE | -21.65 |
| Consensus MFE | -14.83 |
| Energy contribution | -14.83 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.624183 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5492145 95 + 22407834 -CUGUGCACUGCACUACACUCUGUAUCGGUCGACAUGUAAUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCGUUAUGAU -..((((...))))...((.(((..((((((((((..((((((.(.(((.....)))).))))))...))))...))).)))..))).))...... ( -26.00) >DroPse_CAF1 131215 80 + 1 ----U------------CCACUCUACGGCUCGACAUAUAAUUGCGCGCUUUCCUAGCCGCAAUUAAAUUGUCAUAGACAUGACGCAGCAUUAUGAU ----.------------..........((((......((((((((.(((.....)))))))))))....(((((....)))))).)))........ ( -20.30) >DroEre_CAF1 113475 83 + 1 -CUGUGCAC------------UCUAUCGGUCGACAUGUAAUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCGUUAUGAU -(((((((.------------(((((.....((((..((((((.(.(((.....)))).))))))...)))))))))..)).)))))......... ( -20.60) >DroYak_CAF1 120331 95 + 1 -CUGUGCACUGCACCACACAUUCUAUCGCUCGACAUGUAAUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCAUUAUGAU -..((((..(((......((((((((.....((((..((((((.(.(((.....)))).))))))...))))))))).)))..)))))))...... ( -19.00) >DroAna_CAF1 121090 88 + 1 ACUGC-CACGG-------CUCUCUAUCGGCCGACAUGUAAUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCAUUAUGAU .((((-...((-------((.......))))((((..((((((.(.(((.....)))).))))))...))))...........))))......... ( -23.70) >DroPer_CAF1 131742 80 + 1 ----U------------CCACUCUACGGCUCGACAUAUAAUUGCGCGCUUUCCUAGCCGCAAUUAAAUUGUCAUAGACAUGACGCAGCAUUAUGAU ----.------------..........((((......((((((((.(((.....)))))))))))....(((((....)))))).)))........ ( -20.30) >consensus _CUGU_CAC_________CACUCUAUCGCUCGACAUGUAAUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCAUUAUGAU ...............................((((..((((((.(.(((.....)))).))))))...)))).....(((((.......))))).. (-14.83 = -14.83 + 0.00)

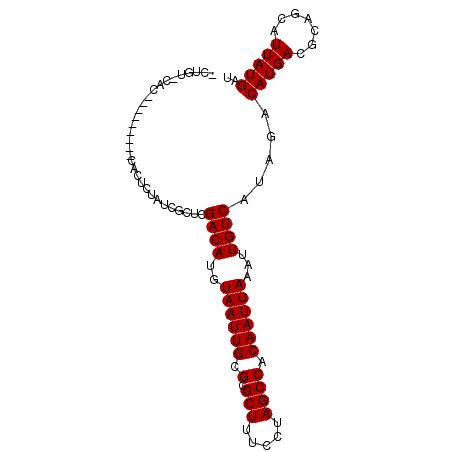
| Location | 5,492,145 – 5,492,240 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 96 |
| Reading direction | reverse |
| Mean pairwise identity | 83.16 |
| Mean single sequence MFE | -25.58 |
| Consensus MFE | -16.48 |
| Energy contribution | -15.98 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.64 |
| SVM decision value | -0.03 |
| SVM RNA-class probability | 0.517202 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5492145 95 - 22407834 AUCAUAACGCUGCGUCAUGUCUAUGACAAUUUAAUUGUGGCUAGGAAAGCGCGCAAUUACAUGUCGACCGAUACAGAGUGUAGUGCAGUGCACAG- ..(((..(((((((((.((((..(((((...(((((((((((.....))).))))))))..)))))...))))..)).)))))))..))).....- ( -26.40) >DroPse_CAF1 131215 80 - 1 AUCAUAAUGCUGCGUCAUGUCUAUGACAAUUUAAUUGCGGCUAGGAAAGCGCGCAAUUAUAUGUCGAGCCGUAGAGUGG------------A---- .((((..(((.(((((((....)))))....(((((((((((.....))).))))))))........)).)))..))))------------.---- ( -24.60) >DroEre_CAF1 113475 83 - 1 AUCAUAACGCUGCGUCAUGUCUAUGACAAUUUAAUUGUGGCUAGGAAAGCGCGCAAUUACAUGUCGACCGAUAGA------------GUGCACAG- ........(.((((((.((((..(((((...(((((((((((.....))).))))))))..)))))...))))))------------.)))))..- ( -21.90) >DroYak_CAF1 120331 95 - 1 AUCAUAAUGCUGCGUCAUGUCUAUGACAAUUUAAUUGUGGCUAGGAAAGCGCGCAAUUACAUGUCGAGCGAUAGAAUGUGUGGUGCAGUGCACAG- ........(((((..(((.(((((((((...(((((((((((.....))).))))))))..)))).....)))))....)))..)))))......- ( -28.30) >DroAna_CAF1 121090 88 - 1 AUCAUAAUGCUGCGUCAUGUCUAUGACAAUUUAAUUGUGGCUAGGAAAGCGCGCAAUUACAUGUCGGCCGAUAGAGAG-------CCGUG-GCAGU .......(((..(...........((((...(((((((((((.....))).))))))))..))))((((......).)-------)))..-))).. ( -27.70) >DroPer_CAF1 131742 80 - 1 AUCAUAAUGCUGCGUCAUGUCUAUGACAAUUUAAUUGCGGCUAGGAAAGCGCGCAAUUAUAUGUCGAGCCGUAGAGUGG------------A---- .((((..(((.(((((((....)))))....(((((((((((.....))).))))))))........)).)))..))))------------.---- ( -24.60) >consensus AUCAUAAUGCUGCGUCAUGUCUAUGACAAUUUAAUUGUGGCUAGGAAAGCGCGCAAUUACAUGUCGACCGAUAGAGUG_________GUG_ACAG_ ..(((.(((...))).)))(((((((((...(((((((((((.....))).))))))))..)))).....)))))..................... (-16.48 = -15.98 + -0.50)



| Location | 5,492,169 – 5,492,279 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.86 |
| Mean single sequence MFE | -30.35 |
| Consensus MFE | -19.16 |
| Energy contribution | -19.43 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.28 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.21 |
| SVM RNA-class probability | 0.637286 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5492169 110 + 22407834 U-----CGGUCGACAU----GUAAUUGCGCGCUUUCCUAGCCACAAUUAAAUUGUCAUAGACAUGACGCAGCGUUAUGAUGGGGCAAUUAUAACUGUCAGAUUUGACAGCAU-UACAGGC .-----..(((((((.----.((((((.(.(((.....)))).))))))...)))).....(((((((...)))))))....)))........(((((......)))))...-....... ( -28.60) >DroVir_CAF1 164119 109 + 1 G-----CGACCGACAU----GUAAUUGCGCGCUUUCCUAGCCACAAUUAAACUGUCAUAGACAUGACGCAGCAUUAUGAAGUCGCAAUUAUAGCUGUCACUUUUGACAGCAC--ACAUGC (-----((((.((((.----.((((((.(.(((.....)))).))))))...)))).....(((((.......)))))..))))).......(((((((....)))))))..--...... ( -33.60) >DroGri_CAF1 173656 103 + 1 ---------CUGACAU----CUAAUUGCGCGCUUUCCUAGCCACAAUUAAACUGUCAUAGACAUGACGCAGCAUUAUGACGUCGCAAUUAUAGCUGUCAUUUCUGACAGCCA--ACAG-- ---------.(((((.----.((((((.(.(((.....)))).))))))...))))).......((((((......)).)))).........(((((((....)))))))..--....-- ( -28.30) >DroWil_CAF1 163600 113 + 1 C-----CGCUCGACAUGUAUGUAAUUGCGCGAUUUUCUACCAACAAUUAAAUUGUCAUAGAGAUGACGAAGCAUUAUGACGGCGCAAUUAAAACUGUCAGAUUUGACAGCAU-UACU-AC .-----.(((.((((.((...((((((((((((((.............)))))((((((...(((......))))))))).)))))))))..)))))).(......))))..-....-.. ( -24.02) >DroMoj_CAF1 181755 116 + 1 GUGCUACGGUCGACAU----GUAAUUGCGCGCUUUCCUAGCCACAAUUAAACUGUCAUAGACAUGACGCAGCAUUAUGACGUCGCAAUUAUAGCUGUCACUUUUGACAGCAACUACACGC .(((....(((((((.----.((((((.(.(((.....)))).))))))...))))...)))..((((((......)).)))))))......(((((((....))))))).......... ( -33.90) >DroPer_CAF1 131751 109 + 1 C-----GGCUCGACAU----AUAAUUGCGCGCUUUCCUAGCCGCAAUUAAAUUGUCAUAGACAUGACGCAGCAUUAUGAUGGGGCAAUUAUAACUGUCAGAUCUGACAGCAU-UACG-GC (-----(((((.((((----(((((((((.(((.....)))))))))))....(((((....))))).......)))).).))))........((((((....))))))...-..))-.. ( -33.70) >consensus C_____CGCUCGACAU____GUAAUUGCGCGCUUUCCUAGCCACAAUUAAACUGUCAUAGACAUGACGCAGCAUUAUGACGGCGCAAUUAUAACUGUCAGAUUUGACAGCAU_UACA_GC ....................((((((((..(((.....)))...........(((((((...(((......))))))))))..))))))))..((((((....))))))........... (-19.16 = -19.43 + 0.28)


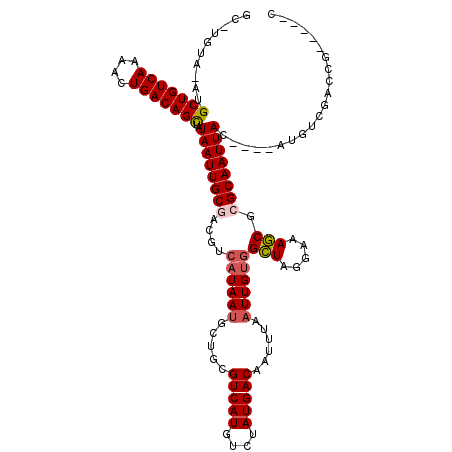
| Location | 5,492,169 – 5,492,279 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.86 |
| Mean single sequence MFE | -33.82 |
| Consensus MFE | -24.83 |
| Energy contribution | -25.13 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.65 |
| SVM RNA-class probability | 0.810533 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5492169 110 - 22407834 GCCUGUA-AUGCUGUCAAAUCUGACAGUUAUAAUUGCCCCAUCAUAACGCUGCGUCAUGUCUAUGACAAUUUAAUUGUGGCUAGGAAAGCGCGCAAUUAC----AUGUCGACCG-----A ....(((-(((((((((....)))))))....))))).....(((.(((...))).)))....(((((...(((((((((((.....))).)))))))).----.)))))....-----. ( -26.20) >DroVir_CAF1 164119 109 - 1 GCAUGU--GUGCUGUCAAAAGUGACAGCUAUAAUUGCGACUUCAUAAUGCUGCGUCAUGUCUAUGACAGUUUAAUUGUGGCUAGGAAAGCGCGCAAUUAC----AUGUCGGUCG-----C ((((((--..(((((((....)))))))...(((((((....((((((...(((((((....))))).))...))))))(((.....))).)))))))))----))))......-----. ( -35.40) >DroGri_CAF1 173656 103 - 1 --CUGU--UGGCUGUCAGAAAUGACAGCUAUAAUUGCGACGUCAUAAUGCUGCGUCAUGUCUAUGACAGUUUAAUUGUGGCUAGGAAAGCGCGCAAUUAG----AUGUCAG--------- --....--(((((((((....))))))))).......((((.((......)))))).......(((((.(((((((((((((.....))).)))))))))----)))))).--------- ( -39.50) >DroWil_CAF1 163600 113 - 1 GU-AGUA-AUGCUGUCAAAUCUGACAGUUUUAAUUGCGCCGUCAUAAUGCUUCGUCAUCUCUAUGACAAUUUAAUUGUUGGUAGAAAAUCGCGCAAUUACAUACAUGUCGAGCG-----G ((-((((-(.(((((((....))))))).)).)))))((((.(((........(((((....)))))....(((((((((((.....)))).))))))).....))).)).)).-----. ( -28.40) >DroMoj_CAF1 181755 116 - 1 GCGUGUAGUUGCUGUCAAAAGUGACAGCUAUAAUUGCGACGUCAUAAUGCUGCGUCAUGUCUAUGACAGUUUAAUUGUGGCUAGGAAAGCGCGCAAUUAC----AUGUCGACCGUAGCAC (((((((((((((((((....)))))))...)))))).)))).....(((((((....(((...((((...(((((((((((.....))).)))))))).----.)))))))))))))). ( -42.50) >DroPer_CAF1 131751 109 - 1 GC-CGUA-AUGCUGUCAGAUCUGACAGUUAUAAUUGCCCCAUCAUAAUGCUGCGUCAUGUCUAUGACAAUUUAAUUGCGGCUAGGAAAGCGCGCAAUUAU----AUGUCGAGCC-----G ..-.(((-(((((((((....)))))))....)))))...........(((.((.((((((...)))....(((((((((((.....))).)))))))).----))).))))).-----. ( -30.90) >consensus GC_UGUA_AUGCUGUCAAAACUGACAGCUAUAAUUGCGACGUCAUAAUGCUGCGUCAUGUCUAUGACAAUUUAAUUGUGGCUAGGAAAGCGCGCAAUUAC____AUGUCGACCG_____C ..........(((((((....)))))))..((((((((....((((((.....(((((....)))))......))))))(((.....))).))))))))..................... (-24.83 = -25.13 + 0.31)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:17:35 2006