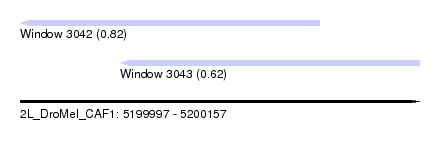
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 5,199,997 – 5,200,157 |
| Length | 160 |
| Max. P | 0.816177 |

| Location | 5,199,997 – 5,200,117 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.78 |
| Mean single sequence MFE | -58.97 |
| Consensus MFE | -31.75 |
| Energy contribution | -30.15 |
| Covariance contribution | -1.60 |
| Combinations/Pair | 1.58 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.66 |
| SVM RNA-class probability | 0.816177 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5199997 120 - 22407834 UUCGCAUGGCGGUGGCCAGCGCGUCGUCACAAUGCCUGGCCAGCUCCUGCUGCUGCUCCUGCAAAGCCUUGCUCUGUUGUUCUCCCUGAUCCAAUUGCUGGAUCAUGGCAUCCAGGCGGU ...(((.((.(.(((((((.((((.......))))))))))).).))))).(((((....((((....)))).(((.((....((.(((((((.....))))))).))))..)))))))) ( -46.20) >DroVir_CAF1 117149 120 - 1 UGCGCAUGGCGGUGGCCAGCGCAUCGUCGCAGUACUUGAGCAGCUCCUGCUGUUGGGCCUUCAGAGCGGCGCUCUGCUGCUCGCCACGUGUCAGCUGCAGCAGCAAUGCGUCCAGGCGCG .((((.((((((.((((.(((......)))........((((((....)))))).)))).)).(((((((.....)))))))))))........(((..(((....)))...))))))). ( -53.70) >DroPse_CAF1 96515 120 - 1 UGCGCAUGGCUGUGGCCAGGGCCUCGUCGCAGUGACGGGACAGCUGUUGCUGCUGCUCCUGCAGGGACUUGCUCUGCUGCUCGCCCUGGUCCAGCUGCUGGAGCAGACCGUCCAGGCGGC ..(((..(((((.(((((((((......((((..((((.....))))..)))).((....((((((.....)))))).))..)))))))))))))).(((((........)))))))).. ( -62.10) >DroMoj_CAF1 137702 120 - 1 UGCGCAUGGCCGUGGCCAGUGCAUCGUCGCAGUACUGCAGCAGCUGCUGCUGCUGCGCCUUCAGCGCUGCGCUCUGCUGCUCGCCGCGCGUCAGCUGCAGCAGCAGCGCGUCGAGGCGCG (((((.((((....)))))))))....(((......((((((((....))))))))(((((..(((((((((((.((((..(((...))).)))).).))).)))))))...)))))))) ( -69.60) >DroAna_CAF1 80244 120 - 1 UGCGCAUGGCAGUGGCCAGGGCAUCGUCGCAGUGGCGGGAUAGCUCCUGUUGCUGCUCCUGCAGAGCCUUGCUUUGCUGUUCGCCUUGGUCCAGUUGCUGGAGCAUGGCGUCAAGGCGGU .((.(.((((....))))).))((((((((((..((((((....))))))..))))....(((((((...)))))))((..((((.((.(((((...))))).)).)))).)).)))))) ( -60.10) >DroPer_CAF1 91136 120 - 1 UGCGCAUGGCUGUGGCCAGGGCCUCGUCGCAGUGACGGGACAGCUGUUGCUGCUGCUCCUGCAGGGACUUGCUCUGCUGCUCGCCCUGGUCCAGCUGCUGGAGCAGACCGUCCAGGCGGC ..(((..(((((.(((((((((......((((..((((.....))))..)))).((....((((((.....)))))).))..)))))))))))))).(((((........)))))))).. ( -62.10) >consensus UGCGCAUGGCGGUGGCCAGGGCAUCGUCGCAGUGCCGGGACAGCUCCUGCUGCUGCUCCUGCAGAGCCUUGCUCUGCUGCUCGCCCUGGUCCAGCUGCUGGAGCAGAGCGUCCAGGCGGC ...((.((((....))))..))......((((((.((((.((((....))))((((....))))...))))...)))))).((((.(((((....(((....))))))))....)))).. (-31.75 = -30.15 + -1.60)
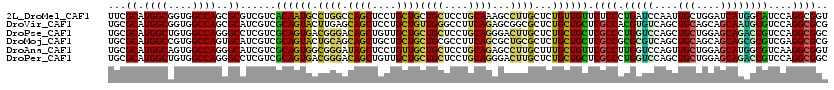
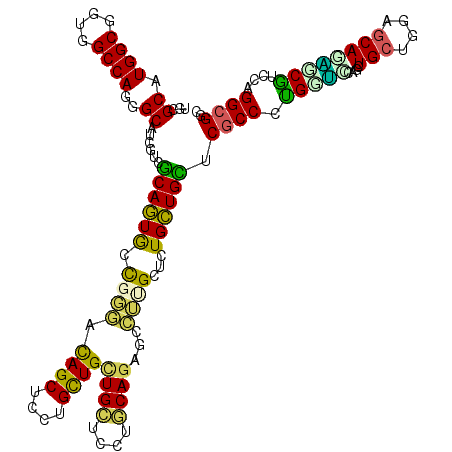
| Location | 5,200,037 – 5,200,157 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.61 |
| Mean single sequence MFE | -51.77 |
| Consensus MFE | -32.35 |
| Energy contribution | -33.80 |
| Covariance contribution | 1.45 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.621175 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5200037 120 - 22407834 CGCAGAUUGCUUAUCCGCUGACCAAUGCUGGCCUGCUCCAUUCGCAUGGCGGUGGCCAGCGCGUCGUCACAAUGCCUGGCCAGCUCCUGCUGCUGCUCCUGCAAAGCCUUGCUCUGUUGU .(((((..((......((((.(((..(((((((.(((((((....)))).))))))))))((((.......)))).))).))))....(((..(((....))).)))...)))))))... ( -43.50) >DroVir_CAF1 117189 120 - 1 CGCAAAUUGCUUAUACGCUGAUUGAUGCUCGCCUGCUCCAUGCGCAUGGCGGUGGCCAGCGCAUCGUCGCAGUACUUGAGCAGCUCCUGCUGUUGGGCCUUCAGAGCGGCGCUCUGCUGC .(((....((.....(((((.((((.((((..((((..(((((((.((((....)))))))))).)..))))......((((((....))))))))))..))))..)))))....))))) ( -46.70) >DroPse_CAF1 96555 120 - 1 CGCAGAUUGUUAAUCCGCUGCCCGAUGCUGGCCUGCUCCAUGCGCAUGGCUGUGGCCAGGGCCUCGUCGCAGUGACGGGACAGCUGUUGCUGCUGCUCCUGCAGGGACUUGCUCUGCUGC .(((((..((...(((.((((..((((..(((((((.....))...((((....))))))))).))))((((..((((.....))))..)))).......)))))))...)))))))... ( -52.30) >DroMoj_CAF1 137742 120 - 1 CGCAGAUUGCUGAUGCGCUGAUUGAUGCUCGCCUGCUCCAUGCGCAUGGCCGUGGCCAGUGCAUCGUCGCAGUACUGCAGCAGCUGCUGCUGCUGCGCCUUCAGCGCUGCGCUCUGCUGC .(((((.(((....(((((((.....((....((((..(((((((.((((....)))))))))).)..))))....((((((((....))))))))))..))))))).))).)))))... ( -63.30) >DroAna_CAF1 80284 120 - 1 CGAAGGUUUCCGAUUCGCUGACCAAUACUGGCCUGCUCCAUGCGCAUGGCAGUGGCCAGGGCAUCGUCGCAGUGGCGGGAUAGCUCCUGUUGCUGCUCCUGCAGAGCCUUGCUUUGCUGU (((.((((..((...))..))))....((((((.(((((((....)))).)))))))))....)))..((((..((((((....))))))..))))....(((((((...)))))))... ( -52.50) >DroPer_CAF1 91176 120 - 1 CGCAGAUUGUUAAUCCGCUGCCCGAUGCUGGCCUGCUCCAUGCGCAUGGCUGUGGCCAGGGCCUCGUCGCAGUGACGGGACAGCUGUUGCUGCUGCUCCUGCAGGGACUUGCUCUGCUGC .(((((..((...(((.((((..((((..(((((((.....))...((((....))))))))).))))((((..((((.....))))..)))).......)))))))...)))))))... ( -52.30) >consensus CGCAGAUUGCUAAUCCGCUGACCGAUGCUGGCCUGCUCCAUGCGCAUGGCGGUGGCCAGGGCAUCGUCGCAGUGCCGGGACAGCUCCUGCUGCUGCUCCUGCAGAGCCUUGCUCUGCUGC .((((..........(((((..(((((((((((....((((....))))....)))))..))))))...)))))......((((....))))))))....((((((.....))))))... (-32.35 = -33.80 + 1.45)

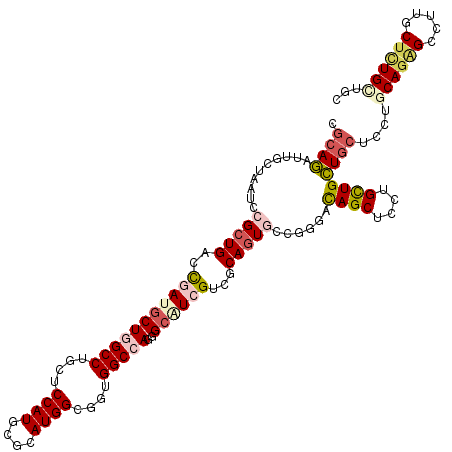
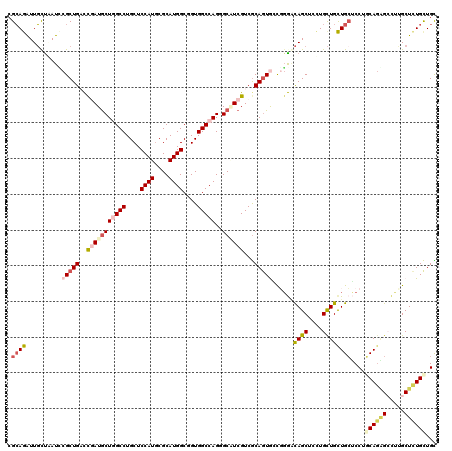
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:13:58 2006