
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 5,155,612 – 5,155,772 |
| Length | 160 |
| Max. P | 0.990572 |

| Location | 5,155,612 – 5,155,732 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.39 |
| Mean single sequence MFE | -52.93 |
| Consensus MFE | -44.01 |
| Energy contribution | -42.93 |
| Covariance contribution | -1.08 |
| Combinations/Pair | 1.27 |
| Mean z-score | -3.17 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.52 |
| SVM RNA-class probability | 0.961001 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5155612 120 + 22407834 GGUGAUCCGCGCCUGAGCGAGAACGUCGAUGAUCUGGUGCGCGUGCUCGACGGCCUCGGCGAAUUGGUCAAGGCCAAGUCGCAGAGCCUCGAGCAGACGCUGGCCCAGAUCGACGUUUAC ((((.....)))).......(((((((((...(((((.((((((((((((.(((.((.((((.(((((....))))).)))).)))))))))))..))))..)))))))))))))))).. ( -60.70) >DroVir_CAF1 76146 120 + 1 GGUGAUCCGCGCCUGAGCGAGAAUGUUGAUGAUUUGGUGCGCGUGCUCGAUGGUCUGGGCGAACUGGUCAAAGCCAAAUCUCAGAGUCUCGAGCAGACGCUAGCCCAAAUCGAUGUCUAU .....(((((......))).)).......((((((((.((((((((((((.(.((((((.....((((....))))...)))))).).))))))..))))..))))))))))........ ( -42.70) >DroGri_CAF1 66475 120 + 1 GGUGAUCCGCGCCUCAGCGAGAAUGUUGAUGAUUUGGUGCGUUUGCUCGAUGGUCUCGGCGAUUUGGUUCGCUCCAAAUCGCAGAGCCUGGAGCAGACGCUGAGCCAAAUCGAUGUAUAC ((((.....))))(((((......)))))(((((((((((((((((((.(.((.(((.(((((((((......))))))))).)))))).))))))))))...)))))))))........ ( -57.90) >DroWil_CAF1 42420 120 + 1 GGUGAUCCACGCCUUAGCGAGAAUGUUGAUGAUUUGGUGCGCGUGCUCGAUGGCUUGGGUGGUGUGGUGAAAACCAAAUCGCAGAGCCUCGAGCAAACGCUGGCCCAAAUCGAUGUCUAU ((((.....))))(((((......)))))((((((((.((((((((((((.(((((..(((((.((((....)))).))))).)))))))))))..))))..))))))))))........ ( -52.60) >DroMoj_CAF1 95688 120 + 1 GGUGAUCCGCGCCUGAGCGAAAAUGUUGACGAUUUGGUGCGCGUGCUCGAUGGCCUCGGUGAACUGGUGAAGACCAAGUCACAGAGUCUCGAGCAGACGCUGGCCCAAAUCGAUGUCUAU ...(((.(((......)))..........((((((((.((((((((((((.(((.((.((((..((((....))))..)))).)))))))))))..))))..))))))))))..)))... ( -47.00) >DroAna_CAF1 40595 120 + 1 GGUGAUCCGCGCCUGAGCGAAAAUGUCGACGAUCUGGUGCGCGUGCUCGAUGGCUUGGGCGAAUUGGUCAAGGCCAAGUCGCAGAGCCUCGAGCAGACGCUAUCCCAGAUUGACGUCUAC ((((.....))))...........(.((.((((((((.(((..(((((((.(((((..((((.(((((....))))).)))).))))))))))))..)))....)))))))).)).)... ( -56.70) >consensus GGUGAUCCGCGCCUGAGCGAGAAUGUUGAUGAUUUGGUGCGCGUGCUCGAUGGCCUCGGCGAAUUGGUCAAGGCCAAAUCGCAGAGCCUCGAGCAGACGCUGGCCCAAAUCGAUGUCUAC ((((.....))))................((((((((.(((..(((((((.(((....((((..((((....))))..))))...))))))))))..)))....))))))))........ (-44.01 = -42.93 + -1.08)



| Location | 5,155,612 – 5,155,732 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.39 |
| Mean single sequence MFE | -48.80 |
| Consensus MFE | -42.51 |
| Energy contribution | -42.68 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.19 |
| Mean z-score | -3.66 |
| Structure conservation index | 0.87 |
| SVM decision value | 2.22 |
| SVM RNA-class probability | 0.990572 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5155612 120 - 22407834 GUAAACGUCGAUCUGGGCCAGCGUCUGCUCGAGGCUCUGCGACUUGGCCUUGACCAAUUCGCCGAGGCCGUCGAGCACGCGCACCAGAUCAUCGACGUUCUCGCUCAGGCGCGGAUCACC ...(((((((((((((....((((.((((((((((((.((((.((((......)))).)))).)).))).))))))).)))).)))))...)))))))).((((......))))...... ( -57.20) >DroVir_CAF1 76146 120 - 1 AUAGACAUCGAUUUGGGCUAGCGUCUGCUCGAGACUCUGAGAUUUGGCUUUGACCAGUUCGCCCAGACCAUCGAGCACGCGCACCAAAUCAUCAACAUUCUCGCUCAGGCGCGGAUCACC .........(((((((....((((.(((((((...((((.((.((((......)))).))...))))...))))))).)))).)))))))......((((.(((....))).)))).... ( -42.80) >DroGri_CAF1 66475 120 - 1 GUAUACAUCGAUUUGGCUCAGCGUCUGCUCCAGGCUCUGCGAUUUGGAGCGAACCAAAUCGCCGAGACCAUCGAGCAAACGCACCAAAUCAUCAACAUUCUCGCUGAGGCGCGGAUCACC .........(((((((....((((.(((((..(((((.(((((((((......))))))))).))).))...))))).)))).)))))))......((((.(((....))).)))).... ( -48.40) >DroWil_CAF1 42420 120 - 1 AUAGACAUCGAUUUGGGCCAGCGUUUGCUCGAGGCUCUGCGAUUUGGUUUUCACCACACCACCCAAGCCAUCGAGCACGCGCACCAAAUCAUCAACAUUCUCGCUAAGGCGUGGAUCACC .........(((((((....((((.(((((((((((........((((....)))).........)))).))))))).)))).)))))))......((((.(((....))).)))).... ( -42.23) >DroMoj_CAF1 95688 120 - 1 AUAGACAUCGAUUUGGGCCAGCGUCUGCUCGAGACUCUGUGACUUGGUCUUCACCAGUUCACCGAGGCCAUCGAGCACGCGCACCAAAUCGUCAACAUUUUCGCUCAGGCGCGGAUCACC ...(((...(((((((....((((.(((((((..(((.((((.(((((....))))).)))).)))....))))))).)))).))))))))))......(((((......)))))..... ( -48.40) >DroAna_CAF1 40595 120 - 1 GUAGACGUCAAUCUGGGAUAGCGUCUGCUCGAGGCUCUGCGACUUGGCCUUGACCAAUUCGCCCAAGCCAUCGAGCACGCGCACCAGAUCGUCGACAUUUUCGCUCAGGCGCGGAUCACC ((.((((...((((((....((((.(((((((((((..((((.((((......)))).))))...)))).))))))).)))).)))))))))).))...(((((......)))))..... ( -53.80) >consensus AUAGACAUCGAUUUGGGCCAGCGUCUGCUCGAGGCUCUGCGACUUGGCCUUGACCAAUUCGCCCAGGCCAUCGAGCACGCGCACCAAAUCAUCAACAUUCUCGCUCAGGCGCGGAUCACC .........(((((((....((((.(((((((((((..((((.((((......)))).))))...)))).))))))).)))).)))))))......((((.(((....))).)))).... (-42.51 = -42.68 + 0.17)


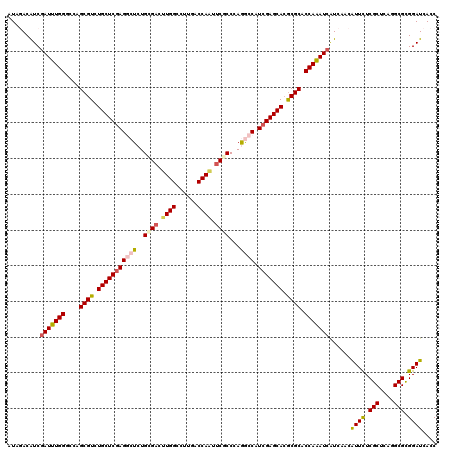
| Location | 5,155,652 – 5,155,772 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.00 |
| Mean single sequence MFE | -45.87 |
| Consensus MFE | -31.80 |
| Energy contribution | -32.83 |
| Covariance contribution | 1.03 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.34 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.627926 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5155652 120 + 22407834 GCGUGCUCGACGGCCUCGGCGAAUUGGUCAAGGCCAAGUCGCAGAGCCUCGAGCAGACGCUGGCCCAGAUCGACGUUUACCAACAGCAGAUGCAGUCCCUGCGCCAGCGUAUCAUCCAGG ...(((((((.(((.((.((((.(((((....))))).)))).)))))))))))).((((((((.(((...((((((....))).((....)).))).))).)))))))).......... ( -55.50) >DroVir_CAF1 76186 120 + 1 GCGUGCUCGAUGGUCUGGGCGAACUGGUCAAAGCCAAAUCUCAGAGUCUCGAGCAGACGCUAGCCCAAAUCGAUGUCUAUCAACAGCAAAUGCAAACGCUGCGCCAGCGUAUUAUACAGG ...(((((((.(.((((((.....((((....))))...)))))).).))))))).(((((.((.......(((....)))..((((..........)))).)).))))).......... ( -35.80) >DroGri_CAF1 66515 120 + 1 GUUUGCUCGAUGGUCUCGGCGAUUUGGUUCGCUCCAAAUCGCAGAGCCUGGAGCAGACGCUGAGCCAAAUCGAUGUAUACCAACAGCAAAUGCAAACGCUCCGUCAGCGUAUUAUACAGG ((((((((.(.((.(((.(((((((((......))))))))).)))))).))))))))...............((((((......((....))..(((((.....)))))..)))))).. ( -45.50) >DroWil_CAF1 42460 120 + 1 GCGUGCUCGAUGGCUUGGGUGGUGUGGUGAAAACCAAAUCGCAGAGCCUCGAGCAAACGCUGGCCCAAAUCGAUGUCUAUCAGCAGCAAAUGCAAACGCUGCGCCAACGGAUUAUACAGG ((((((((((.(((((..(((((.((((....)))).))))).)))))))))))..))))((((.......(((....))).(((((..........))))))))).............. ( -49.50) >DroMoj_CAF1 95728 120 + 1 GCGUGCUCGAUGGCCUCGGUGAACUGGUGAAGACCAAGUCACAGAGUCUCGAGCAGACGCUGGCCCAAAUCGAUGUCUAUCAACAGCAAAUGCAAACGCUCCGCCAGCGUAUUAUACAGG ...(((((((.(((.((.((((..((((....))))..)))).)))))))))))).((((((((.......(((....)))....((....)).........)))))))).......... ( -43.50) >DroPer_CAF1 47015 120 + 1 GCGUGCUCGAUGGCUUGGGUGAAUUGGUGAAGGCCAAGUCCCAGAGCCUCGAACAGACGCUCGCCCAGAUCGAUGUCUACCAGCAGCAGAUGCAAACGCUGCGGCAGCGCAUCAUCCAGG (((((.((((.((((((((....(((((....)))))..)))).)))))))).)..))))...((..(((.(((((((.((.(((((..........))))))).)).))))))))..)) ( -45.40) >consensus GCGUGCUCGAUGGCCUCGGCGAAUUGGUGAAGGCCAAAUCGCAGAGCCUCGAGCAGACGCUGGCCCAAAUCGAUGUCUACCAACAGCAAAUGCAAACGCUGCGCCAGCGUAUUAUACAGG ...(((((((.(((....((((..((((....))))..))))...)))))))))).((((((((.........(((......)))((....)).........)))))))).......... (-31.80 = -32.83 + 1.03)



| Location | 5,155,652 – 5,155,772 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.00 |
| Mean single sequence MFE | -46.84 |
| Consensus MFE | -32.47 |
| Energy contribution | -33.08 |
| Covariance contribution | 0.62 |
| Combinations/Pair | 1.28 |
| Mean z-score | -2.12 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.07 |
| SVM RNA-class probability | 0.570411 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5155652 120 - 22407834 CCUGGAUGAUACGCUGGCGCAGGGACUGCAUCUGCUGUUGGUAAACGUCGAUCUGGGCCAGCGUCUGCUCGAGGCUCUGCGACUUGGCCUUGACCAAUUCGCCGAGGCCGUCGAGCACGC ..........((((((((.(((((((.((....)).(((....))))))..)))).)))))))).((((((((((((.((((.((((......)))).)))).)).))).)))))))... ( -54.40) >DroVir_CAF1 76186 120 - 1 CCUGUAUAAUACGCUGGCGCAGCGUUUGCAUUUGCUGUUGAUAGACAUCGAUUUGGGCUAGCGUCUGCUCGAGACUCUGAGAUUUGGCUUUGACCAGUUCGCCCAGACCAUCGAGCACGC ..........((((((((((((((........))))))((((....))))......)))))))).(((((((...((((.((.((((......)))).))...))))...)))))))... ( -40.50) >DroGri_CAF1 66515 120 - 1 CCUGUAUAAUACGCUGACGGAGCGUUUGCAUUUGCUGUUGGUAUACAUCGAUUUGGCUCAGCGUCUGCUCCAGGCUCUGCGAUUUGGAGCGAACCAAAUCGCCGAGACCAUCGAGCAAAC ............(((((.((((((..(((....(((((((((....))))))..)))...)))..)))))).(((((.(((((((((......))))))))).))).)).)).))).... ( -44.40) >DroWil_CAF1 42460 120 - 1 CCUGUAUAAUCCGUUGGCGCAGCGUUUGCAUUUGCUGCUGAUAGACAUCGAUUUGGGCCAGCGUUUGCUCGAGGCUCUGCGAUUUGGUUUUCACCACACCACCCAAGCCAUCGAGCACGC ...........(((((((((((((........))))))((((....))))......)))))))..(((((((((((........((((....)))).........)))).)))))))... ( -40.93) >DroMoj_CAF1 95728 120 - 1 CCUGUAUAAUACGCUGGCGGAGCGUUUGCAUUUGCUGUUGAUAGACAUCGAUUUGGGCCAGCGUCUGCUCGAGACUCUGUGACUUGGUCUUCACCAGUUCACCGAGGCCAUCGAGCACGC ..........((((((((..((((........))))((((((....))))))....)))))))).(((((((..(((.((((.(((((....))))).)))).)))....)))))))... ( -46.10) >DroPer_CAF1 47015 120 - 1 CCUGGAUGAUGCGCUGCCGCAGCGUUUGCAUCUGCUGCUGGUAGACAUCGAUCUGGGCGAGCGUCUGUUCGAGGCUCUGGGACUUGGCCUUCACCAAUUCACCCAAGCCAUCGAGCACGC ((..(((((((..(((((((((((........)))))).))))).)))).)))..))...((((..((((((((((.((((..((((......))))....)))))))).)))))))))) ( -54.70) >consensus CCUGUAUAAUACGCUGGCGCAGCGUUUGCAUUUGCUGUUGAUAGACAUCGAUUUGGGCCAGCGUCUGCUCGAGGCUCUGCGACUUGGCCUUCACCAAUUCACCCAGGCCAUCGAGCACGC ..........((((((((((((((........))))))((((....))))......)))))))).(((((((((((..(.((.((((......)))).)).)...)))).)))))))... (-32.47 = -33.08 + 0.62)

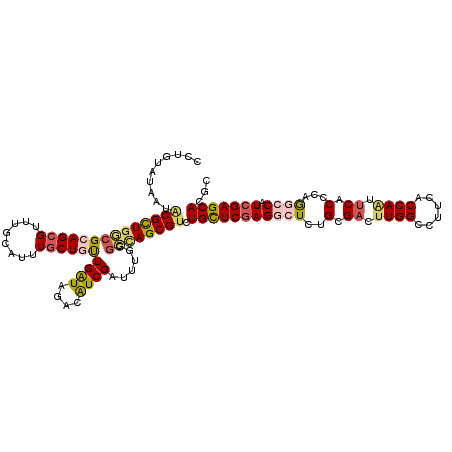

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:13:37 2006