
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 5,100,153 – 5,100,300 |
| Length | 147 |
| Max. P | 0.996533 |

| Location | 5,100,153 – 5,100,267 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 92.28 |
| Mean single sequence MFE | -36.72 |
| Consensus MFE | -29.02 |
| Energy contribution | -29.10 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.94 |
| SVM RNA-class probability | 0.983229 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5100153 114 + 22407834 AAAUUUGUACGCAAAAUGUAUUCUGACACGAAACACCCUGUAACUGUCAGGCUGACAGCGUCAGCUGGCCUAACAGCAAGCUAACGGCGCAGAUGUUAGUUUACAUUUACCGAU .........((..(((((((..(((((((....((.((((.......)))).))...(((((((((.((......)).))))...))))).).))))))..)))))))..)).. ( -33.60) >DroSec_CAF1 552 114 + 1 AAAUUUGUACGCAAAAUGUAUUGUGACACGAAACACCCUGUAGCUGUCAGGCUGACAGCGUCAGCUGGCCUAACAGCGAGCUAACAGCGCAGAUGUUAGUUUACAUUUACCGAU .....(((.(((((......))))))))((............(((((.((((...((((....)))))))).)))))(((((((((.......)))))))))........)).. ( -34.20) >DroSim_CAF1 552 114 + 1 AAAUUUGUACGCAAAAUGUAUUGUGACACGAAACACCCUGUAGCUGUCAGGCUGACAGCGUCAGCUGGCCUAACAGCGAGCUAACAGCGCAGAUGUUAGUUUACAUUUGCCGAU .....(((.(((((......))))))))((.......((((.((((((.....))))))...((((.((......)).)))).)))).((((((((......)))))))))).. ( -36.40) >DroEre_CAF1 551 114 + 1 AAAUUUGUUCGCAAAAUGUAUUGUGACAGGAUUCACCCUGUGGCUGUCAGGCUUACAGCGUCAGCUGGCCUAACAGCGUGUUUACAGAGCAGGUGUUAGUUUACAUUUUCCGAA .......((((.((((((((((((((((((......))))).(((((.((((...((((....)))))))).))))).....)))))(((....)))....)))))))).)))) ( -37.70) >DroYak_CAF1 548 114 + 1 AAAUUUGUUCGCAAAAUGUAUUGUGACAGGAAUCACCCUGUGGCUGUCAGGCUGACAGCGUCAGCUGGCUUAACAGCGAGCUAACGGCGCAGGUGUUAGUUUACAUUUUCCGAU ........(((.((((((((..((((......))))((((((((((((.....))))))((.((((.(((....))).)))).))..))))))........)))))))).))). ( -41.70) >consensus AAAUUUGUACGCAAAAUGUAUUGUGACACGAAACACCCUGUAGCUGUCAGGCUGACAGCGUCAGCUGGCCUAACAGCGAGCUAACAGCGCAGAUGUUAGUUUACAUUUACCGAU .........((..(((((((...(((((.(......)((((.(((((.((((...((((....)))))))).)))))..((.....)))))).)))))...)))))))..)).. (-29.02 = -29.10 + 0.08)

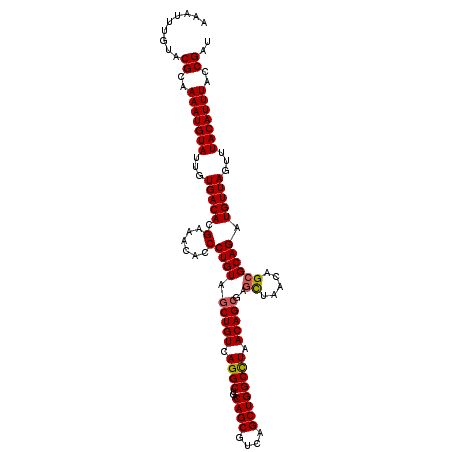
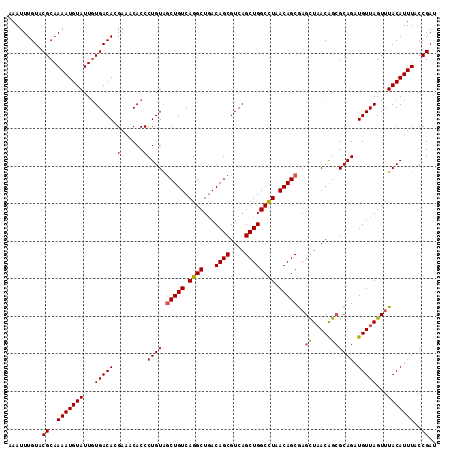
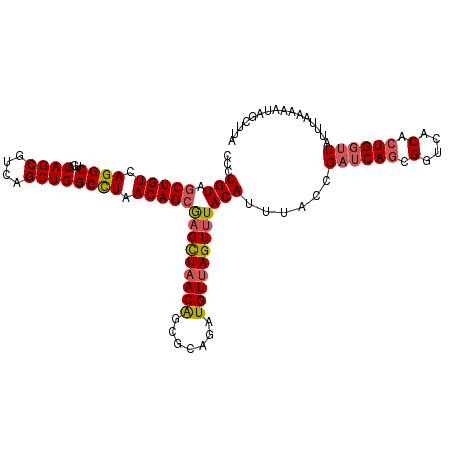
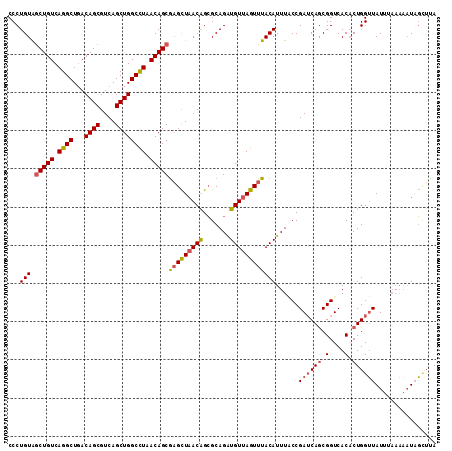
| Location | 5,100,153 – 5,100,267 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 92.28 |
| Mean single sequence MFE | -40.40 |
| Consensus MFE | -30.56 |
| Energy contribution | -30.96 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.94 |
| Structure conservation index | 0.76 |
| SVM decision value | 2.22 |
| SVM RNA-class probability | 0.990614 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5100153 114 - 22407834 AUCGGUAAAUGUAAACUAACAUCUGCGCCGUUAGCUUGCUGUUAGGCCAGCUGACGCUGUCAGCCUGACAGUUACAGGGUGUUUCGUGUCAGAAUACAUUUUGCGUACAAAUUU ....(((((((((..((.((((..((((((((((((.(((....))).)))))))((((((.....)))))).....)))))...)))).))..)))).))))).......... ( -38.80) >DroSec_CAF1 552 114 - 1 AUCGGUAAAUGUAAACUAACAUCUGCGCUGUUAGCUCGCUGUUAGGCCAGCUGACGCUGUCAGCCUGACAGCUACAGGGUGUUUCGUGUCACAAUACAUUUUGCGUACAAAUUU ....(((((((((.....((((..((((((((((((.(((....))).)))))))((((((.....)))))).....)))))...)))).....)))).))))).......... ( -36.50) >DroSim_CAF1 552 114 - 1 AUCGGCAAAUGUAAACUAACAUCUGCGCUGUUAGCUCGCUGUUAGGCCAGCUGACGCUGUCAGCCUGACAGCUACAGGGUGUUUCGUGUCACAAUACAUUUUGCGUACAAAUUU ....(((((((((.....((((..((((((((((((.(((....))).)))))))((((((.....)))))).....)))))...)))).....)))).))))).......... ( -38.50) >DroEre_CAF1 551 114 - 1 UUCGGAAAAUGUAAACUAACACCUGCUCUGUAAACACGCUGUUAGGCCAGCUGACGCUGUAAGCCUGACAGCCACAGGGUGAAUCCUGUCACAAUACAUUUUGCGAACAAAUUU ((((.((((((((........((((....((....))((((((((((((((....))))...))))))))))..))))((((......))))..)))))))).))))....... ( -41.80) >DroYak_CAF1 548 114 - 1 AUCGGAAAAUGUAAACUAACACCUGCGCCGUUAGCUCGCUGUUAAGCCAGCUGACGCUGUCAGCCUGACAGCCACAGGGUGAUUCCUGUCACAAUACAUUUUGCGAACAAAUUU .(((.((((((((........((((.(..(((((((.(((....))).)))))))((((((.....))))))).))))(((((....)))))..)))))))).)))........ ( -46.40) >consensus AUCGGAAAAUGUAAACUAACAUCUGCGCUGUUAGCUCGCUGUUAGGCCAGCUGACGCUGUCAGCCUGACAGCUACAGGGUGUUUCGUGUCACAAUACAUUUUGCGUACAAAUUU ..((..(((((((........((((.((.....))..((((((((((((((....))))...))))))))))..))))(((........)))..)))))))..))......... (-30.56 = -30.96 + 0.40)

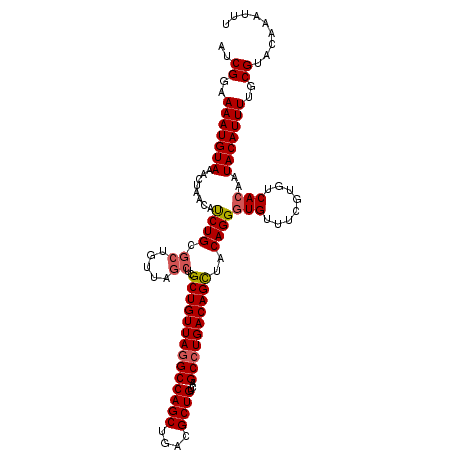
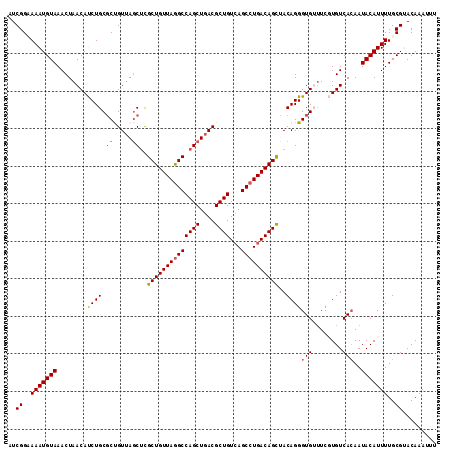
| Location | 5,100,188 – 5,100,300 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 89.38 |
| Mean single sequence MFE | -36.06 |
| Consensus MFE | -28.46 |
| Energy contribution | -28.94 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.22 |
| SVM RNA-class probability | 0.932504 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5100188 112 + 22407834 CCCUGUAACUGUCAGGCUGACAGCGUCAGCUGGCCUAACAGCAAGCUAACGGCGCAGAUGUUAGUUUACAUUUACCGAUCAGCGGUCACACUGGUUAUUUAAAAAUAGCUUA ........((((.((((...((((....)))))))).)))).((((((.(((...((((((......)))))).)))(((((.(....).)))))..........)))))). ( -32.60) >DroSec_CAF1 587 112 + 1 CCCUGUAGCUGUCAGGCUGACAGCGUCAGCUGGCCUAACAGCGAGCUAACAGCGCAGAUGUUAGUUUACAUUUACCGAUCAGCGGUCACACUGGUUAUUUAAAAAUAGCUCA .......(((((.((((...((((....)))))))).)))))((((((.......((((((......))))))...((((((.(....).)))))).........)))))). ( -38.40) >DroSim_CAF1 587 112 + 1 CCCUGUAGCUGUCAGGCUGACAGCGUCAGCUGGCCUAACAGCGAGCUAACAGCGCAGAUGUUAGUUUACAUUUGCCGAUCAGCGGUCACACUGGUUAUUUAAAAAUAGCUUA .......(((((.((((...((((....)))))))).)))))((((((...(.((((((((......)))))))))((((((.(....).)))))).........)))))). ( -42.20) >DroEre_CAF1 586 111 + 1 CCCUGUGGCUGUCAGGCUUACAGCGUCAGCUGGCCUAACAGCGUGUUUACAGAGCAGGUGUUAGUUUACAUUUUCCGAACAGCGGUCACACUGGAUAUUUAAUU-UUUUUGA .((.((((((((.((((...((((....)))))))).)))))((((..((..(((....))).))..))))...(((.....)))...))).))..........-....... ( -30.70) >DroYak_CAF1 583 112 + 1 CCCUGUGGCUGUCAGGCUGACAGCGUCAGCUGGCUUAACAGCGAGCUAACGGCGCAGGUGUUAGUUUACAUUUUCCGAUCAACGGUCACUCUGGUUAUUUCAAAAUCGGGUA .((((((((((((.....))))))((.((((.(((....))).)))).))..))))))................(((.....)))..((.((((((.......)))))))). ( -36.40) >consensus CCCUGUAGCUGUCAGGCUGACAGCGUCAGCUGGCCUAACAGCGAGCUAACAGCGCAGAUGUUAGUUUACAUUUACCGAUCAGCGGUCACACUGGUUAUUUAAAAAUAGCUUA ...(((.(((((.((((...((((....)))))))).)))))(((((((((.......))))))))))))......((((((.(....).))))))................ (-28.46 = -28.94 + 0.48)

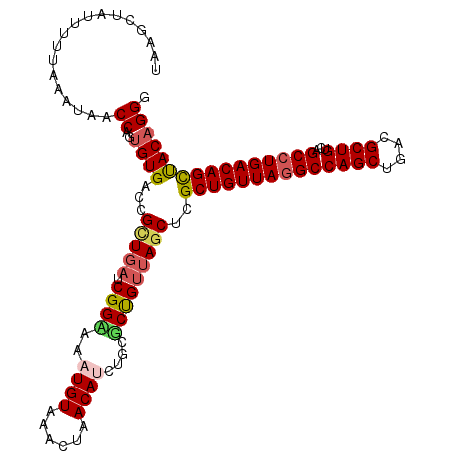
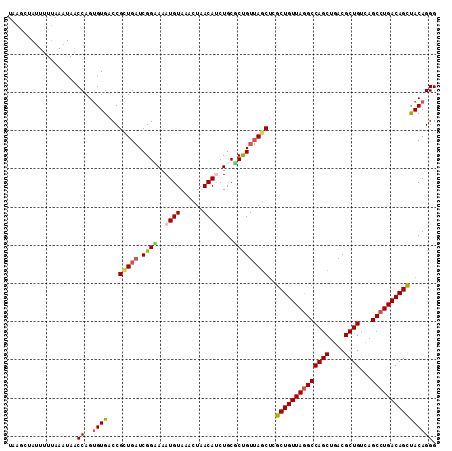
| Location | 5,100,188 – 5,100,300 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 89.38 |
| Mean single sequence MFE | -36.53 |
| Consensus MFE | -31.12 |
| Energy contribution | -31.64 |
| Covariance contribution | 0.52 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.85 |
| SVM decision value | 2.71 |
| SVM RNA-class probability | 0.996533 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 5100188 112 - 22407834 UAAGCUAUUUUUAAAUAACCAGUGUGACCGCUGAUCGGUAAAUGUAAACUAACAUCUGCGCCGUUAGCUUGCUGUUAGGCCAGCUGACGCUGUCAGCCUGACAGUUACAGGG .((((((............(((((....)))))..((((..((((......))))....)))).))))))((((((((((((((....))))...))))))))))....... ( -35.80) >DroSec_CAF1 587 112 - 1 UGAGCUAUUUUUAAAUAACCAGUGUGACCGCUGAUCGGUAAAUGUAAACUAACAUCUGCGCUGUUAGCUCGCUGUUAGGCCAGCUGACGCUGUCAGCCUGACAGCUACAGGG .((((((............(((((....)))))....(((.((((......)))).))).....))))))((((((((((((((....))))...))))))))))....... ( -40.20) >DroSim_CAF1 587 112 - 1 UAAGCUAUUUUUAAAUAACCAGUGUGACCGCUGAUCGGCAAAUGUAAACUAACAUCUGCGCUGUUAGCUCGCUGUUAGGCCAGCUGACGCUGUCAGCCUGACAGCUACAGGG ..................((..((((...((((((.((((.((((......)))).))).).))))))..((((((((((((((....))))...)))))))))))))).)) ( -39.30) >DroEre_CAF1 586 111 - 1 UCAAAAA-AAUUAAAUAUCCAGUGUGACCGCUGUUCGGAAAAUGUAAACUAACACCUGCUCUGUAAACACGCUGUUAGGCCAGCUGACGCUGUAAGCCUGACAGCCACAGGG .......-...........(((.(((.(((.....))).....(....)...))))))((((((......((((((((((((((....))))...)))))))))).)))))) ( -31.30) >DroYak_CAF1 583 112 - 1 UACCCGAUUUUGAAAUAACCAGAGUGACCGUUGAUCGGAAAAUGUAAACUAACACCUGCGCCGUUAGCUCGCUGUUAAGCCAGCUGACGCUGUCAGCCUGACAGCCACAGGG ...((((((........((....)).......))))))................((((.(..(((((((.(((....))).)))))))((((((.....))))))).)))). ( -36.06) >consensus UAAGCUAUUUUUAAAUAACCAGUGUGACCGCUGAUCGGAAAAUGUAAACUAACAUCUGCGCUGUUAGCUCGCUGUUAGGCCAGCUGACGCUGUCAGCCUGACAGCUACAGGG ..................((..((((...(((((.((((..((((......))))....)))))))))..((((((((((((((....))))...)))))))))))))))). (-31.12 = -31.64 + 0.52)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:13:08 2006