
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 4,954,776 – 4,954,976 |
| Length | 200 |
| Max. P | 0.972792 |

| Location | 4,954,776 – 4,954,896 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.11 |
| Mean single sequence MFE | -50.78 |
| Consensus MFE | -44.08 |
| Energy contribution | -43.95 |
| Covariance contribution | -0.13 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.35 |
| SVM RNA-class probability | 0.698916 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
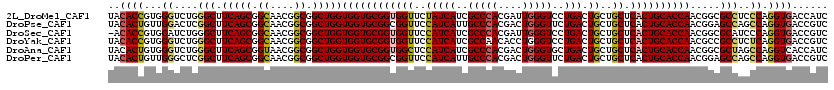
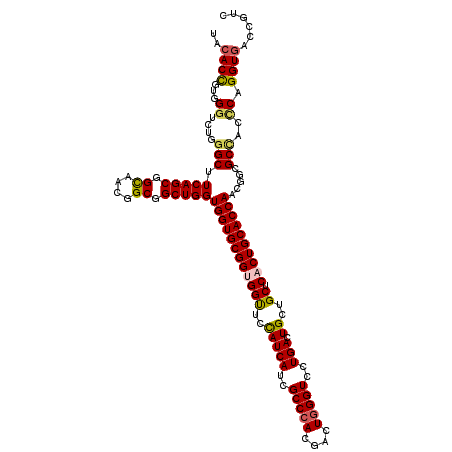
>2L_DroMel_CAF1 4954776 120 + 22407834 CAGUUGUGGUUCUGGAUGAAGUCGCCGCUGCCGACGGUGUGGGUGAACUGGGCGUUGGUGCGCCAGGUGGCUCCGUAGUAGAUGGUCACCUGGGAGGCGCCGUUGGUGCAGUGAGCAGCA ..(((((..(((.....))).((((.((.((((((((((((((((((((.(((((....))))).)))....((((.....)))))))))).....))))))))))))).))))))))). ( -51.20) >DroPse_CAF1 2775 120 + 1 CAGUUGUGGUUCUGGAUGAAGUUACCGCUGCCAAUGGUGUGGGUGAACUGGGCGUUGGUGCGCCAGGUUGCGCCGUAGUAGACGGUCACCUGGCUGGCUCCGUUGGUGCAGUGAGCAGCA ..((((((((.((......))..)))((.((((((((.((.((((((((.(((((....))))).)))).))))........(((((....))))))).)))))))))).....))))). ( -49.40) >DroEre_CAF1 3096 120 + 1 CAGUUGUGGUUCUGGAUGAAGUUGCCGCUACCGAUGGUGUGGGUGAACUGGGCGUUAGUGCGCCAGGUGGCUCCGUAGUAGACGGUCACCUGAGAGGCGCCGUUGGUGCAGUGAGCAGCA (((........)))......(((((((((((((((((((((((((((((.(((((....))))).)))....((((.....)))))))))).....)))))))))))..)))).))))). ( -51.40) >DroYak_CAF1 2385 120 + 1 CAGUUGUGGUUCUGGAUGAAGUCGCCGCUGCCGAUGGUGUGGGUGAACUGGGCGUUGGUGCGCCAGGUGGCUCCGUAGUAGACGGUCACCUGAGAGGCGGCGUUGGUGCAGUGAGCAGCA ..(((((....(((.((.((((((((((.(((((((.(.(((.....))).))))))))))..(((((((..((((.....)))))))))))...)))))).)).)).)))...))))). ( -50.90) >DroAna_CAF1 2845 120 + 1 CAGUUUCCGUUCUGACGGAAUUCGUGGGAUCCCACGGUGUGGGUGAACUGGGCGUUGGUGCGCCAGGUGGCUCCGUAGUAGAUGGUGACCUGGCUAGCGCCGUUGGUGCAGUGAGCAGCA ....((((((....)))))).((((((....))))))(((..((..((((.((..(((((((((((((.(((.(......)..))).)))))))..))))))...)).))))..)).))) ( -52.40) >DroPer_CAF1 2489 120 + 1 CAGUUGUGGUUCUGGAUGAAGUUACCGCUGCCAAUGGUGUGGGUGAACUGGGCGUUGGUGCGCCAGGUUGCGCCGUAGUAGACGGUCACCUGGCUGGCUCCGUUGGUGCAGUGAGCAGCA ..((((((((.((......))..)))((.((((((((.((.((((((((.(((((....))))).)))).))))........(((((....))))))).)))))))))).....))))). ( -49.40) >consensus CAGUUGUGGUUCUGGAUGAAGUCGCCGCUGCCGAUGGUGUGGGUGAACUGGGCGUUGGUGCGCCAGGUGGCUCCGUAGUAGACGGUCACCUGGCAGGCGCCGUUGGUGCAGUGAGCAGCA ..(((((..(((.....))).((((.((.((((((((((((((((((((.(((((....))))).)))....((((.....)))))))))).....))))))))))))).))))))))). (-44.08 = -43.95 + -0.13)

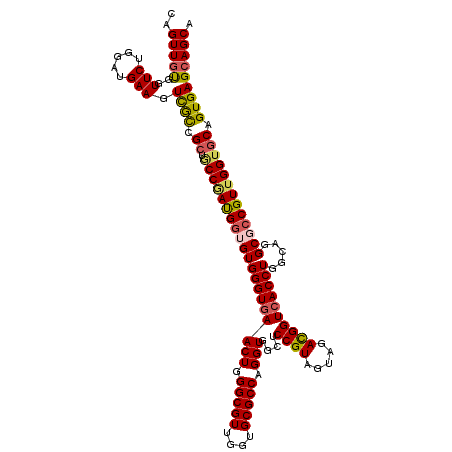
| Location | 4,954,816 – 4,954,936 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.22 |
| Mean single sequence MFE | -49.47 |
| Consensus MFE | -41.53 |
| Energy contribution | -42.25 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.83 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.70 |
| SVM RNA-class probability | 0.972792 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 4954816 120 - 22407834 GGUGCGGUGGUUCUAUCAUCGCCCACGAUUGGGUCCUGACUGCUGCUCACUGCACCAACGGCGCCUCCCAGGUGACCAUCUACUACGGAGCCACCUGGCGCACCAACGCCCAGUUCACCC (((((((((((.(..(((..(((((....)))))..)))..)..)).)))))))))...((.((...(((((((....(((.....)))..))))))).)).))................ ( -45.40) >DroPse_CAF1 2815 120 - 1 GGUGCGGCGGUUCCAUCAUUGCCCACGACUGGGUUCUGACUGCUGCUCACUGCACCAACGGAGCCAGCCAGGUGACCGUCUACUACGGCGCAACCUGGCGCACCAACGCCCAGUUCACCC (((((...((((((.(((..(((((....)))))..))).((.(((.....))).))..)))))).(((((((...((((......))))..))))))))))))................ ( -52.70) >DroEre_CAF1 3136 120 - 1 GGUGCGGUGGUUCCAUCAUCGCCCACGACUGGGUCUUGACUGCUGCUCACUGCACCAACGGCGCCUCUCAGGUGACCGUCUACUACGGAGCCACCUGGCGCACUAACGCCCAGUUCACCC ((((.(((((((((.(((..(((((....)))))..))).((.(((.....))).))((((((((.....)))).)))).......))))))))).((((......)))).....)))). ( -51.00) >DroYak_CAF1 2425 120 - 1 GGUGCGGUGGUUCCAUCAUCGCCAACACCUGGGUCCUGACUGCUGCUCACUGCACCAACGCCGCCUCUCAGGUGACCGUCUACUACGGAGCCACCUGGCGCACCAACGCCCAGUUCACCC ((((.(((((((((...........((((((((..(.(..((.(((.....))).))...).)...))))))))...((.....))))))))))).((((......)))).....)))). ( -43.10) >DroAna_CAF1 2885 120 - 1 GGUGCGGUGGCUCCAUCAUCGCCCACGACUGGGUGCUGACUGCUGCUCACUGCACCAACGGCGCUAGCCAGGUCACCAUCUACUACGGAGCCACCUGGCGCACCAACGCCCAGUUCACCC (((((((((((..(((((.((((((....)))))).))).))..)).)))))))))...((((...(((((((.....(((.....)))...))))))).......)))).......... ( -51.90) >DroPer_CAF1 2529 120 - 1 GGUGCGGCGGUUCCAUCAUUGCCCACGACUGGGUUCUGACUGCUGCUCACUGCACCAACGGAGCCAGCCAGGUGACCGUCUACUACGGCGCAACCUGGCGCACCAACGCCCAGUUCACCC (((((...((((((.(((..(((((....)))))..))).((.(((.....))).))..)))))).(((((((...((((......))))..))))))))))))................ ( -52.70) >consensus GGUGCGGUGGUUCCAUCAUCGCCCACGACUGGGUCCUGACUGCUGCUCACUGCACCAACGGCGCCACCCAGGUGACCGUCUACUACGGAGCCACCUGGCGCACCAACGCCCAGUUCACCC (((((((((((..(((((..(((((....)))))..))).))..)).)))))))))..............(((((((((.....))))......((((.((......)))))).))))). (-41.53 = -42.25 + 0.72)

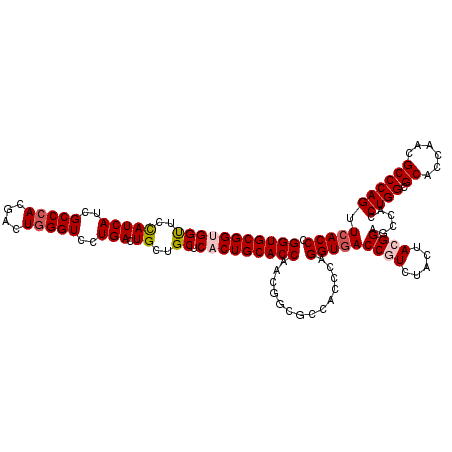

| Location | 4,954,856 – 4,954,976 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.72 |
| Mean single sequence MFE | -54.32 |
| Consensus MFE | -46.02 |
| Energy contribution | -46.13 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.28 |
| SVM RNA-class probability | 0.666769 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 4954856 120 - 22407834 UACACCGUGGGUCUGGGCUUCAGCGGCAACGGCGGCUGGUGGUGCGGUGGUUCUAUCAUCGCCCACGAUUGGGUCCUGACUGCUGCUCACUGCACCAACGGCGCCUCCCAGGUGACCAUC ..((((.((((...((((((((((.((....)).)))))((((((((((((.(..(((..(((((....)))))..)))..)..)).))))))))))..))).)).))))))))...... ( -56.70) >DroPse_CAF1 2855 120 - 1 UACACUGUUGGACUCGGCUUCAGCGGCAACGGCGGCUGGUGGUGCGGCGGUUCCAUCAUUGCCCACGACUGGGUUCUGACUGCUGCUCACUGCACCAACGGAGCCAGCCAGGUGACCGUC ..((((..(((.((.(((((((((.((....)).)))(.((((((((.(((..(((((..(((((....)))))..))).))..)).).)))))))).))))))))))))))))...... ( -51.90) >DroSec_CAF1 1854 119 - 1 -ACACCGUGGAUCUGGGCUUCAGCGGCAACGGCGGCUGGUGGUGCGGUGGUUCCAUCAUCGCCCACGAUUGGGUCCUGACUGCUGCUCACUGCACCAACGGCGCAUCCCAGGUGACCGUC -.((((.(((...((.((((((((.((....)).)))))((((((((((((..(((((..(((((....)))))..))).))..)).))))))))))..))).))..)))))))...... ( -54.10) >DroYak_CAF1 2465 120 - 1 UACACCGUGGGUCUGGGCUUCAGCGGCAACGGCGGCUGGUGGUGCGGUGGUUCCAUCAUCGCCAACACCUGGGUCCUGACUGCUGCUCACUGCACCAACGCCGCCUCUCAGGUGACCGUC ..((((.((((.....((....))(....)((((((...((((((((((((..(((((..(((........)))..))).))..)).))))))))))..)))))).))))))))...... ( -51.70) >DroAna_CAF1 2925 120 - 1 UACACUGUGGGUCUGGGCUUCAGCGGUAACGGCGGCUGGUGGUGCGGUGGCUCCAUCAUCGCCCACGACUGGGUGCUGACUGCUGCUCACUGCACCAACGGCGCUAGCCAGGUCACCAUC ......(((((.((.((((..(((.((....)).((((.((((((((((((..(((((.((((((....)))))).))).))..)).)))))))))).))))))))))).)).).)))). ( -55.90) >DroPer_CAF1 2569 120 - 1 UACACUGUUGGGCUCGGCUUCAGCGGCAACGGCGGCUGGUGGUGCGGCGGUUCCAUCAUUGCCCACGACUGGGUUCUGACUGCUGCUCACUGCACCAACGGAGCCAGCCAGGUGACCGUC ..((((....((((.(((((((((.((....)).)))(.((((((((.(((..(((((..(((((....)))))..))).))..)).).)))))))).))))))))))).))))...... ( -55.60) >consensus UACACCGUGGGUCUGGGCUUCAGCGGCAACGGCGGCUGGUGGUGCGGUGGUUCCAUCAUCGCCCACGACUGGGUCCUGACUGCUGCUCACUGCACCAACGGCGCCACCCAGGUGACCGUC ..((((...((....(((.(((((.((....)).)))))((((((((((((..(((((..(((((....)))))..))).))..)).)))))))))).....)))..)).))))...... (-46.02 = -46.13 + 0.11)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:11:32 2006