
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 4,555,280 – 4,555,440 |
| Length | 160 |
| Max. P | 0.955391 |

| Location | 4,555,280 – 4,555,400 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.50 |
| Mean single sequence MFE | -56.32 |
| Consensus MFE | -49.16 |
| Energy contribution | -48.75 |
| Covariance contribution | -0.41 |
| Combinations/Pair | 1.28 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.45 |
| SVM RNA-class probability | 0.955391 |
| Prediction | RNA |
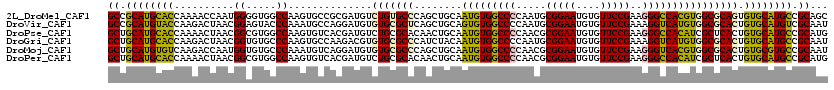
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 4555280 120 - 22407834 AUGUCUGUGCCCAGCUGCAAUGUGGCCCCAAUGCGGAAUGUGUUCCGAAGGGCCACGUGGCGCAGUGUGCAUGCCGCAGCGGCUACGAUGGCCAACCCGCCGACCGGGUGGCCGGCUGUA ........(((.((((((...(((((((.....(((((....)))))..)))))))(((((((.....))..))))).)))))).....(((((.((((.....)))))))))))).... ( -61.00) >DroVir_CAF1 75 120 - 1 AUGUGUGCGCUCAGCUGCAGUGUGGCCCCAAUGCGGAAUGUGUUCCGAAAGGUCAUGUGGCGCACUGUGCAUGUCGCAAUGGCUACGACGGCCAACCCGCUGACCGGCUGGCCGGUUGCA .....((((..((((.((((((((.((((.....)).(((((..((....)).))))))))))))))))).)).)))).(((((.....)))))....((.((((((....)))))))). ( -48.40) >DroPse_CAF1 75 120 - 1 AUGUCUGCGCACAACUGCAAUGUGGCCCCAACGCGGAAUGUGUUCCGAAGGGCCACAUCGCUCACUGUGCAUGCCGCAUGGGCUACGACGGCCAACCCGCUGACCGGGUGGCCGGCUGUA ......(((((((...((.(((((((((.....(((((....)))))..))))))))).))....)))))..(((.....))).....((((((.((((.....)))))))))))).... ( -57.60) >DroGri_CAF1 75 120 - 1 ACGUGUGCGCCCAUCUACAAUGUGGCCCCAAUGCGGAAUGUGUUCCGAAAGGUCAUGUGGCGCACUGUGCAUGCCGCAAUGGCUACGACGGCCAACCCGCUGACCGGGUGGCCGGCUGCA (((.(((((((........((((((((......(((((....)))))...))))))))))))))))))(((.(((((....))......(((((.((((.....))))))))))))))). ( -53.70) >DroAna_CAF1 75 120 - 1 AUGUCUGCGCCCAGCUGCAAUGUGGCCCCAACGCGGAAUGUGUUCCGAAGGGCCACGUGGCGCAGUGUGCAUGCCGCACCGGCUAUGACGGCCAGCCCGCCGACCGGGUGGCCGGUUGUA ..((((((((...(((((.(((((((((.....(((((....)))))..)))))))))...)))))))))).((((...))))...)))(((((.((((.....)))))))))....... ( -59.60) >DroPer_CAF1 75 120 - 1 AUGUCUGCGCACAACUGCAAUGUGGCCCCAACGCGGAAUGUGUUCCGAAGGGCCACAUCGCUCACUGUGCAUGCCGCAUGGGCUACGACGGCCAACCCGCUGACCGGGUGGCCGGCUGUA ......(((((((...((.(((((((((.....(((((....)))))..))))))))).))....)))))..(((.....))).....((((((.((((.....)))))))))))).... ( -57.60) >consensus AUGUCUGCGCCCAGCUGCAAUGUGGCCCCAACGCGGAAUGUGUUCCGAAGGGCCACGUGGCGCACUGUGCAUGCCGCAACGGCUACGACGGCCAACCCGCUGACCGGGUGGCCGGCUGUA ....(((((((........(((((((((.....(((((....)))))..))))))))))))))))..((((.(((((....))......((((.(((((.....)))))))))))))))) (-49.16 = -48.75 + -0.41)

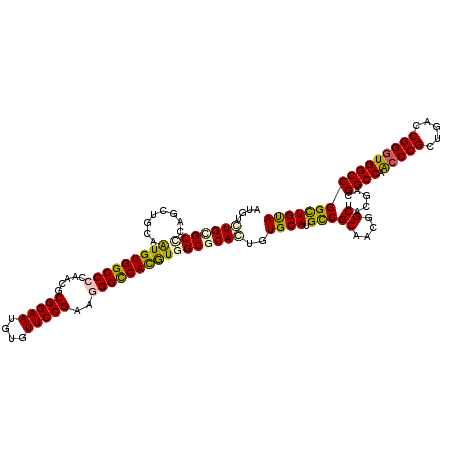

| Location | 4,555,320 – 4,555,440 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.72 |
| Mean single sequence MFE | -50.25 |
| Consensus MFE | -42.66 |
| Energy contribution | -42.05 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.18 |
| SVM RNA-class probability | 0.927208 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 4555320 120 - 22407834 GCCGCAUGCACCAAAACCAAUGGGGUGGCCAAGUGCCGCGAUGUCUGUGCCCAGCUGCAAUGUGGCCCCAAUGCGGAAUGUGUUCCGAAGGGCCACGUGGCGCAGUGUGCAUGCCGCAGC ((.((((((((.........(((((((((.....)))))(....)....))))(((((.(((((((((.....(((((....)))))..)))))))))...))))))))))))).))... ( -59.10) >DroVir_CAF1 115 120 - 1 GCCGCAUGUACCAAGACUAACGGAGUACCCAAAUGCCAGGAUGUGUGCGCUCAGCUGCAGUGUGGCCCCAAUGCGGAAUGUGUUCCGAAAGGUCAUGUGGCGCACUGUGCAUGUCGCAAU ((.((((((((..........((.....))..............(((((((...(((((...((....)).))))).(((((..((....)).)))))))))))).)))))))).))... ( -41.50) >DroPse_CAF1 115 120 - 1 GCUGCAUGCACCAAAACUAACGGCGUGGCCAAGUGUCACGAUGUCUGCGCACAACUGCAAUGUGGCCCCAACGCGGAAUGUGUUCCGAAGGGCCACAUCGCUCACUGUGCAUGCCGCAUG ((.((((((((........(((.((((((.....)))))).)))..(((((....))).(((((((((.....(((((....)))))..))))))))).)).....)))))))).))... ( -51.80) >DroGri_CAF1 115 120 - 1 GCUGCAUGCACCAAGACUAACGGUGUGCCCAAGUGCCAAGACGUGUGCGCCCAUCUACAAUGUGGCCCCAAUGCGGAAUGUGUUCCGAAAGGUCAUGUGGCGCACUGUGCAUGCCGCAAU ((.((((((((........(((.((.((......))))...)))(((((((........((((((((......(((((....)))))...))))))))))))))).)))))))).))... ( -47.50) >DroMoj_CAF1 106 120 - 1 GCUGCAUGUGUCAAGACCAAUGGUGUGCCCAAAUGUCAGGAUGUGUGCGCCCAGCUGCAAUGUGGCCCCAACGCGGAAUGUGUUCCGAAGGGUCACGUGGCGCACUGUGCGUGCCGCAAU ((.(((((((((..(((...(((.....)))...)))..)))..(((((((........(((((((((.....(((((....)))))..))))))))))))))))...)))))).))... ( -49.80) >DroPer_CAF1 115 120 - 1 GCUGCAUGCACCAAAACUAACGGCGUGGCCAAGUGUCACGAUGUCUGCGCACAACUGCAAUGUGGCCCCAACGCGGAAUGUGUUCCGAAGGGCCACAUCGCUCACUGUGCAUGCCGCAUG ((.((((((((........(((.((((((.....)))))).)))..(((((....))).(((((((((.....(((((....)))))..))))))))).)).....)))))))).))... ( -51.80) >consensus GCUGCAUGCACCAAAACUAACGGCGUGCCCAAGUGCCACGAUGUCUGCGCCCAGCUGCAAUGUGGCCCCAACGCGGAAUGUGUUCCGAAGGGCCACGUGGCGCACUGUGCAUGCCGCAAU ((.((((((((..........((.....))..............(((((((........(((((((((.....(((((....)))))..)))))))))))))))).)))))))).))... (-42.66 = -42.05 + -0.61)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:08:13 2006