
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 4,520,306 – 4,520,520 |
| Length | 214 |
| Max. P | 0.965604 |

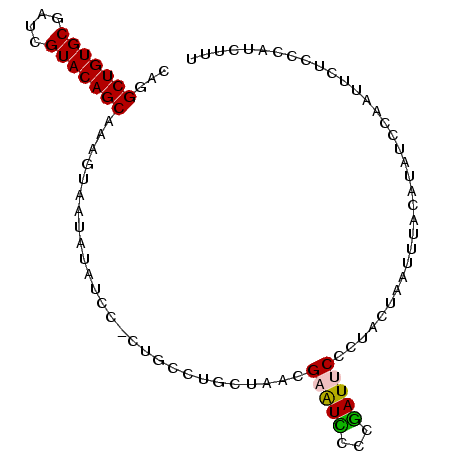
| Location | 4,520,306 – 4,520,400 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 95 |
| Reading direction | reverse |
| Mean pairwise identity | 76.38 |
| Mean single sequence MFE | -13.93 |
| Consensus MFE | -12.18 |
| Energy contribution | -11.85 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.08 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.78 |
| SVM RNA-class probability | 0.848533 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 4520306 94 - 22407834 CAGGCUGUGCGAUCGUACAGCAAAGUAAUAUAUUC-CUGCCUGCUAACGAAUUCGCGAUUCCCUACUAAUUUACAUAUCCAAUUCUCCUAUCUUC .(((...(((((((((..((((..(((........-.))).)))).))))..)))))....)))............................... ( -14.80) >DroPse_CAF1 35963 90 - 1 CCGGCUGUGCUGUUGUACAGCAAAGUA-UAUAUCAAUUGCAUGCCAACGUAUACCUUAUCCUCCUU-CACUCACA---CCACUUUUCCCUUAUUU ...(((((((....)))))))...(((-(((.................))))))............-........---................. ( -12.93) >DroSec_CAF1 35030 94 - 1 CAGGCUGUGCGAUCGUACAGCAAAGUAAUAUAUCC-CUUCCUGCUAACGAGUCUGCGAUUCCCUACUAAUUUACAUAUCCAAUUCUCCCAUCUUC ...(((((((....)))))))..((((....(((.-(...((.(....)))...).)))....))))............................ ( -14.70) >DroEre_CAF1 35542 94 - 1 CAGGCUGUGCGGUCGUACAGCAAAGUAAUAUAUUU-CUGCCUGCUAACGAAUCUACGAUUCCCUACUAAUUUACAUAACCAAUUCUCGAAUUUUU ((((((((((....)))))))...(((........-.)))))).....(((((...))))).................................. ( -13.80) >DroYak_CAF1 25044 94 - 1 CAGGCUGUGCGAUCGUACAGCAAAGUAAUAUAUCC-CUGCCUGCUAAUGAAUCUCAGAUUCUUUACUAAUUUACAUAUCAAAUUCUCCCAUCUCU ...(((((((....)))))))..(((((.......-..(....)....(((((...))))).)))))............................ ( -14.40) >DroPer_CAF1 37611 90 - 1 CCGGCUGUGCUGUUGUACAGCAAAGUA-UAUAUCAAUUGCAUGCCAACGUAUACCUUAUCCUCCUC-CACUCACA---CCACUUUUCCCUUAUUU ...(((((((....)))))))...(((-(((.................))))))............-........---................. ( -12.93) >consensus CAGGCUGUGCGAUCGUACAGCAAAGUAAUAUAUCC_CUGCCUGCUAACGAAUCCCCGAUUCCCUACUAAUUUACAUAUCCAAUUCUCCCAUCUUU ...(((((((....)))))))...........................(((((...))))).................................. (-12.18 = -11.85 + -0.33)

| Location | 4,520,366 – 4,520,480 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.85 |
| Mean single sequence MFE | -39.09 |
| Consensus MFE | -24.78 |
| Energy contribution | -25.32 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.32 |
| Structure conservation index | 0.63 |
| SVM decision value | 1.58 |
| SVM RNA-class probability | 0.965604 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 4520366 114 - 22407834 CGGCCUGCACUCCGUGUGCACGGUCAUCAAGCACCGACCAGCAUGCGAGUGUGUGCCAGGUUACACCGGAGAUCCCUUCUCAGGCUGUGCGAUCGUACAGCAAAGUAAUAUA------UU .(((.(((((((.((((((..((((..........)))).))))))))))))).)))..(((((....((((.....))))..(((((((....)))))))...)))))...------.. ( -46.20) >DroPse_CAF1 36020 113 - 1 UGGCCAGAACUCUCUGUGCCAGGUGAUCAAGCAUCGUCCAACAUGCGAAUGCGUGCCUGGUUACACCGGCGAUCCGUUCUCCGGCUGUGCUGUUGUACAGCAAAGUA-UAUA------UC ((((((((....)))).))))((((((.....)))).))...(((((((((..((((.((.....))))))...)))))....(((((((....)))))))...)))-)...------.. ( -39.20) >DroGri_CAF1 36682 114 - 1 UGGCAUACACUCACUGUGUACUGUUAUCAAGCAUAAUCCGAACUGUCAAUGCGAGCCUGGUUACACCGGAGAUCCAUUCUCUGGCUGUGCUAUUAUACAACAAAGUA-UAAAA-----UC .(((.(((((.....)))))..........((((..............))))..)))((((.((((((((((.....))))))).)))))))((((((......)))-)))..-----.. ( -30.24) >DroMoj_CAF1 43900 119 - 1 CGGCAUACACUCGCAGUGUACUGUUAUUAAGCAUAAUCCGAACUGUCAAUGCGAGCCUGGUUACACCGGAGAUCCUUUCUCCGGCUGUGCCAUUAUACACCAAAGUA-UACAAAUUUAUC .((((((..((((((.((.((.(((((((....))))...))).)))).))))))..........(((((((.....))))))).))))))..(((((......)))-)).......... ( -34.20) >DroAna_CAF1 35828 114 - 1 UGGCCUGCACUCCGUGUGCACAGCCAUCAAGCACCGACCGACGUGCGAGUGUGUGCCUGGUUACACCGGAGAUCCGUUCUCCGGCUGUGCGAUUGUGCAGCAAAGUAAUGAG------CC .(((((((((......(((((((((((((.((((((..((.....))..)).)))).))))......(((((.....))))))))))))))...))))))((......)).)------)) ( -46.20) >DroPer_CAF1 37668 113 - 1 UGGCCAGAACUCUCUGUGCCAGGUGAUCAAGCAUCGUCCAACAUGCGAAUGCGUUCCUGGUUACACCGGCGAUCCGUUCUCCGGCUGUGCUGUUGUACAGCAAAGUA-UAUA------UC ((((((((....)))).)))).(((((((.(((((((.......))).)))).....)))))))..(((.((.....)).)))(((((((....)))))))......-....------.. ( -38.50) >consensus UGGCCUGCACUCCCUGUGCACGGUCAUCAAGCAUCGUCCAACAUGCGAAUGCGUGCCUGGUUACACCGGAGAUCCGUUCUCCGGCUGUGCUAUUGUACAGCAAAGUA_UAUA______UC .(((..((((.....))))...........(((((((.......))).))))..)))....(((..((((((.....))))))(((((((....)))))))...)))............. (-24.78 = -25.32 + 0.53)

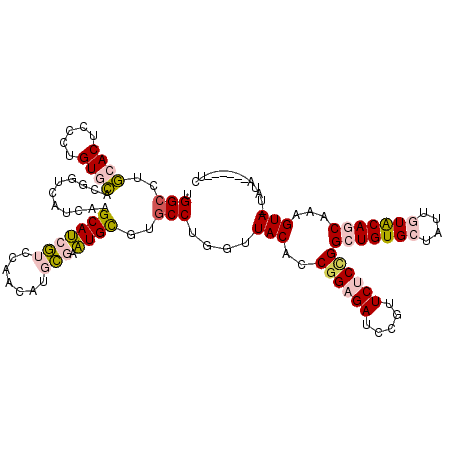

| Location | 4,520,400 – 4,520,520 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.78 |
| Mean single sequence MFE | -46.12 |
| Consensus MFE | -33.00 |
| Energy contribution | -32.20 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.48 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.72 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.920731 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 4520400 120 + 22407834 AGAAGGGAUCUCCGGUGUAACCUGGCACACACUCGCAUGCUGGUCGGUGCUUGAUGACCGUGCACACGGAGUGCAGGCCGCAAGCGCCGAUACAUGGGUUCGAGCAGCGCUGGUUCACAC .....((....)).(((.((((.(((.(...((((((((...(((((((((((.((.((.(((((.....))))))).))))))))))))).))))....))))..).)))))))))).. ( -48.90) >DroPse_CAF1 36053 120 + 1 AGAACGGAUCGCCGGUGUAACCAGGCACGCAUUCGCAUGUUGGACGAUGCUUGAUCACCUGGCACAGAGAGUUCUGGCCACAAGCUCCAACACACGGAUCUGCGCAGCGCUGGUUGACAC ....(((....)))((((((((((((.((((((((..(((((((....(((((......((((.(((......)))))))))))))))))))..))))..))))..)).)))))).)))) ( -48.00) >DroGri_CAF1 36716 120 + 1 AGAAUGGAUCUCCGGUGUAACCAGGCUCGCAUUGACAGUUCGGAUUAUGCUUGAUAACAGUACACAGUGAGUGUAUGCCACAUGCACCGGCACAUGGAUUAGCGCAGCGCUGGUUUACAC ....(((....)))((((((((((((((((.....((((((((....(((.((......((((((.....))))))....)).))))))).)).)).....))).))).)))).)))))) ( -36.10) >DroMoj_CAF1 43939 120 + 1 AGAAAGGAUCUCCGGUGUAACCAGGCUCGCAUUGACAGUUCGGAUUAUGCUUAAUAACAGUACACUGCGAGUGUAUGCCGCAUGCACCGAUACAUGGGUUUGCACAGCGCUGGUUAACAC .....((....)).(((((((((((((((((.((((.(((...((((....))))))).)).)).)))))(((((.(((.((((........))))))).))))).)).)))))).)))) ( -38.60) >DroAna_CAF1 35862 120 + 1 AGAACGGAUCUCCGGUGUAACCAGGCACACACUCGCACGUCGGUCGGUGCUUGAUGGCUGUGCACACGGAGUGCAGGCCACAGGCACCGAUGCACGGGUUGGUGCAGCGCUGGUUCACGC ....(((....)))(((.((((((((...((((.((.(((..(((((((((((.(((((.(((((.....)))))))))))))))))))))..))).)).))))..)).))))))))).. ( -62.30) >DroPer_CAF1 37701 120 + 1 AGAACGGAUCGCCGGUGUAACCAGGAACGCAUUCGCAUGUUGGACGAUGCUUGAUCACCUGGCACAGAGAGUUCUGGCCACAAGCUCCAACACACGGAUCUGCGCAGCGUUGGUUGACAC ....(((....)))(((((((((((..((((((((..(((((((....(((((......((((.(((......)))))))))))))))))))..))))..))))...).)))))).)))) ( -42.80) >consensus AGAACGGAUCUCCGGUGUAACCAGGCACGCAUUCGCAUGUCGGACGAUGCUUGAUAACCGUGCACAGAGAGUGCAGGCCACAAGCACCGACACACGGAUUUGCGCAGCGCUGGUUCACAC ....(((....)))((((((((((((.((((((((..(((((((....(((((....((.(((((.....)))))))...))))))))))))..)))))..)))..)).)))))).)))) (-33.00 = -32.20 + -0.80)

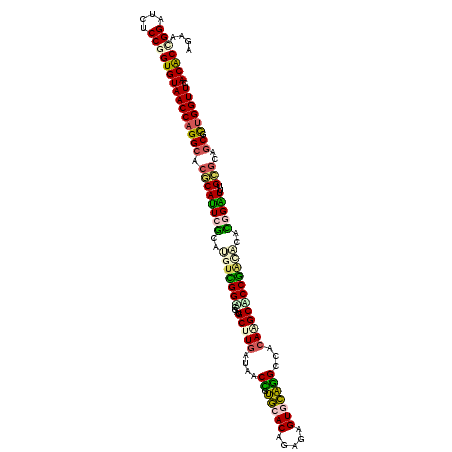
| Location | 4,520,400 – 4,520,520 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.78 |
| Mean single sequence MFE | -47.35 |
| Consensus MFE | -28.48 |
| Energy contribution | -30.10 |
| Covariance contribution | 1.62 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.60 |
| SVM decision value | 1.43 |
| SVM RNA-class probability | 0.953045 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 4520400 120 - 22407834 GUGUGAACCAGCGCUGCUCGAACCCAUGUAUCGGCGCUUGCGGCCUGCACUCCGUGUGCACGGUCAUCAAGCACCGACCAGCAUGCGAGUGUGUGCCAGGUUACACCGGAGAUCCCUUCU ((((((.((.((((((((((....(((((.((((.(((((.(((((((((.....))))).))))..))))).))))...))))))))))).))))..)))))))).((((....)))). ( -52.90) >DroPse_CAF1 36053 120 - 1 GUGUCAACCAGCGCUGCGCAGAUCCGUGUGUUGGAGCUUGUGGCCAGAACUCUCUGUGCCAGGUGAUCAAGCAUCGUCCAACAUGCGAAUGCGUGCCUGGUUACACCGGCGAUCCGUUCU ((((.((((((.((.(((((....((((((((((((((((((((((((....)))).))))......)))))....)))))))))))..)))))))))))))))))(((....))).... ( -53.50) >DroGri_CAF1 36716 120 - 1 GUGUAAACCAGCGCUGCGCUAAUCCAUGUGCCGGUGCAUGUGGCAUACACUCACUGUGUACUGUUAUCAAGCAUAAUCCGAACUGUCAAUGCGAGCCUGGUUACACCGGAGAUCCAUUCU ((((((.((((.(((.(((.....((.((..((((((.((((((((((((.....))))).))))).)).)))....))).)))).....)))))))))))))))).((....))..... ( -40.10) >DroMoj_CAF1 43939 120 - 1 GUGUUAACCAGCGCUGUGCAAACCCAUGUAUCGGUGCAUGCGGCAUACACUCGCAGUGUACUGUUAUUAAGCAUAAUCCGAACUGUCAAUGCGAGCCUGGUUACACCGGAGAUCCUUUCU ((((.((((((.(((.((((....((.((.((((...(((((((((((((.....))))).)))).....))))...))))))))....))))))))))))))))).((....))..... ( -37.30) >DroAna_CAF1 35862 120 - 1 GCGUGAACCAGCGCUGCACCAACCCGUGCAUCGGUGCCUGUGGCCUGCACUCCGUGUGCACAGCCAUCAAGCACCGACCGACGUGCGAGUGUGUGCCUGGUUACACCGGAGAUCCGUUCU ..(((((((((.((.((((..((.((..(.(((((((.((((((.(((((.....)))))..)))).)).))))))).....)..)).)))))))))))))).)))(((....))).... ( -53.40) >DroPer_CAF1 37701 120 - 1 GUGUCAACCAACGCUGCGCAGAUCCGUGUGUUGGAGCUUGUGGCCAGAACUCUCUGUGCCAGGUGAUCAAGCAUCGUCCAACAUGCGAAUGCGUUCCUGGUUACACCGGCGAUCCGUUCU ((((.(((((.....(((((....((((((((((((((((((((((((....)))).))))......)))))....)))))))))))..)))))...)))))))))(((....))).... ( -46.90) >consensus GUGUCAACCAGCGCUGCGCAAACCCAUGUAUCGGUGCUUGUGGCCUGCACUCCCUGUGCACGGUCAUCAAGCAUCGUCCAACAUGCGAAUGCGUGCCUGGUUACACCGGAGAUCCGUUCU ((((.((((((.((.(((((....(((((.((((.((.((((((((((((.....))))).))))).)).)).....)))).)))))..))))))))))))))))).((....))..... (-28.48 = -30.10 + 1.62)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:07:59 2006