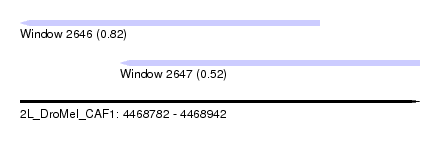
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 4,468,782 – 4,468,942 |
| Length | 160 |
| Max. P | 0.820418 |

| Location | 4,468,782 – 4,468,902 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 86.39 |
| Mean single sequence MFE | -34.32 |
| Consensus MFE | -29.16 |
| Energy contribution | -28.35 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.820418 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
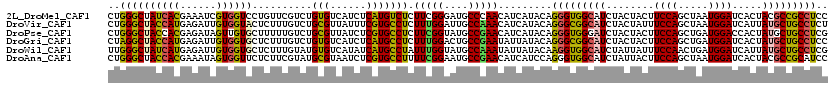
>2L_DroMel_CAF1 4468782 120 - 22407834 UCUCAUGUCUCUUCGGGAUGCCCAACAUCAUACAGGGUGGCAUCUACUACUUCCAGCUAAUGGAUCACUACGCCGCCUCCGUGACGAUCAUGUUUCUGGCCUUCUGUCAAAUGAUUGCCA ...((((((((..(((((((.....)))).....(((((((..........((((.....)))).......)))))))))).)).)).)))).....(((..((........))..))). ( -32.43) >DroVir_CAF1 4500 120 - 1 UUUCGUGCCUCUUUGGAUUGCCAAACAUCAUACAGGGCGGCAUCUACUAUUUCCAGCUAAUGGAUCAUUAUGCUGCCUCUGUGACAAUUAUGUUUCUGGCUUUCUGCCAGAUGAUUGCCA ...........(((((....))))).....(((((((((((((........((((.....)))).....)))))))).))))).((((((....((((((.....))))))))))))... ( -38.02) >DroGri_CAF1 4173 120 - 1 UCUCAUGCCUCUUUGGACUGCCGAAUAUUAUACAGGGCGGCAUCUACUACUUCCAGCUGAUGGAUCACUAUGCUGCCUCCGUGACGAUUAUGUUUCUGGCCUUCUGUCAGAUGAUUGCCA ..(((((....(((((....))))).........(((((((((........((((.....)))).....))))))))).)))))((((((....((((((.....))))))))))))... ( -31.62) >DroWil_CAF1 3539 120 - 1 UAUCAUGCCUAUUUGGUAUGCCAAAUAUUAUACAAGGUGGCAUCUAUUAUUUCCAACUGAUGGAUCAUUAUGCUGCCUCGGUGACAAUAAUGUUCUUGGCCUUUUGCCAAAUGAUUGCCA ......((.(((((((((.((((((((((((...(((..((((........((((.....)))).....))))..))).(....).)))))))..)))))....)))))))))...)).. ( -34.52) >DroMoj_CAF1 4239 120 - 1 UCGCGUGCCUCUUCGGAUUGCCGAACAUUAUACAGGGCGGCAUCUACUACUUCCAGCUAAUGGAUCACUAUGCUGCCUCCGUGACGAUUAUGUUUCUGGCCUUCUGCCAGAUGAUUGCCA (((((......(((((....))))).........(((((((((........((((.....)))).....))))))))).)))))((((((....((((((.....))))))))))))... ( -37.82) >DroAna_CAF1 7479 120 - 1 UCUCGUGCCUUUUCGGAAUGCCGAACAUCAUCCAGGGUGGCAUCUAUUACUUCCAGCUAAUGGAUCACUACGCCGCAUCCGUGACGAUCAUGUUUCUGGCCUUCUGCCAGAUGAUAGCUA ......((...(((((....))))).((((((((((((((.((((((((........))))))))..))))((((.(..((((.....))))..).))))..))))...)))))).)).. ( -31.50) >consensus UCUCAUGCCUCUUCGGAAUGCCGAACAUCAUACAGGGCGGCAUCUACUACUUCCAGCUAAUGGAUCACUAUGCUGCCUCCGUGACGAUUAUGUUUCUGGCCUUCUGCCAGAUGAUUGCCA ...........(((((....)))))..((((...(((((((((........((((.....)))).....)))))))))..))))(((((((....(((((.....))))))))))))... (-29.16 = -28.35 + -0.80)

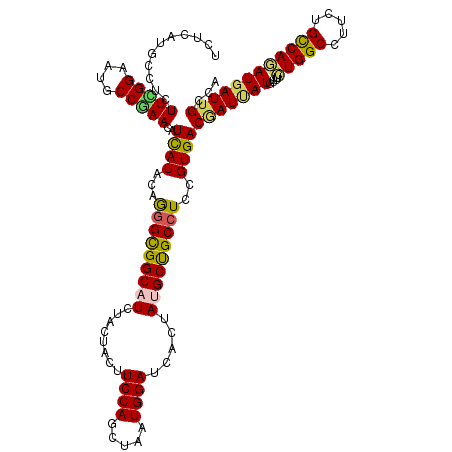
| Location | 4,468,822 – 4,468,942 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.67 |
| Mean single sequence MFE | -34.47 |
| Consensus MFE | -27.40 |
| Energy contribution | -26.49 |
| Covariance contribution | -0.91 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.45 |
| Structure conservation index | 0.79 |
| SVM decision value | -0.03 |
| SVM RNA-class probability | 0.517128 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 4468822 120 - 22407834 CUGGGCUAUCACGAAAUCGUGGUCCUGUUCGUCUGUGUCAUCUCAUGUCUCUUCGGGAUGCCCAACAUCAUACAGGGUGGCAUCUACUACUUCCAGCUAAUGGAUCACUACGCCGCCUCC .(((((...((((....))))((((((.......(((......))).......)))))))))))..........(((((((..........((((.....)))).......))))))).. ( -36.27) >DroVir_CAF1 4540 120 - 1 CUGGGCUACCAUGAGAUUGUGGUACUCUUUGUCUGCGUUAUUUCGUGCCUCUUUGGAUUGCCAAACAUCAUACAGGGCGGCAUCUACUAUUUCCAGCUAAUGGAUCAUUAUGCUGCCUCU ((((((((((((......))))))..........(((......))))))..(((((....))))).......)))((((((((........((((.....)))).....))))))))... ( -33.62) >DroPse_CAF1 28167 120 - 1 CUGGGCUACCACGAGAUAGUUGUGCUUUUUGUCUGCGUUAUCUCGUGCCUCUUCGGUAUGCCGAACAUCAUACAGGGUGGGAUCUACUACUUCCAGCUGAUGGACCACUAUGCUGCCUCG ..((((...((((((((((...(((.........)))))))))))))....(((((....)))))...((((..((((((......)))).((((.....))))))..))))..)))).. ( -34.20) >DroGri_CAF1 4213 120 - 1 CUAGGCUACCAUGAGAUUGUGGUGCUCUUUGUCUGUGUCAUCUCAUGCCUCUUUGGACUGCCGAAUAUUAUACAGGGCGGCAUCUACUACUUCCAGCUGAUGGAUCACUAUGCUGCCUCC ...(((...(((((((...(((..(.........)..))))))))))((.....))...)))............(((((((((........((((.....)))).....))))))))).. ( -34.72) >DroWil_CAF1 3579 120 - 1 UUGGGCUAUCAUGAGAUUGUGGUGCUCUUUGUAUGUGUCAUAUCAUGCCUAUUUGGUAUGCCAAAUAUUAUACAAGGUGGCAUCUAUUAUUUCCAACUGAUGGAUCAUUAUGCUGCCUCG ..((((...(((((((....(((((((((((((((........((((((.....))))))........))))))))).)))))).......((((.....)))))).)))))..)))).. ( -35.89) >DroAna_CAF1 7519 120 - 1 CUGGGCUACCACGAAAUAGUGGUUCUCUUCGUAUGCGUAAUCUCGUGCCUUUUCGGAAUGCCGAACAUCAUCCAGGGUGGCAUCUAUUACUUCCAGCUAAUGGAUCACUACGCCGCAUCC (((((..(((((......))))).......(((((.(....).)))))...(((((....))))).....))))).(((((((((((((........))))))))......))))).... ( -32.10) >consensus CUGGGCUACCACGAGAUUGUGGUGCUCUUUGUCUGCGUCAUCUCAUGCCUCUUCGGAAUGCCGAACAUCAUACAGGGUGGCAUCUACUACUUCCAGCUAAUGGAUCACUAUGCUGCCUCC ..((((((((((......))))))..........(((......))))))).(((((....))))).........(((((((((........((((.....)))).....))))))))).. (-27.40 = -26.49 + -0.91)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:07:37 2006