
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
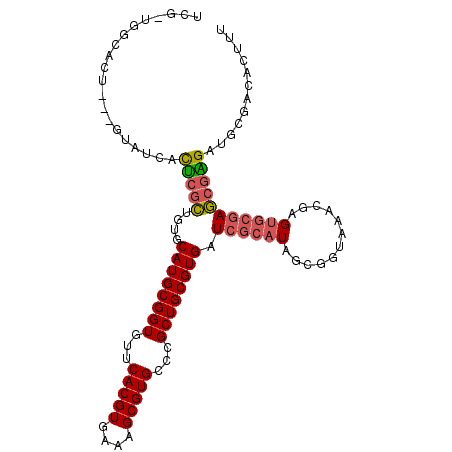
| Location | 479,153 – 479,259 |
| Length | 106 |
| Max. P | 0.996281 |

| Location | 479,153 – 479,259 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 75.57 |
| Mean single sequence MFE | -45.58 |
| Consensus MFE | -27.15 |
| Energy contribution | -29.02 |
| Covariance contribution | 1.86 |
| Combinations/Pair | 1.17 |
| Mean z-score | -3.05 |
| Structure conservation index | 0.60 |
| SVM decision value | 2.68 |
| SVM RNA-class probability | 0.996281 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
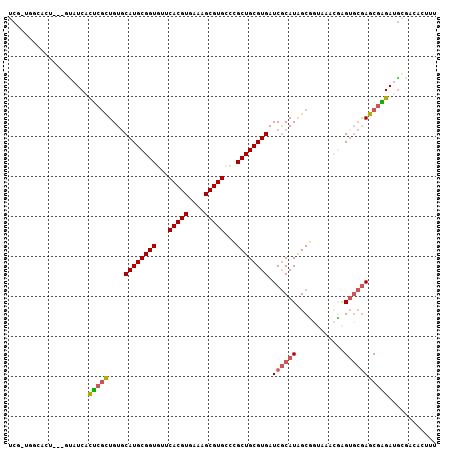
>2L_DroMel_CAF1 479153 106 + 22407834 UCGCUCGCACUCACACAUCACUCGCUGUGCAUGCGGUGUUCACGUGAAAGCGUGUUGGCUGCGUGAUCGCAUGGCUGUGUGCGAGUGCGAGCGGGACGAGGCACU-U ((((((((((((.((((.((...((((((((((((((...(((((....)))))...)))))))....))))))))))))).))))))))))))...........-. ( -54.90) >DroVir_CAF1 33347 103 + 1 GUU-UGGGUUU---GUAUCACUCGUUGUGCAUGCGGUGUUCACGUGAAAGCGUGCCCGCUGCGUGAUCGCAUAGCGCUACACUAGAGCGAACGAGAUGCGAGACUUU ...-.((((((---(((((..(((((((((((((((((..(((((....)))))..)))))))))..)))....((((.......)))))))))))))).)))))). ( -41.20) >DroGri_CAF1 27202 103 + 1 GUG-UGUGUCU---GUAUCACUCGCUGUGCAUGCGGUGUUCACGUGAAAGCGUGCCCGCUGCGUGAUCGCAUAGCGUUCAAUGGGUGCAAGCGAGAUACAAGACUUU ...-...((((---(((((..(((((.(((((((((((..(((((....)))))..)))))).(((.(((...))).)))....))))))))))))))).))))... ( -45.30) >DroSim_CAF1 24978 106 + 1 UCGCUCGCACUCACACAUCACUCGCUGUGCAUGCGGUGUUCACGUGAAAGCGUGCUGGCUGCGUGAUCGCAUGGCUGUGUGCGAGUGCGAGCGGGACGGGGCACU-U ((((((((((((.((((.((...((((((((((((((...(((((....)))))...)))))))....))))))))))))).))))))))))))...........-. ( -54.20) >DroMoj_CAF1 26717 100 + 1 AUA-UG---CU---GUAUCACUCGCUGUGCAUGCGGUGUUCACGUGAAAGCGUGCCCGCUGCGUGAUCGCAUAGCGUUACUGCGGUGCGAGCGAGAUGCGAGACUUU ...-..---.(---(((((..((((....(((((((((..(((((....)))))..))))))))).((((((.(((....))).))))))))))))))))....... ( -44.60) >DroAna_CAF1 31125 107 + 1 UCGCUUCCACUCGCCAAUCAUCCACUGUCCAUGCGGUGUUCACGUGAACGCGUGCUGGCUGCGUGAUCACAUAGCUGACAAUGAGUCAGAGCAGGAUGGAACACUUU ..(.(((((..(((((......((((((....))))))..(((((....))))).))))(((((....))....(((((.....))))).))))..))))))..... ( -33.30) >consensus UCG_UGGCACU___GUAUCACUCGCUGUGCAUGCGGUGUUCACGUGAAAGCGUGCCCGCUGCGUGAUCGCAUAGCGGUAAACGAGUGCGAGCGAGAUGCGACACUUU ....................(((((....((((((((...(((((....)))))...)))))))).((((((............)))))))))))............ (-27.15 = -29.02 + 1.86)

| Location | 479,153 – 479,259 |
|---|---|
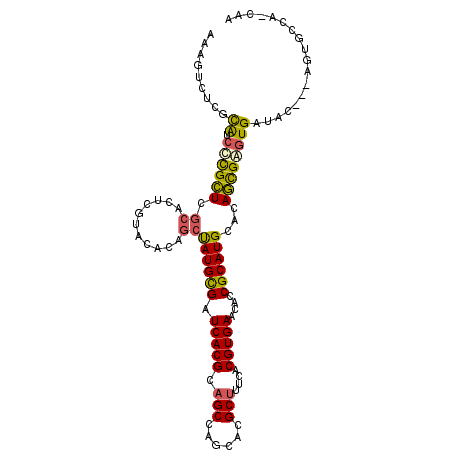
| Length | 106 |
| Sequences | 6 |
| Columns | 107 |
| Reading direction | reverse |
| Mean pairwise identity | 75.57 |
| Mean single sequence MFE | -38.95 |
| Consensus MFE | -17.36 |
| Energy contribution | -16.92 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.80 |
| Structure conservation index | 0.45 |
| SVM decision value | 2.10 |
| SVM RNA-class probability | 0.987974 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
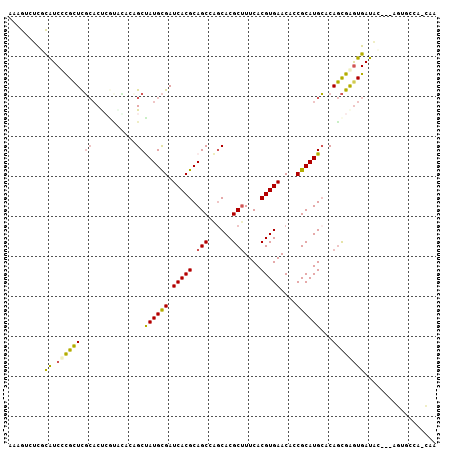
>2L_DroMel_CAF1 479153 106 - 22407834 A-AGUGCCUCGUCCCGCUCGCACUCGCACACAGCCAUGCGAUCACGCAGCCAACACGCUUUCACGUGAACACCGCAUGCACAGCGAGUGAUGUGUGAGUGCGAGCGA .-............((((((((((((((((....((((((.(((((.(((......)))....)))))....))))))(((.....))).)))))))))))))))). ( -47.70) >DroVir_CAF1 33347 103 - 1 AAAGUCUCGCAUCUCGUUCGCUCUAGUGUAGCGCUAUGCGAUCACGCAGCGGGCACGCUUUCACGUGAACACCGCAUGCACAACGAGUGAUAC---AAACCCA-AAC ...(((((((((..(((((((....))).))))..))))))....(((((((.((((......))))....)))).))).........)))..---.......-... ( -31.20) >DroGri_CAF1 27202 103 - 1 AAAGUCUUGUAUCUCGCUUGCACCCAUUGAACGCUAUGCGAUCACGCAGCGGGCACGCUUUCACGUGAACACCGCAUGCACAGCGAGUGAUAC---AGACACA-CAC ...((((.(((((..((((((......(((.(((...))).))).(((((((.((((......))))....)))).)))...)))))))))))---))))...-... ( -38.50) >DroSim_CAF1 24978 106 - 1 A-AGUGCCCCGUCCCGCUCGCACUCGCACACAGCCAUGCGAUCACGCAGCCAGCACGCUUUCACGUGAACACCGCAUGCACAGCGAGUGAUGUGUGAGUGCGAGCGA .-............((((((((((((((((....((((((.(((((.(((......)))....)))))....))))))(((.....))).)))))))))))))))). ( -47.70) >DroMoj_CAF1 26717 100 - 1 AAAGUCUCGCAUCUCGCUCGCACCGCAGUAACGCUAUGCGAUCACGCAGCGGGCACGCUUUCACGUGAACACCGCAUGCACAGCGAGUGAUAC---AG---CA-UAU ........((...((((((((..((((.........)))).....(((((((.((((......))))....)))).)))...))))))))...---.)---).-... ( -36.10) >DroAna_CAF1 31125 107 - 1 AAAGUGUUCCAUCCUGCUCUGACUCAUUGUCAGCUAUGUGAUCACGCAGCCAGCACGCGUUCACGUGAACACCGCAUGGACAGUGGAUGAUUGGCGAGUGGAAGCGA ...((.(((((..((((.(((((.....)))))....((....)))))(((((((((((((((.(((.....))).))))).)))..)).))))))..))))))).. ( -32.50) >consensus AAAGUCUCGCAUCCCGCUCGCACUCGUACACAGCUAUGCGAUCACGCAGCCAGCACGCUUUCACGUGAACACCGCAUGCACAGCGAGUGAUAC___AGUGCCA_CAA .........((.((((((.((...........))((((((.(((((.(((......)))....)))))....))))))...)))))))).................. (-17.36 = -16.92 + -0.44)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:27:45 2006