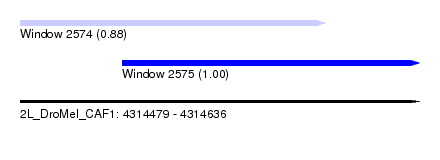
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 4,314,479 – 4,314,636 |
| Length | 157 |
| Max. P | 0.995558 |

| Location | 4,314,479 – 4,314,599 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.83 |
| Mean single sequence MFE | -35.79 |
| Consensus MFE | -33.01 |
| Energy contribution | -32.82 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.28 |
| Structure conservation index | 0.92 |
| SVM decision value | 0.93 |
| SVM RNA-class probability | 0.884316 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 4314479 120 + 22407834 GUGCCGGCACGUGUCAAGCACAAGGCGAUUACCGCCGGCGUCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGA ..((((((..(((.....)))..((......)))))))).(((((.(((....)))........(((((((((...(((((.((((....)))))))))..))))))))).....))))) ( -39.60) >DroVir_CAF1 44644 120 + 1 GUGCCGGCGCGUGUCAAACAUAAAGCUAUAACAGCCGGCGUCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGA ..((((((..((..................)).)))))).(((((.(((....)))........(((((((((...(((((.((((....)))))))))..))))))))).....))))) ( -34.27) >DroGri_CAF1 63636 120 + 1 GUGCCGGCCCGUGUCAAACAUAAAGCAAUAACAGCCGGCGUCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGA ..((((((...(((..........)))......)))))).(((((.(((....)))........(((((((((...(((((.((((....)))))))))..))))))))).....))))) ( -33.20) >DroWil_CAF1 79349 120 + 1 GUGCCGGCACGUGUCAAGCAUAAAGCUAUCACCGCCGGUGUCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGA ..((((((..(((...(((.....)))..))).)))))).(((((.(((....)))........(((((((((...(((((.((((....)))))))))..))))))))).....))))) ( -37.40) >DroMoj_CAF1 49621 120 + 1 GUGCCGGCGCGUGUCAAACAUAAAGCUAUAACAGCCGGCGUCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGA ..((((((..((..................)).)))))).(((((.(((....)))........(((((((((...(((((.((((....)))))))))..))))))))).....))))) ( -34.27) >DroAna_CAF1 46044 120 + 1 GUGCCGGCACGAGUCAAGCACAAGGCGAUAACCGCCGGCGUCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGA (((((..(((((.....((....((((.....)))).)).))).))..)))))..(((((....(((((((((...(((((.((((....)))))))))..)))))))))....))))). ( -36.00) >consensus GUGCCGGCACGUGUCAAACAUAAAGCUAUAACAGCCGGCGUCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGA ..((((((..(((.....)))...(......).)))))).(((((.(((....)))........(((((((((...(((((.((((....)))))))))..))))))))).....))))) (-33.01 = -32.82 + -0.19)

| Location | 4,314,519 – 4,314,636 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.09 |
| Mean single sequence MFE | -34.72 |
| Consensus MFE | -30.13 |
| Energy contribution | -30.13 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.87 |
| Structure conservation index | 0.87 |
| SVM decision value | 2.59 |
| SVM RNA-class probability | 0.995558 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 4314519 117 + 22407834 UCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGACGAAAACAGGAAAAAGGUAACACUGAGGC--UAAAGCUA- ..(((...(((.((((((((((..(((((((((...(((((.((((....)))))))))..))))))))).((....))..))))))))))...((......))...))--)..)))..- ( -33.40) >DroVir_CAF1 44684 116 + 1 UCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGACGAAAACAGGAAAAAGGUAACACAGCC---GAAAUGCUA- ........((((((((((((((..(((((((((...(((((.((((....)))))))))..))))))))).((....))..))))))))))....(((......)))---....)))).- ( -34.90) >DroGri_CAF1 63676 119 + 1 UCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGACGAAAACAGGAAAAAGGUAACACAGACUUUCAAAAGCUG- ...(((..(((.((((((((((..(((((((((...(((((.((((....)))))))))..))))))))).((....))..))))))))))....(......).).))..)))......- ( -33.00) >DroWil_CAF1 79389 119 + 1 UCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGACGAAAACAGGAAAAAGGUAACACUAAGGC-GAAAAGCUAC (((((..((...((((((((((..(((((((((...(((((.((((....)))))))))..))))))))).((....))..))))))))))....(....).))..)))-))........ ( -33.50) >DroMoj_CAF1 49661 116 + 1 UCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGACGAAAACAGGAAAAAGGUAACACAGGC---CAAAUGCUG- ........((((((((((((((..(((((((((...(((((.((((....)))))))))..))))))))).((....))..))))))))))....(((.......))---)...)))).- ( -34.50) >DroPer_CAF1 88953 118 + 1 UCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGACGAAAACAGGAAAAAGGUAACACUGAGGC-CCAGAGCUA- ..(((..((((.((((((((((..(((((((((...(((((.((((....)))))))))..))))))))).((....))..))))))))))...((......))...))-))..)))..- ( -39.00) >consensus UCGUUGGGGGCAUCCUGUUUUUUAUUGUUGCGAUCAUUUUAUCGGUUUGUGCCGUAAAAAUUUGCAAUAAACGUAAACGACGAAAACAGGAAAAAGGUAACACAGACGC_CAAAAGCUA_ ........(((.((((((((((..(((((((((...(((((.((((....)))))))))..))))))))).((....))..))))))))))....(....)..............))).. (-30.13 = -30.13 + 0.00)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:06:27 2006