
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 3,867,626 – 3,867,778 |
| Length | 152 |
| Max. P | 0.996292 |

| Location | 3,867,626 – 3,867,741 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.56 |
| Mean single sequence MFE | -30.14 |
| Consensus MFE | -23.24 |
| Energy contribution | -23.28 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.98 |
| Structure conservation index | 0.77 |
| SVM decision value | 2.39 |
| SVM RNA-class probability | 0.993267 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
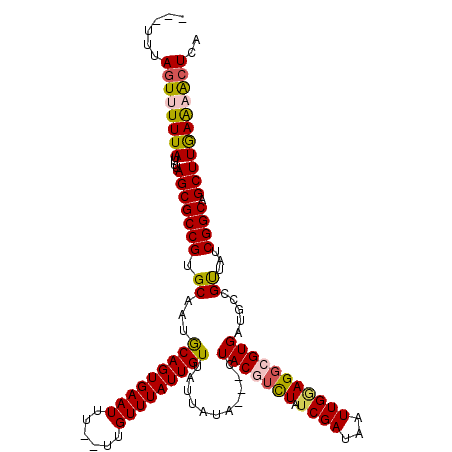
>2L_DroMel_CAF1 3867626 115 + 22407834 AAGUCUAAGGAAGUGCAAUCGAUAACUAGCGGCGCAGUCUUGAGUUUUCAAGCUGCCGAUAACGACAUCACGCCUCCAAUUAUCGAUAGACGUAG---UAUAAUAACAAUAAACAA--AA ..(((((.(......)..(((((((...(.(((((((.((((......)))))))).(((......)))..))).)...))))))))))))....---..................--.. ( -26.60) >DroSec_CAF1 4054 115 + 1 AAGCAUAACGAAGUGCAAUCGAUAAUUAGCGGCGCAGUCUUGAGUUUUCAAGCUGCCGAUAGCUGAAUCACGCCUCCAAUUAUCGAUAGACGUAG---UAUAAUAACAAUAAACAA--AA ..((((......)))).((((((((((.(.(((((((.((((......)))))))).(((......)))..))).).))))))))))........---..................--.. ( -31.70) >DroSim_CAF1 4009 115 + 1 AAGCAUAACGAAGUGCAAUCGAUAAUUAGCGGCGCAGUCUUGAGUUUUUAAGCUGCCGAUAACGGCAUCACGCCUUCAAUUAUCGAUAGACGUAG---UAUAAUAACAAUAAACAA--AA ..((((......)))).((((((((((...((((.((.(((((....)))))))((((....))))....))))...))))))))))........---..................--.. ( -28.70) >DroEre_CAF1 4035 118 + 1 AGACCCAGCGAAGUGCAAUCGAUAAAUGGUGGCGCAGUCUUGAGCGCUCAAGCUGCCGAUAACGGCAUCACGCCUACAAUUAUCGAUAGCUGUAGCUGUAUAAUAACAAUAAACAA--AA .....((((.....((.((((((((.((..((((.((.(((((....)))))))((((....))))....))))..)).)))))))).))....))))..................--.. ( -38.20) >DroYak_CAF1 4089 109 + 1 --------CACAGUGUAAUCGAUAAAUAUUGGCGCAGUCUUGAGUAUUCAAGCUGCCGAUAACGGCUUCACGCCUCCAAUUAUCGAUAAAUGUAG---UAUAAUAACAAUAAACAACAAA --------.(((...(.((((((((.....((((.((.((((......))))))((((....))))....)))).....)))))))).).)))..---...................... ( -25.50) >consensus AAGCAUAACGAAGUGCAAUCGAUAAAUAGCGGCGCAGUCUUGAGUUUUCAAGCUGCCGAUAACGGCAUCACGCCUCCAAUUAUCGAUAGACGUAG___UAUAAUAACAAUAAACAA__AA .................((((((((.....(((((((.(((((....))))))))).(((......)))..))).....))))))))................................. (-23.24 = -23.28 + 0.04)



| Location | 3,867,626 – 3,867,741 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.56 |
| Mean single sequence MFE | -34.32 |
| Consensus MFE | -26.94 |
| Energy contribution | -27.70 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.07 |
| Mean z-score | -3.11 |
| Structure conservation index | 0.78 |
| SVM decision value | 2.68 |
| SVM RNA-class probability | 0.996292 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 3867626 115 - 22407834 UU--UUGUUUAUUGUUAUUAUA---CUACGUCUAUCGAUAAUUGGAGGCGUGAUGUCGUUAUCGGCAGCUUGAAAACUCAAGACUGCGCCGCUAGUUAUCGAUUGCACUUCCUUAGACUU ..--..(((((..((......)---)...((..((((((((((((.(((.(((((....)))))(((((((((....))))).))))))).)))))))))))).)).......))))).. ( -36.90) >DroSec_CAF1 4054 115 - 1 UU--UUGUUUAUUGUUAUUAUA---CUACGUCUAUCGAUAAUUGGAGGCGUGAUUCAGCUAUCGGCAGCUUGAAAACUCAAGACUGCGCCGCUAAUUAUCGAUUGCACUUCGUUAUGCUU ..--..................---....((..((((((((((((.(((.((((......))))(((((((((....))))).))))))).)))))))))))).)).............. ( -34.70) >DroSim_CAF1 4009 115 - 1 UU--UUGUUUAUUGUUAUUAUA---CUACGUCUAUCGAUAAUUGAAGGCGUGAUGCCGUUAUCGGCAGCUUAAAAACUCAAGACUGCGCCGCUAAUUAUCGAUUGCACUUCGUUAUGCUU ..--..................---....((..(((((((((((..(((((..(((((....))))).(((........)))...)))))..))))))))))).)).............. ( -29.50) >DroEre_CAF1 4035 118 - 1 UU--UUGUUUAUUGUUAUUAUACAGCUACAGCUAUCGAUAAUUGUAGGCGUGAUGCCGUUAUCGGCAGCUUGAGCGCUCAAGACUGCGCCACCAUUUAUCGAUUGCACUUCGCUGGGUCU ..--..................((((....((.((((((((.((..(((((..(((((....))))).(((((....)))))...)))))..)).)))))))).)).....))))..... ( -40.10) >DroYak_CAF1 4089 109 - 1 UUUGUUGUUUAUUGUUAUUAUA---CUACAUUUAUCGAUAAUUGGAGGCGUGAAGCCGUUAUCGGCAGCUUGAAUACUCAAGACUGCGCCAAUAUUUAUCGAUUACACUGUG-------- ......................---.((((...((((((((.((..(((((...((((....))))(((((((....))))).)))))))..)).)))))))).....))))-------- ( -30.40) >consensus UU__UUGUUUAUUGUUAUUAUA___CUACGUCUAUCGAUAAUUGGAGGCGUGAUGCCGUUAUCGGCAGCUUGAAAACUCAAGACUGCGCCGCUAAUUAUCGAUUGCACUUCGUUAGGCUU .................................(((((((((((..(((.(((((....)))))(((((((((....))))).)))))))..)))))))))))................. (-26.94 = -27.70 + 0.76)



| Location | 3,867,666 – 3,867,778 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.10 |
| Mean single sequence MFE | -29.88 |
| Consensus MFE | -24.14 |
| Energy contribution | -24.46 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.26 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.81 |
| SVM RNA-class probability | 0.978452 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 3867666 112 - 22407834 ---CUUAAUUUUUAUUUUAGCGCCGUGCAAUACAGUGAAUUU--UUGUUUAUUGUUAUUAUA---CUACGUCUAUCGAUAAUUGGAGGCGUGAUGUCGUUAUCGGCAGCUUGAAAACUCA ---...........(((.(((((((.((((((((((((((..--..)))))))).))))(((---.(((((((.((((...))))))))))).))).))...)))).))).)))...... ( -26.00) >DroSec_CAF1 4094 112 - 1 ---UUUAGUUUUUAUUUUAGCGCCGUGCAAUGCAGUGAAUUU--UUGUUUAUUGUUAUUAUA---CUACGUCUAUCGAUAAUUGGAGGCGUGAUUCAGCUAUCGGCAGCUUGAAAACUCA ---...((((((((....(((((((.((...(((((((((..--..))))))))).......---.(((((((.((((...))))))))))).....))...)))).))))))))))).. ( -30.90) >DroSim_CAF1 4049 112 - 1 ---UUUAGUUUUUAUUUUAGCGCCGAGCAAUGCAGUGAAUUU--UUGUUUAUUGUUAUUAUA---CUACGUCUAUCGAUAAUUGAAGGCGUGAUGCCGUUAUCGGCAGCUUAAAAACUCA ---...((((((((....(((((((((((..(((((((((..--..))))))))).......---.(((((((.((((...))))))))))).))).....))))).))))))))))).. ( -31.30) >DroEre_CAF1 4075 118 - 1 UUGGUUAGUUUUUAUUUUAGCGCCGUGCAAUGCAGUGAAUUU--UUGUUUAUUGUUAUUAUACAGCUACAGCUAUCGAUAAUUGUAGGCGUGAUGCCGUUAUCGGCAGCUUGAGCGCUCA ...(((((........)))))((.(((.((((((((((((..--..)))))))).)))).))).(((..((((..(((((((....(((.....))))))))))..))))..)))))... ( -33.20) >DroYak_CAF1 4121 114 - 1 ---UUUAGUUUUUAUUUUAGCGCCGCGCAAUGCAGUGAAUUUUGUUGUUUAUUGUUAUUAUA---CUACAUUUAUCGAUAAUUGGAGGCGUGAAGCCGUUAUCGGCAGCUUGAAUACUCA ---.........(((((.(((..(((((...(((((((((......))))))))).......---((.((.(((....))).)).)))))))..((((....)))).))).))))).... ( -28.00) >consensus ___UUUAGUUUUUAUUUUAGCGCCGUGCAAUGCAGUGAAUUU__UUGUUUAUUGUUAUUAUA___CUACGUCUAUCGAUAAUUGGAGGCGUGAUGCCGUUAUCGGCAGCUUGAAAACUCA ......((((((((....(((((((.((...(((((((((......)))))))))...........(((((((.((((...))))))))))).....))...)))).))))))))))).. (-24.14 = -24.46 + 0.32)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:03:13 2006