| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 3,750,830 – 3,750,972 |
| Length | 142 |
| Max. P | 0.623295 |

| Location | 3,750,830 – 3,750,947 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.56 |
| Mean single sequence MFE | -44.53 |
| Consensus MFE | -30.04 |
| Energy contribution | -30.27 |
| Covariance contribution | 0.23 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.608345 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
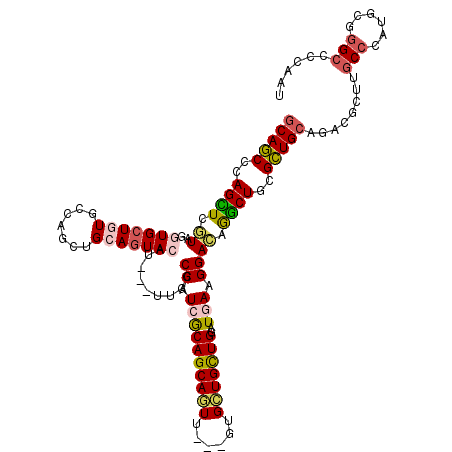
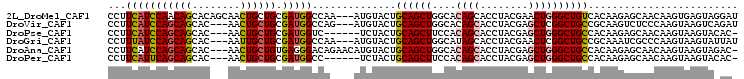
>2L_DroMel_CAF1 3750830 117 - 22407834 ACAGCCCAGUUCGUAGGUGCUGUGCCAGCUGCAGUACAU---UUGGCCAUCGCAGCAGUUGCUGUGCUGUUGGAUGAAGGACAAACUCCGCUGCAGCCGCUUGCCCAUCUUGGCACCAAU (((((((........)).)))))((..((((((((.(((---(..((...((((((....))))))..))..))))..(((.....))))))))))).)).((((......))))..... ( -48.20) >DroPse_CAF1 238561 111 - 1 GCAGCCCAGCUCGUAGGUGCUGUGGAAGCUGCAGUAGA------GACCAUCGCAGCAGUU---GUGCUGCUGGAUGAAGGACAGGCUGCGCUGCAGACGCUUGCCCAUGCGGGCCCCAAU (((((.((((..((...(((((..(...)..)))))..------...((((.((((((..---...))))))))))....))..)))).)))))....((((((....))))))...... ( -48.00) >DroGri_CAF1 259474 114 - 1 GCAGCCGAGUUCGUAGGUGCUAUGCCAGCUGCAGUACAU---UUGGCCAUCGCAGCAAUU---GUGCUGCUGGAUAAAGGAAAGGCUGCGCUGCAGUCGCUUGCCCAUUUUGGCGCCAAU ((.((((((...(((((((...(((.(((.(((((...(---(((.(((..((((((...---.))))))))).))))......)))))))))))..)))))))....)))))))).... ( -44.80) >DroWil_CAF1 350653 114 - 1 GCAACCGAGUUCAUAGGUGCUAUGCCAACUGCAAUACAU---UUGGCCAUCACAGCAAUU---GUGUUGUUGUAUAAAGGAUAAGCUGCGUUGCAAUCUCUUGCCCAUGCGUGCUCCGAU (((.((.........)))))...(((((.((.....)).---))))).(((((((((((.---..)))))))).....(((...(((((((.((((....))))..))))).)))))))) ( -28.70) >DroAna_CAF1 241768 117 - 1 GCAGCCCAGCUCGUAGGUGCUGUGCCAGCUGCAGUACAUGUUCUGUCCCUCACAGCAGUU---GUGCUGCUGGAUGAAGGACAGGCUGCGCUGCAAACGCUUCCCCAUCCUGGCGCCAAU (((((.((((......(((((((.......))))))).....((((((.(((((((((..---...))))))..))).)))))))))).)))))...((((..........))))..... ( -50.40) >DroPer_CAF1 237862 111 - 1 GCAGCCCAGCUCGUAGGUGCUGUGGAAGCUGCAGUAGA------GGCCAUCGCAGCAGUU---GUGCUGCUGAAUGAAGGACAGGCUGCGCUGCAGACGCUUGCCCAUGCGGGCCCCAAU ...((((.((..(((((((((((...(((.(((((...------..((.((.((((((..---...))))))...)).))....)))))))))))).)))))))....))))))...... ( -47.10) >consensus GCAGCCCAGCUCGUAGGUGCUGUGCCAGCUGCAGUACAU___UUGGCCAUCGCAGCAGUU___GUGCUGCUGGAUGAAGGACAGGCUGCGCUGCAGACGCUUGCCCAUGCGGGCCCCAAU (((((..((((.((..(((((((.......))))))).........((.((((((((((......)))))))..))).)))).))))..)))))........(((......)))...... (-30.04 = -30.27 + 0.23)


| Location | 3,750,870 – 3,750,972 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 83.23 |
| Mean single sequence MFE | -29.54 |
| Consensus MFE | -21.50 |
| Energy contribution | -21.95 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.623295 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
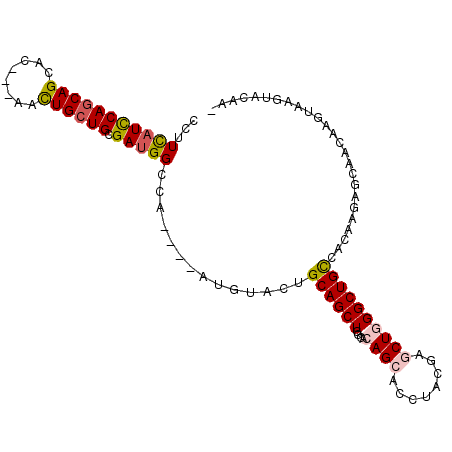
>2L_DroMel_CAF1 3750870 102 + 22407834 CCUUCAUCCAACAGCACAGCAACUGCUGCGAUGGCCAA---AUGUACUGCAGCUGGCACAGCACCUACGAACUGGGCUGUCACAAGAGCAACAAGUGAGUAGGAU ....((((...(((((.......))))).))))((((.---.((.....))..))))......(((((..(((..(((........)))....)))..))))).. ( -30.00) >DroVir_CAF1 285676 99 + 1 CCUUCAUCCAGCAGCAC---AACUGCUGCGAUGGCCAG---AUGUACUGCAGCUGGCACAGCACCUACGAGCUCGGCUGCCGCAAGUCUCCCAAGUAAGUCAGAU ...(((((((((((...---..)))))).)))))...(---((.(((((((((((((.............)).)))))))(....).......)))).))).... ( -33.02) >DroPse_CAF1 238601 95 + 1 CCUUCAUCCAGCAGCAC---AACUGCUGCGAUGGUC------UCUACUGCAGCUUCCACAGCACCUACGAGCUGGGCUGCCACAAGAGCAACAAGUAAGUACAC- ....((((((((((...---..)))))).))))(.(------(((...((((((....((((........))))))))))....)))))...............- ( -30.80) >DroGri_CAF1 259514 99 + 1 CCUUUAUCCAGCAGCAC---AAUUGCUGCGAUGGCCAA---AUGUACUGCAGCUGGCAUAGCACCUACGAACUCGGCUGCCGCAAAUCGCCCAAGUAAGUAUUAU ..........((((((.---...))))))...(((...---.(((...((((((((....(......)....)))))))).)))....))).............. ( -25.10) >DroAna_CAF1 241808 101 + 1 CCUUCAUCCAGCAGCAC---AACUGCUGUGAGGGACAGAACAUGUACUGCAGCUGGCACAGCACCUACGAGCUGGGCUGCCACAAGAGCAACAAGUAAGUAGAC- ((((((..((((((...---..))))))))))))(((.....))).((((.(.(((((((((........))))...))))))....((.....))..))))..- ( -30.40) >DroPer_CAF1 237902 95 + 1 CCUUCAUUCAGCAGCAC---AACUGCUGCGAUGGCC------UCUACUGCAGCUUCCACAGCACCUACGAGCUGGGCUGCCACAAGAGCAACAAGUAAGUACAC- ....((((((((((...---..)))))).))))((.------(((...((((((....((((........))))))))))....)))))...............- ( -27.90) >consensus CCUUCAUCCAGCAGCAC___AACUGCUGCGAUGGCCA____AUGUACUGCAGCUGGCACAGCACCUACGAGCUGGGCUGCCACAAGAGCAACAAGUAAGUACAA_ ...(((((((((((........)))))).)))))..............((((((....((((........))))))))))......................... (-21.50 = -21.95 + 0.45)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:01:55 2006