
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 3,546,676 – 3,546,774 |
| Length | 98 |
| Max. P | 0.739901 |

| Location | 3,546,676 – 3,546,774 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 77.45 |
| Mean single sequence MFE | -16.32 |
| Consensus MFE | -10.55 |
| Energy contribution | -11.78 |
| Covariance contribution | 1.22 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.45 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.739901 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
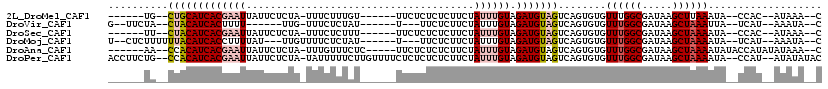
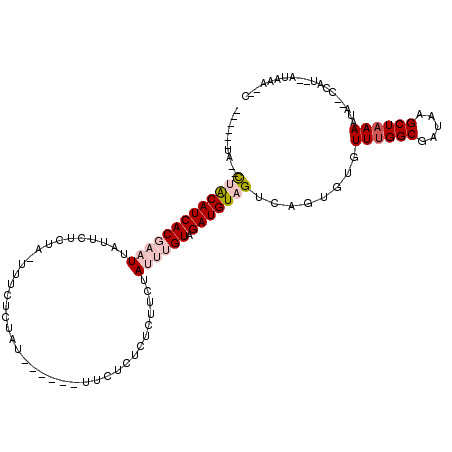
>2L_DroMel_CAF1 3546676 98 + 22407834 ------UG--CUGCAUCACGAAUUAUUCUCUA-UUUCUUUGU------UUCUCUCUCUUCUAUUUGUAGAUGUAGUCAGUGUGUUUGGCGAUAAGCUUAAAUA--CCAC--AUAAA--C ------.(--(((((((((((((.........-.........------.............)))))).))))))))..(((((((((((.....)).))))))--.)))--.....--. ( -19.88) >DroVir_CAF1 17907 93 + 1 G--UUCUA--CUACAUCACUUUU------UUG-UUUCUCUAU------U---UUCUCUUCUAUUUGUAGAUGUAGUCAGUGUGUUUGGCGAUAAGCUAAAUUA--UCAU--AAAUA--C .--....(--(((((((((....------...-.........------.---.............)).))))))))..(((.(((((((.....)))))))..--.)))--.....--. ( -16.31) >DroSec_CAF1 15603 98 + 1 ------UU--CUACAUCACGAAUUAUUCUCUA-UUUCUCUUU------UUCUCUCUCUUCUAUUUGUAGAUGUAGUCAGUGUGUUUGGCGAUAAGCUAAAAUA--CCAC--AUAAA--C ------..--(((((((((((((.........-.........------.............)))))).)))))))...(((((((((((.....))))))...--.)))--))...--. ( -19.28) >DroMoj_CAF1 21622 99 + 1 U--CUCUUUUUUACAUCACCUUUUAU---UUGUUUUCUCUAU------U---UUCUCUUCUAUUUGUAGAUGUAGUCAGUGUGUUUGGCGAUAAGCUAAAAUA--UCAU--AAAUA--C .--.......(((((((.........---.............------.---................)))))))...(((..((((((.....))))))...--.)))--.....--. ( -10.53) >DroAna_CAF1 16186 103 + 1 ------AA--CCACAUCACGAAUUAUUCUCUA-UUUGUUUCUC-----UUCUCUCUCUUCUAUUUGUAGAUGUAGUCAGUGUGUUUGGCGAUAAGCUAAAAUAUACCAUAUAUAAA--C ------.(--(.(((((((((((.........-..........-----.............)))))).))))).))..(((((((((((.....))).))))))))..........--. ( -16.15) >DroPer_CAF1 16579 112 + 1 ACCUUCUG--CCACAUCACGAAUUAUUCUCUA-UAUUUUUCUUGUUUUCUCUCUCUCUUCUAUUUGUAGAUGUAGUCAGUGUGUUUGGCGAUAAGCUAAAAUA--CCAU--AUAUAUAC .....(((--(.(((((((((((.........-............................)))))).))))).).))).(((((((((.....))).)))))--)...--........ ( -15.79) >consensus ______UA__CUACAUCACGAAUUAUUCUCUA_UUUCUCUAU______UUCUCUCUCUUCUAUUUGUAGAUGUAGUCAGUGUGUUUGGCGAUAAGCUAAAAUA__CCAU__AUAAA__C ..........(((((((((((((......................................)))))).)))))))........((((((.....))))))................... (-10.55 = -11.78 + 1.22)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:00:02 2006