
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 3,414,599 – 3,414,717 |
| Length | 118 |
| Max. P | 0.898026 |

| Location | 3,414,599 – 3,414,717 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
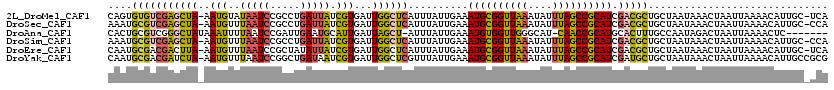
| Mean pairwise identity | 84.66 |
| Mean single sequence MFE | -30.77 |
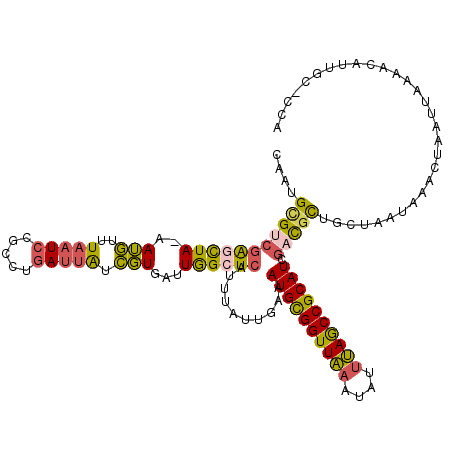
| Consensus MFE | -24.86 |
| Energy contribution | -24.65 |
| Covariance contribution | -0.21 |
| Combinations/Pair | 1.41 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.00 |
| SVM RNA-class probability | 0.898026 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 3414599 118 - 22407834 CAGUGUGUCGAGCUA-AAUGUAUAAUCCGCCUGAUUAUCGUGAUUGGCUCAUUUAUUGAAAUGCGGUUAAAUAUUUAGCCGCAUCGACGCUGCUAAUAAACUAAUUAAAACAUUGC-UCA .((((((((((((((-((((.((((((.....)))))))))..)))))))..........((((((((((....)))))))))).))))).)))......................-... ( -34.30) >DroSec_CAF1 3516 118 - 1 AAAUGCGUCGAGCUA-AAUGUUUAAUCCGCCUGAUUAUCGUGAUUGGCUCAUUUAUUGAAAUGCGGUUAAAUAUUUAGCCGCAUCGACGCUGCUAAUAAACUAAUUAAAACAUUGC-CCA ....(((((((((((-(((((((((.(((((..(((.....)))..).(((.....)))...)))))))))))))))))....)))))))..........................-... ( -34.00) >DroAna_CAF1 6236 111 - 1 CACUGCGUCGGGCUAUAAAUUUUAAUCCGAUUGAAUGCAUUGAUUAGCU-AUUUAUUGAAAUGUGGUUGGGCAU-CAACCGCAUGCACUUUGCCAAUAGACUAAUUAAAACUC------- ...((((((((..((.......))..))))).....)))((((((((..-.(((((((..(((((((((.....-)))))))))((.....))))))))))))))))).....------- ( -29.70) >DroSim_CAF1 3770 118 - 1 AAAUGCGUCGAGCUA-AAUGUUUAAUCCGCCUGAUUAUCGUGAUUGGCUCAUUUAUUGAAAUGCGGUUAAAUAUUUAGCCGCAUCGACGCUGCUAAUAAACUAAUUAAAACAUUGC-CCA ....(((((((((((-(((((((((.(((((..(((.....)))..).(((.....)))...)))))))))))))))))....)))))))..........................-... ( -34.00) >DroEre_CAF1 3752 118 - 1 CAAUGCGACGACUUA-AAUGUUUAAUCCGCUAUAUUAUCGUGAUUGGCUCAUUUAUUGAAAUGCGGUUAAAUAUUUAGCCGCAUCGACGCUGCUAAUAAACUAAUUAAAACAUUGC-UCA ....(((.(((..((-((((.((((((((.........)).))))))..))))))......(((((((((....)))))))))))).)))..........................-... ( -24.70) >DroYak_CAF1 4182 119 - 1 CAAUGCGACGAUCUA-AAUGUUUAAUCCGGCUGAUAAUCGUGAUUGGCUCGUUUAUUGAAAUGCGGUUAAAUAUUUAGCCGCAUCGAUGCUGCUAAUAAACUAAUUAAAACAUUGCCGCG ....(((.((((.((-(..(((((....(((..((.......))..))).((.((((((..(((((((((....)))))))))))))))..))...)))))...)))....)))).))). ( -27.90) >consensus CAAUGCGUCGAGCUA_AAUGUUUAAUCCGCCUGAUUAUCGUGAUUGGCUCAUUUAUUGAAAUGCGGUUAAAUAUUUAGCCGCAUCGACGCUGCUAAUAAACUAAUUAAAACAUUGC_CCA ....(((((((((((..(((..(((((.....))))).)))...))))))..........((((((((((....)))))))))).))))).............................. (-24.86 = -24.65 + -0.21)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:58:30 2006