
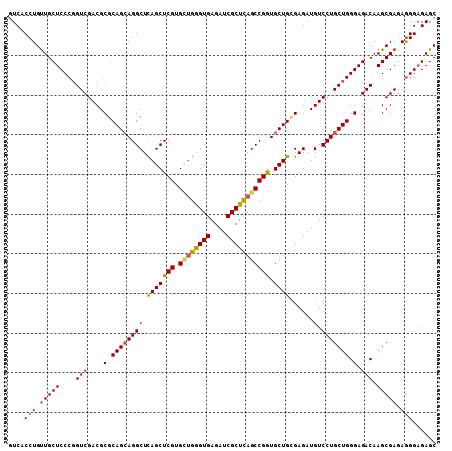
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 3,365,593 – 3,365,701 |
| Length | 108 |
| Max. P | 0.960721 |

| Location | 3,365,593 – 3,365,701 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 95.06 |
| Mean single sequence MFE | -49.63 |
| Consensus MFE | -44.37 |
| Energy contribution | -44.48 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.56 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.01 |
| SVM RNA-class probability | 0.899410 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 3365593 108 + 22407834 GUCACCUGUUGCUCCCGGUCGACGCGCAGCAGGCUCAGCUCGUGCUGGGUGAGAUCGCUCAGCCGAUGCUGCGAGAUGUCCUGCUGGGAGACAAGCGAAAGGGAGAGC .......(((.(((((..(((.(...((((((((.(((((((.((((((((....))))))))))).))))......).)))))))(....)..))))..)))))))) ( -50.20) >DroSec_CAF1 56302 108 + 1 GUCACCUGUUGCUCCCGGUCGACGCGCAGCAGGCUCAGCUCGUGUUGGGUGAGAUCGCUCAGCCGGUGCUGCGAGAUGUCCUGCUGGGAGACAAGCGAGAGGGUGAGC .(((((((((.((((((((.(((.((((((((((...)))((.((((((((....)))))))))).)))))))....)))..))))))))...)))....)))))).. ( -50.50) >DroSim_CAF1 52387 108 + 1 GUCACCUGUUGCUCCCGGUCGACGCGCAGCAGGCUUAGCUCGUGUUGGGUGAGAUCGCUUAGCCGGUGCUGCGAGAUGUCCUGCUGGGAGACAAGCGAGAGGGAGAGC .((.(((.(((((((((((.(((.((((((((((...)))((.((((((((....)))))))))).)))))))....)))..))))))).....)))).))))).... ( -45.70) >DroEre_CAF1 50830 108 + 1 GUCACCUGUUGCUCCCGGUCGACGCGCAGCAGGCUCAGCUCGUGCUGGGUGAGAUCGCUCAGCCGGUGCUGUGAGAUGUCCUGCUGGGAGACAAGCGAGAAGGAGAGC ..........((((((..((..(((.((((((((.(((((((.((((((((....))))))))))).))))......).)))))))(....)..))).)).)).)))) ( -49.10) >DroYak_CAF1 52088 108 + 1 GUCACCUGUUGCUCCCGGUCGACGCGCAGCAGGCUCAGCUCGUGCUGGGUGAGAUCGCUCAGCCGGUGCUGCGAGAUGUCCUGCUGGGAGACAAGCGAGAGGGAGAGC .((.(((.(((((....(((....(.(((((((..((.((((((((((.((((....)))).)))))...))))).)).))))))).).))).))))).))))).... ( -51.30) >DroAna_CAF1 60481 108 + 1 GUCACCUGCUGCUCCCUGUCGACGCGCAGGAGGCUCAGCUCGUGGGGAGUGAGGUCGCUCAGCCGGUGCUGGGAGAUGUCCUGCUGGGAGACAAGCGAGAGGGAGAGC .......(((.((((((.(((.(..((((((.(((((((.(.(((.(((((....)))))..)))).))))))...).))))))..(....)..)))).))))))))) ( -51.00) >consensus GUCACCUGUUGCUCCCGGUCGACGCGCAGCAGGCUCAGCUCGUGCUGGGUGAGAUCGCUCAGCCGGUGCUGCGAGAUGUCCUGCUGGGAGACAAGCGAGAGGGAGAGC ....(((.(((((....(((....(.((((((((.(((((((.((((((((....))))))))))).))))......).))))))).).))).))))).)))...... (-44.37 = -44.48 + 0.11)


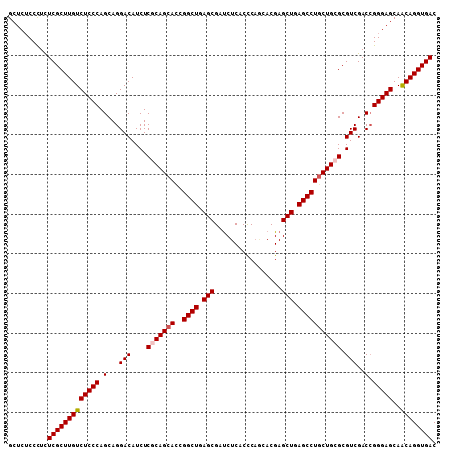
| Location | 3,365,593 – 3,365,701 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 95.06 |
| Mean single sequence MFE | -40.98 |
| Consensus MFE | -38.22 |
| Energy contribution | -38.58 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.44 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.52 |
| SVM RNA-class probability | 0.960721 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 3365593 108 - 22407834 GCUCUCCCUUUCGCUUGUCUCCCAGCAGGACAUCUCGCAGCAUCGGCUGAGCGAUCUCACCCAGCACGAGCUGAGCCUGCUGCGCGUCGACCGGGAGCAACAGGUGAC ..........(((((((((((((....((.(..(.(((((((..(((((((....)))...((((....))))))))))))))).)..).)))))))..)))))))). ( -43.20) >DroSec_CAF1 56302 108 - 1 GCUCACCCUCUCGCUUGUCUCCCAGCAGGACAUCUCGCAGCACCGGCUGAGCGAUCUCACCCAACACGAGCUGAGCCUGCUGCGCGUCGACCGGGAGCAACAGGUGAC ..........(((((((((((((....((.(..(.(((((((..((((.(((.................))).))))))))))).)..).)))))))..)))))))). ( -42.13) >DroSim_CAF1 52387 108 - 1 GCUCUCCCUCUCGCUUGUCUCCCAGCAGGACAUCUCGCAGCACCGGCUAAGCGAUCUCACCCAACACGAGCUAAGCCUGCUGCGCGUCGACCGGGAGCAACAGGUGAC ..........(((((((((((((....((.(..(.(((((((..((((.(((.................))).))))))))))).)..).)))))))..)))))))). ( -40.03) >DroEre_CAF1 50830 108 - 1 GCUCUCCUUCUCGCUUGUCUCCCAGCAGGACAUCUCACAGCACCGGCUGAGCGAUCUCACCCAGCACGAGCUGAGCCUGCUGCGCGUCGACCGGGAGCAACAGGUGAC ..........(((((((((((((((((((.((((.(.((((....)))).).)))......((((....)))).))))))).((...))...)))))..)))))))). ( -38.30) >DroYak_CAF1 52088 108 - 1 GCUCUCCCUCUCGCUUGUCUCCCAGCAGGACAUCUCGCAGCACCGGCUGAGCGAUCUCACCCAGCACGAGCUGAGCCUGCUGCGCGUCGACCGGGAGCAACAGGUGAC ..........(((((((((((((....((.(..(.(((((((..(((((((....)))...((((....))))))))))))))).)..).)))))))..)))))))). ( -43.10) >DroAna_CAF1 60481 108 - 1 GCUCUCCCUCUCGCUUGUCUCCCAGCAGGACAUCUCCCAGCACCGGCUGAGCGACCUCACUCCCCACGAGCUGAGCCUCCUGCGCGUCGACAGGGAGCAGCAGGUGAC (((((((((.(((..(((......((((((..((.(.((((....)))).).)).(((((((.....))).))))..))))))))).))).)))))).)))....... ( -39.10) >consensus GCUCUCCCUCUCGCUUGUCUCCCAGCAGGACAUCUCGCAGCACCGGCUGAGCGAUCUCACCCAGCACGAGCUGAGCCUGCUGCGCGUCGACCGGGAGCAACAGGUGAC ..........(((((((((((((.(...(((....(((((((..((((.(((.................))).))))))))))).)))..).)))))..)))))))). (-38.22 = -38.58 + 0.36)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:58:10 2006