
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 3,110,408 – 3,110,568 |
| Length | 160 |
| Max. P | 0.999699 |

| Location | 3,110,408 – 3,110,528 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.33 |
| Mean single sequence MFE | -46.90 |
| Consensus MFE | -40.18 |
| Energy contribution | -40.22 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.93 |
| SVM RNA-class probability | 0.983027 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 3110408 120 + 22407834 UCCGGAUCGCUAUGGAUUCGGCUGGACAUCGAGUGAUCGAGGUGUUAGCUAUCUCUAUGGUCGCGAUGUCCUCGAGAAGUUUCUGCAAAAGAACGACUUCGAUCUGGUGUGUCGUGCCCA .(((((((.........((((..(((((((..((((((((((((.....))))))...)))))))))))))))))(((((((((.....)))..)))))))))))))............. ( -41.40) >DroSec_CAF1 35528 120 + 1 UCCGGAUCGCUAUGGCUUCGGCUGGACAUCGAGUGAUCGGGGUGUUAGCUACCUCUAUGGGCGCGAUGUCCUCGAGAAGUUUCUGCAAAAGAACGACUUCGAUCUGGUGUGUCGUGCCCA ....(((((((.((((....)))).......)))))))((((((.....))))))..(((((((((..(((....(((((((((.....)))..)))))).....)).)..))))))))) ( -47.80) >DroSim_CAF1 35817 120 + 1 UCCGGAUCGCUAUGGCUUCGGCUGGACAUCGAGUGAUCGGGGUGUUAGCUACCUCUAUGGGCGCGAUGUACUCGAGAAGUUUCUGCAAAAGAACGACUUCGAUCUGGUGUGUCGUGCCCA ....(((((((.((((....)))).......)))))))((((((.....))))))..(((((((((..((((.(((((((((((.....)))..))))))..)).))))..))))))))) ( -51.20) >DroEre_CAF1 36196 120 + 1 UCCGGACCGCUAUGGUUUCGGCUGGACACCGAGUGAUCGGGGUGUUAGCUACCUCUAUGGGCGCGAUGUCAUCGAGAGGUUUCUGCAAAAGAAUGACUUCGAUCUGGUGUGUCGUGCCCA (((((.(((.........))))))))..((((....))))((((.....))))....(((((((((((.(((((.(((((((((.....)))..))))))....)))))))))))))))) ( -46.90) >DroYak_CAF1 36702 120 + 1 UCCGGAUCGCUAUGGUUUCGGCUGGACAGCGAGUGAUCGGGGUGUUAGCUACCUCUAUGGGCGCGAUGUGCUCGAGAAGUUUCUGCAAAGGAAUGACUUCGAUCUGGUGUGUCGUGCCCA ....(((((((...((((.....))))....)))))))((((((.....))))))..(((((((((((..((.(((((((((((.....)))..))))))..)).))..))))))))))) ( -47.20) >consensus UCCGGAUCGCUAUGGCUUCGGCUGGACAUCGAGUGAUCGGGGUGUUAGCUACCUCUAUGGGCGCGAUGUCCUCGAGAAGUUUCUGCAAAAGAACGACUUCGAUCUGGUGUGUCGUGCCCA ....(((((((.(((..((.....))..))))))))))((((((.....))))))..(((((((((((..((.(((((((((((.....)))..))))))..)).))..))))))))))) (-40.18 = -40.22 + 0.04)

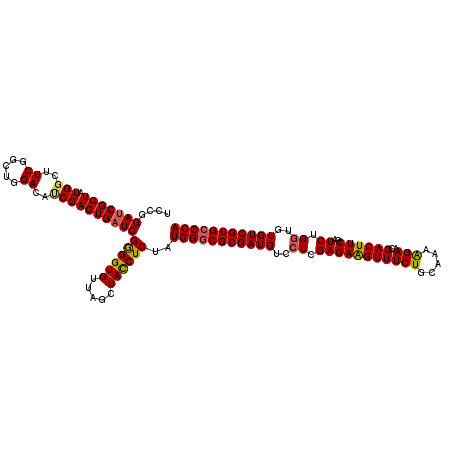
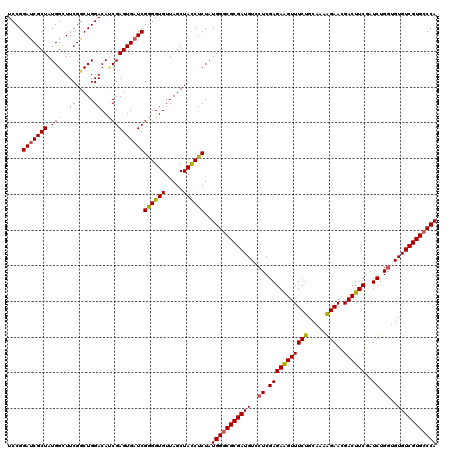
| Location | 3,110,448 – 3,110,568 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.58 |
| Mean single sequence MFE | -46.68 |
| Consensus MFE | -43.74 |
| Energy contribution | -43.66 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.45 |
| Structure conservation index | 0.94 |
| SVM decision value | 3.91 |
| SVM RNA-class probability | 0.999699 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 3110448 120 + 22407834 GGUGUUAGCUAUCUCUAUGGUCGCGAUGUCCUCGAGAAGUUUCUGCAAAAGAACGACUUCGAUCUGGUGUGUCGUGCCCACCAGGUGGUGGAGGAUGGAUACGAGUUCUUUGCCAAACGC .(((((....((((...(((.(((((..(((....(((((((((.....)))..)))))).....)).)..))))).)))..))))((..((((((........))))))..)).))))) ( -37.40) >DroSec_CAF1 35568 120 + 1 GGUGUUAGCUACCUCUAUGGGCGCGAUGUCCUCGAGAAGUUUCUGCAAAAGAACGACUUCGAUCUGGUGUGUCGUGCCCACCAGGUGGUGGAGGAUGGAUACGAGUUCUUUGCCAAACGC .(((((....((((...(((((((((..(((....(((((((((.....)))..)))))).....)).)..)))))))))..))))((..((((((........))))))..)).))))) ( -47.10) >DroSim_CAF1 35857 120 + 1 GGUGUUAGCUACCUCUAUGGGCGCGAUGUACUCGAGAAGUUUCUGCAAAAGAACGACUUCGAUCUGGUGUGUCGUGCCCACCAGGUGGUGGAGGAUGGAUACGAGUUCUUUGCCAAACGC .(((((....((((...(((((((((..((((.(((((((((((.....)))..))))))..)).))))..)))))))))..))))((..((((((........))))))..)).))))) ( -50.50) >DroEre_CAF1 36236 120 + 1 GGUGUUAGCUACCUCUAUGGGCGCGAUGUCAUCGAGAGGUUUCUGCAAAAGAAUGACUUCGAUCUGGUGUGUCGUGCCCAUCAGGUGGUGGAGGAUGGUUACGAGUUCUUUGCCAAACGC .(((((....((((..((((((((((((.(((((.(((((((((.....)))..))))))....))))))))))))))))).))))((..((((((........))))))..)).))))) ( -49.20) >DroYak_CAF1 36742 120 + 1 GGUGUUAGCUACCUCUAUGGGCGCGAUGUGCUCGAGAAGUUUCUGCAAAGGAAUGACUUCGAUCUGGUGUGUCGUGCCCACCAGGUGGUGGAGGAUGGAUACGAGUUCUUUGCCAAACGC .(((((....((((...(((((((((((..((.(((((((((((.....)))..))))))..)).))..)))))))))))..))))((..((((((........))))))..)).))))) ( -49.20) >consensus GGUGUUAGCUACCUCUAUGGGCGCGAUGUCCUCGAGAAGUUUCUGCAAAAGAACGACUUCGAUCUGGUGUGUCGUGCCCACCAGGUGGUGGAGGAUGGAUACGAGUUCUUUGCCAAACGC .(((((....((((...(((((((((((..((.(((((((((((.....)))..))))))..)).))..)))))))))))..))))((..((((((........))))))..)).))))) (-43.74 = -43.66 + -0.08)

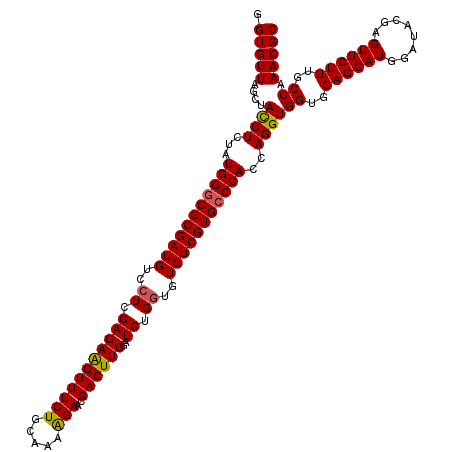
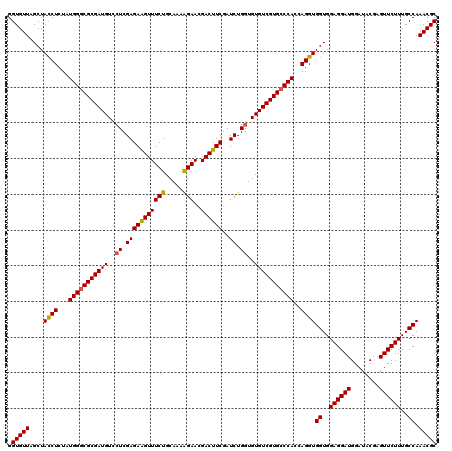
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:54:42 2006