
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 3,046,720 – 3,046,859 |
| Length | 139 |
| Max. P | 0.996880 |

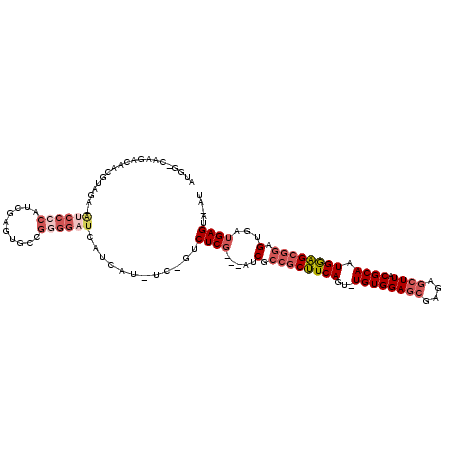
| Location | 3,046,720 – 3,046,830 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 71.34 |
| Mean single sequence MFE | -40.37 |
| Consensus MFE | -23.14 |
| Energy contribution | -25.34 |
| Covariance contribution | 2.20 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.57 |
| SVM decision value | 2.76 |
| SVM RNA-class probability | 0.996880 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 3046720 110 + 22407834 AUGG-CAAGCCAACGUAGA-AUCCCCAUCGAGUGUCGGGGAUCAUCAU----GUCUCG---AUCGCCGCUUCAGU-UGUGGAGCGAGAGCUUACGCAAUGGAGCGGAGUGAUGAGCACAU .(((-....))).....((-.(((((..(....)..)))))))...((----(((((.---(((.((((((((.(-((((((((....)))).)))))))))))))...)))))).)))) ( -44.10) >DroSec_CAF1 6359 113 + 1 AUGC-CAAUACAACGUAGA-AUCCCCAUCGAGUGCCGGGGAUCAUUCUAUCUAUCUCG---AUCGCCGCUUCAGU-UGUGGAGCGAGAGCUUACGCAAUGGAGCGGAGUGAUGAGUA-GU ....-.........(((((-((((((..........))))))...)))))(((.(((.---(((.((((((((.(-((((((((....)))).)))))))))))))...))))))))-). ( -41.90) >DroSim_CAF1 6594 113 + 1 AUGC-CAAUACAACGUAGA-GUCCCCAUCGAGUGUAGGGGAUCAUUUUAUCUAUCUCG---AUCGCCGCUUCAGU-UGUGGAGCGAGAGCUUACGCAAUGGAGCGGAGUGAUGAGUA-GU ....-.........(((((-((((((..........))))))...)))))(((.(((.---(((.((((((((.(-((((((((....)))).)))))))))))))...))))))))-). ( -39.60) >DroYak_CAF1 6559 110 + 1 AUGG-CAAGUCCCAGUAGA-ACCCCCAUCGACGACUGUGGAUCAGCAU----GUCUCG---AUCGCCGCUUCAGU-UGUGGAGCGAGAGCUUACGCAAUGGAGCGGAGUGAUGAGCACAU ...(-(..((((((((.((-.......))....)))).))))..))((----(((((.---(((.((((((((.(-((((((((....)))).)))))))))))))...)))))).)))) ( -43.40) >DroAna_CAF1 5956 110 + 1 CUCGUCACAUUCAUGUAGACAGGCCCCUCGGUUUC-----AACGUCAAGUC-GACUCCAAACUCCCCUCGUCAGUUUGUGGAACACCA-AUUCCGCAUUGAUGUUUGGGACUGAGUA--- ...((((((....))).)))......((((((..(-----((((((((...-(((..............)))....(((((((.....-.))))))))))))).)))..))))))..--- ( -32.84) >consensus AUGG_CAAGACAACGUAGA_AUCCCCAUCGAGUGCCGGGGAUCAUCAU_UC_GUCUCG___AUCGCCGCUUCAGU_UGUGGAGCGAGAGCUUACGCAAUGGAGCGGAGUGAUGAGUA_AU ....................((((((..........))))))............((((.....(.((((((((...((((((((....)))).)))).)))))))).)...))))..... (-23.14 = -25.34 + 2.20)

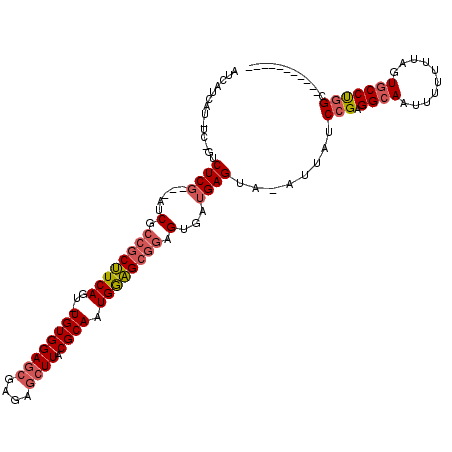
| Location | 3,046,758 – 3,046,859 |
|---|---|
| Length | 101 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.81 |
| Mean single sequence MFE | -36.88 |
| Consensus MFE | -25.78 |
| Energy contribution | -26.74 |
| Covariance contribution | 0.96 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.36 |
| Structure conservation index | 0.70 |
| SVM decision value | 2.31 |
| SVM RNA-class probability | 0.992114 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 3046758 101 + 22407834 AUCAUCAU----GUCUCG---AUCGCCGCUUCAGU-UGUGGAGCGAGAGCUUACGCAAUGGAGCGGAGUGAUGAGCACAUUAUCCGAGGCAAUUUUUUUAGUGCCUGGC----------- ......((----(((((.---(((.((((((((.(-((((((((....)))).)))))))))))))...)))))).))))...((.(((((..........))))))).----------- ( -38.80) >DroSec_CAF1 6397 104 + 1 AUCAUUCUAUCUAUCUCG---AUCGCCGCUUCAGU-UGUGGAGCGAGAGCUUACGCAAUGGAGCGGAGUGAUGAGUA-GUUAUCCGAGGCAAUUUUUUUAGUGCCUGGC----------- ..........(((.(((.---(((.((((((((.(-((((((((....)))).)))))))))))))...))))))))-)....((.(((((..........))))))).----------- ( -37.40) >DroSim_CAF1 6632 104 + 1 AUCAUUUUAUCUAUCUCG---AUCGCCGCUUCAGU-UGUGGAGCGAGAGCUUACGCAAUGGAGCGGAGUGAUGAGUA-GUUAUCCGAGGCAAUUUUUUUAGUGCCUGGC----------- ..........(((.(((.---(((.((((((((.(-((((((((....)))).)))))))))))))...))))))))-)....((.(((((..........))))))).----------- ( -37.40) >DroYak_CAF1 6597 102 + 1 AUCAGCAU----GUCUCG---AUCGCCGCUUCAGU-UGUGGAGCGAGAGCUUACGCAAUGGAGCGGAGUGAUGAGCACAUUAUCCGAGGCAAUUUUUUUAGUGCCUAGCC---------- ....((((----(((((.---(((.((((((((.(-((((((((....)))).)))))))))))))...)))))).))))......(((((..........))))).)).---------- ( -39.00) >DroAna_CAF1 5991 115 + 1 AACGUCAAGUC-GACUCCAAACUCCCCUCGUCAGUUUGUGGAACACCA-AUUCCGCAUUGAUGUUUGGGACUGAGUA---UAUCCGAGGCAAUUUUUUGAGUACCCGGCUGCAACUGGGU .((.(((((..-..(((...(((((((.((((((..(((((((.....-.)))))))))))))...)))...)))).---.....))).......)))))))((((((......)))))) ( -31.80) >consensus AUCAUCAU_UC_GUCUCG___AUCGCCGCUUCAGU_UGUGGAGCGAGAGCUUACGCAAUGGAGCGGAGUGAUGAGUA_AUUAUCCGAGGCAAUUUUUUUAGUGCCUGGC___________ ..............((((.....(.((((((((...((((((((....)))).)))).)))))))).)...))))........(((.((((..........)))))))............ (-25.78 = -26.74 + 0.96)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:54:20 2006