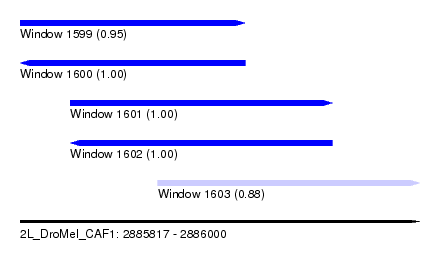
| Sequence ID | 2L_DroMel_CAF1 |
|---|---|
| Location | 2,885,817 – 2,886,000 |
| Length | 183 |
| Max. P | 0.999616 |

| Location | 2,885,817 – 2,885,920 |
|---|---|
| Length | 103 |
| Sequences | 5 |
| Columns | 103 |
| Reading direction | forward |
| Mean pairwise identity | 90.58 |
| Mean single sequence MFE | -36.44 |
| Consensus MFE | -27.56 |
| Energy contribution | -27.48 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.53 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.38 |
| SVM RNA-class probability | 0.948621 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2885817 103 + 22407834 AGCAAAUAUGCGGUACCGACAUUUUCCAGAACUGGACCUAUCGUCGCACAGCUGAUCGCCCGCCAAUCAGCUGGGCGAUAGUUGCCGUAUCGAUAGCCGACCC .(((....)))(((..........((((....)))).((((((..((.((((((.((((((((......)).))))))))))))..))..))))))...))). ( -36.30) >DroSec_CAF1 4534 103 + 1 AGCAAAUAUGCAGUACCGACAUUUACCAGCACUGGACCUAUCGCCGCACAGCUGAUCGCCCGCCAAUCAGCUGGGCGAUAGUUGCUGUAUCGAUAGCCGACCC .(((....))).....((.......(((....)))..((((((..(((((((((.((((((((......)).)))))))))))).)))..)))))).)).... ( -34.70) >DroSim_CAF1 4600 103 + 1 AGCAAAUAUGCAGUACCGACAAUUACCAGCACUGGACCUAUCGCCGCACAGCUGAUUGCCCGCCAAUCAGCUGGGCGAUAGUUGCUGUAUCGAUAGCCGACCC .((....((((((((..........(((....)))..((((((((...((((((((((.....)))))))))))))))))).)))))))).....))...... ( -38.40) >DroEre_CAF1 4671 103 + 1 AGGAAAAAUGCCGUACCGACAUUUUUCAACACUGGACCUAUCGCGGCACAGCUGAUCGCCCGCCAAUCAGCUGGGCGAUAGUUGCCGUAUCGAUAGCCGAGCC ..((((((((.((...)).)))))))).....(((..(((((((((((..((((.((((((((......)).)))))))))))))))...))))))))).... ( -38.60) >DroYak_CAF1 4828 103 + 1 AGAAAAAAUGUUGUACCGACAUUUGCCAGCACUGAACCUAUCGCUGCACAGCUGAUCGCCCGCCAAUCAGCUGGGCGAUAGUUGCCGUGUCGAUAACCGUGGC .....((((((((...))))))))((((((((.(...((((((((...((((((((.(.....).))))))))))))))))...).))))((.....)))))) ( -34.20) >consensus AGCAAAUAUGCAGUACCGACAUUUACCAGCACUGGACCUAUCGCCGCACAGCUGAUCGCCCGCCAAUCAGCUGGGCGAUAGUUGCCGUAUCGAUAGCCGACCC .......((((((((..........(((....)))..((((((((...((((((((.(.....).)))))))))))))))).))))))))............. (-27.56 = -27.48 + -0.08)

| Location | 2,885,817 – 2,885,920 |
|---|---|
| Length | 103 |
| Sequences | 5 |
| Columns | 103 |
| Reading direction | reverse |
| Mean pairwise identity | 90.58 |
| Mean single sequence MFE | -43.76 |
| Consensus MFE | -37.26 |
| Energy contribution | -37.02 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.15 |
| Mean z-score | -3.12 |
| Structure conservation index | 0.85 |
| SVM decision value | 3.79 |
| SVM RNA-class probability | 0.999616 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2885817 103 - 22407834 GGGUCGGCUAUCGAUACGGCAACUAUCGCCCAGCUGAUUGGCGGGCGAUCAGCUGUGCGACGAUAGGUCCAGUUCUGGAAAAUGUCGGUACCGCAUAUUUGCU .((((.....(((.((((((....(((((((.(((....))))))))))..)))))))))(((((..((((....))))...)))))).)))(((....))). ( -41.60) >DroSec_CAF1 4534 103 - 1 GGGUCGGCUAUCGAUACAGCAACUAUCGCCCAGCUGAUUGGCGGGCGAUCAGCUGUGCGGCGAUAGGUCCAGUGCUGGUAAAUGUCGGUACUGCAUAUUUGCU (..(((.....)))..)(((((((((((((((((((((((.....))))))))))...))))))))((.(((((((((......))))))))).))..))))) ( -45.70) >DroSim_CAF1 4600 103 - 1 GGGUCGGCUAUCGAUACAGCAACUAUCGCCCAGCUGAUUGGCGGGCAAUCAGCUGUGCGGCGAUAGGUCCAGUGCUGGUAAUUGUCGGUACUGCAUAUUUGCU (..(((.....)))..)(((((((((((((((((((((((.....))))))))))...))))))))((.(((((((((......))))))))).))..))))) ( -46.00) >DroEre_CAF1 4671 103 - 1 GGCUCGGCUAUCGAUACGGCAACUAUCGCCCAGCUGAUUGGCGGGCGAUCAGCUGUGCCGCGAUAGGUCCAGUGUUGAAAAAUGUCGGUACGGCAUUUUUCCU .(((.((((((((...(((((.(((((((((.(((....)))))))))).))...)))))))))).))).)))...(((((((((((...))))))))))).. ( -44.70) >DroYak_CAF1 4828 103 - 1 GCCACGGUUAUCGACACGGCAACUAUCGCCCAGCUGAUUGGCGGGCGAUCAGCUGUGCAGCGAUAGGUUCAGUGCUGGCAAAUGUCGGUACAACAUUUUUUCU (((...(((...)))..)))..((((((((((((((((((.....)))))))))).)..))))))).....((((((((....))))))))............ ( -40.80) >consensus GGGUCGGCUAUCGAUACGGCAACUAUCGCCCAGCUGAUUGGCGGGCGAUCAGCUGUGCGGCGAUAGGUCCAGUGCUGGAAAAUGUCGGUACUGCAUAUUUGCU ......(((((((((((((((.((((((((((((((((((.....))))))))))...))))))))......))))).....))))))))..))......... (-37.26 = -37.02 + -0.24)


| Location | 2,885,840 – 2,885,960 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.42 |
| Mean single sequence MFE | -38.23 |
| Consensus MFE | -34.30 |
| Energy contribution | -34.02 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.89 |
| SVM RNA-class probability | 0.997576 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2885840 120 + 22407834 UUCCAGAACUGGACCUAUCGUCGCACAGCUGAUCGCCCGCCAAUCAGCUGGGCGAUAGUUGCCGUAUCGAUAGCCGACCCAUCGAUAACGAUAUGUGUAUUUUCCUGCUCUCUCUGCACC .((((....)))).((((((((...((((((((.(.....).)))))))))))))))).((((((((((.((..(((....))).)).))))))).)))......(((.......))).. ( -37.00) >DroSec_CAF1 4557 120 + 1 UACCAGCACUGGACCUAUCGCCGCACAGCUGAUCGCCCGCCAAUCAGCUGGGCGAUAGUUGCUGUAUCGAUAGCCGACCCAUCGAUAACGAUAUGUGUAUUUUCCUGCUCUCUCUGCACC .....(((((((..((((((..(((((((((.((((((((......)).)))))))))))).)))..)))))))))....((((....))))..)))).......(((.......))).. ( -38.50) >DroSim_CAF1 4623 120 + 1 UACCAGCACUGGACCUAUCGCCGCACAGCUGAUUGCCCGCCAAUCAGCUGGGCGAUAGUUGCUGUAUCGAUAGCCGACCCAUCGAUAACGAUAUGUGUAUUUUCCUGCUCUCUCUGCACC ....((((..(((.((((((((...((((((((((.....)))))))))))))))))).((((((((((.((..(((....))).)).))))))).)))...)))))))........... ( -41.10) >DroEre_CAF1 4694 120 + 1 UUUCAACACUGGACCUAUCGCGGCACAGCUGAUCGCCCGCCAAUCAGCUGGGCGAUAGUUGCCGUAUCGAUAGCCGAGCCAUCGAUAACGAUAUGUGUAUUUUCCUGCUCUCUCUGCACC .........((.((.((((((((((..((((.((((((((......)).)))))))))))))))(((((((.((...)).))))))).))))).)).))......(((.......))).. ( -39.50) >DroYak_CAF1 4851 120 + 1 UGCCAGCACUGAACCUAUCGCUGCACAGCUGAUCGCCCGCCAAUCAGCUGGGCGAUAGUUGCCGUGUCGAUAACCGUGGCAUCGAUAACGAUAUGUGUAUUUUCCUGCUCUCUCUGCACC .((((((((.(...((((((((...((((((((.(.....).))))))))))))))))...).))))((.....))))))((((....))))..(((((...............))))). ( -35.06) >consensus UACCAGCACUGGACCUAUCGCCGCACAGCUGAUCGCCCGCCAAUCAGCUGGGCGAUAGUUGCCGUAUCGAUAGCCGACCCAUCGAUAACGAUAUGUGUAUUUUCCUGCUCUCUCUGCACC ..........(((.((((((((...((((((((.(.....).)))))))))))))))).((((((((((.((..((......)).)).))))))).)))...)))(((.......))).. (-34.30 = -34.02 + -0.28)

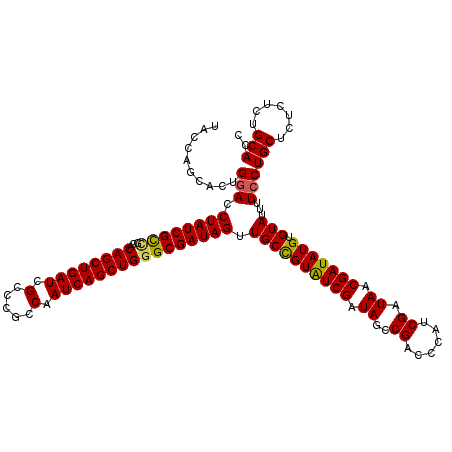
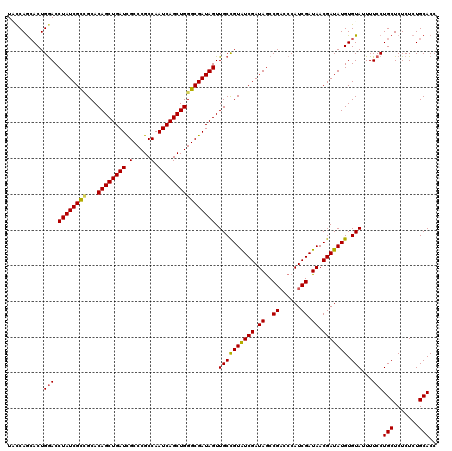
| Location | 2,885,840 – 2,885,960 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.42 |
| Mean single sequence MFE | -44.98 |
| Consensus MFE | -39.82 |
| Energy contribution | -40.46 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.26 |
| Structure conservation index | 0.89 |
| SVM decision value | 3.07 |
| SVM RNA-class probability | 0.998343 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2885840 120 - 22407834 GGUGCAGAGAGAGCAGGAAAAUACACAUAUCGUUAUCGAUGGGUCGGCUAUCGAUACGGCAACUAUCGCCCAGCUGAUUGGCGGGCGAUCAGCUGUGCGACGAUAGGUCCAGUUCUGGAA .........(((((.(((...........((((.((((((((.....))))))))))))...((((((((((((((((((.....)))))))))).).).)))))).))).))))).... ( -44.40) >DroSec_CAF1 4557 120 - 1 GGUGCAGAGAGAGCAGGAAAAUACACAUAUCGUUAUCGAUGGGUCGGCUAUCGAUACAGCAACUAUCGCCCAGCUGAUUGGCGGGCGAUCAGCUGUGCGGCGAUAGGUCCAGUGCUGGUA ..(((.......)))......(((.........(((((((((.....)))))))))(((((.((((((((((((((((((.....))))))))))...))))))))......)))))))) ( -46.70) >DroSim_CAF1 4623 120 - 1 GGUGCAGAGAGAGCAGGAAAAUACACAUAUCGUUAUCGAUGGGUCGGCUAUCGAUACAGCAACUAUCGCCCAGCUGAUUGGCGGGCAAUCAGCUGUGCGGCGAUAGGUCCAGUGCUGGUA ..(((.......)))......(((.........(((((((((.....)))))))))(((((.((((((((((((((((((.....))))))))))...))))))))......)))))))) ( -47.00) >DroEre_CAF1 4694 120 - 1 GGUGCAGAGAGAGCAGGAAAAUACACAUAUCGUUAUCGAUGGCUCGGCUAUCGAUACGGCAACUAUCGCCCAGCUGAUUGGCGGGCGAUCAGCUGUGCCGCGAUAGGUCCAGUGUUGAAA ..(((.......)))....(((((((.((((((((((((((((...)))))))))).((((.(((((((((.(((....)))))))))).))...)))))))))).))...))))).... ( -44.90) >DroYak_CAF1 4851 120 - 1 GGUGCAGAGAGAGCAGGAAAAUACACAUAUCGUUAUCGAUGCCACGGUUAUCGACACGGCAACUAUCGCCCAGCUGAUUGGCGGGCGAUCAGCUGUGCAGCGAUAGGUUCAGUGCUGGCA ........(..((((.((....((...(((((((.(((((((....).))))))((((((....(((((((.(((....))))))))))..)))))).))))))).))))..))))..). ( -41.90) >consensus GGUGCAGAGAGAGCAGGAAAAUACACAUAUCGUUAUCGAUGGGUCGGCUAUCGAUACGGCAACUAUCGCCCAGCUGAUUGGCGGGCGAUCAGCUGUGCGGCGAUAGGUCCAGUGCUGGAA ..(((.......)))..................(((((((((.....)))))))))(((((.((((((((((((((((((.....))))))))))...))))))))......)))))... (-39.82 = -40.46 + 0.64)

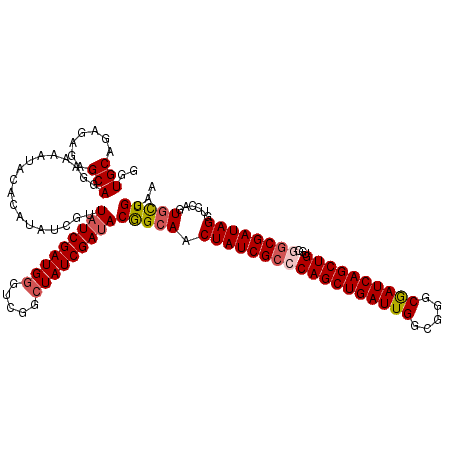
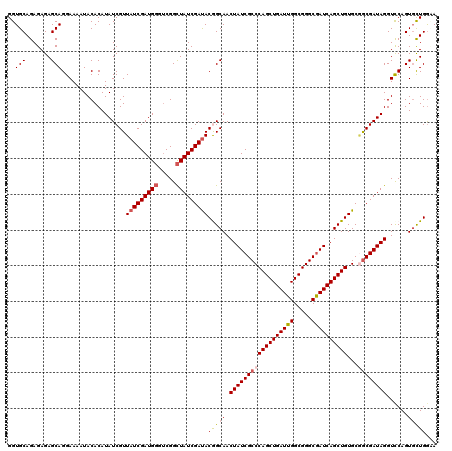
| Location | 2,885,880 – 2,886,000 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.83 |
| Mean single sequence MFE | -26.76 |
| Consensus MFE | -25.66 |
| Energy contribution | -25.26 |
| Covariance contribution | -0.40 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.32 |
| Structure conservation index | 0.96 |
| SVM decision value | 0.92 |
| SVM RNA-class probability | 0.882725 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2L_DroMel_CAF1 2885880 120 + 22407834 CAAUCAGCUGGGCGAUAGUUGCCGUAUCGAUAGCCGACCCAUCGAUAACGAUAUGUGUAUUUUCCUGCUCUCUCUGCACCAACACAAAACACGCAACUAAAUUUCGGUUAAAAACCCCUA .....(((((((...((((((((((((((((.(.....).)))))).)))...(((((.......(((.......)))...)))))......)))))))...)))))))........... ( -26.00) >DroSec_CAF1 4597 120 + 1 CAAUCAGCUGGGCGAUAGUUGCUGUAUCGAUAGCCGACCCAUCGAUAACGAUAUGUGUAUUUUCCUGCUCUCUCUGCACCAACACAAAACACGCAACUAAAUUUCGGUUAAAAACCCCAG .......((((....(((((((.((((((.((..(((....))).)).))))))((((.......(((.......)))...)))).......)))))))......(((.....))))))) ( -26.50) >DroSim_CAF1 4663 120 + 1 CAAUCAGCUGGGCGAUAGUUGCUGUAUCGAUAGCCGACCCAUCGAUAACGAUAUGUGUAUUUUCCUGCUCUCUCUGCACCAACACAAAACACGCAACUAAAUUUCGGUUAAAAACCCCAG .......((((....(((((((.((((((.((..(((....))).)).))))))((((.......(((.......)))...)))).......)))))))......(((.....))))))) ( -26.50) >DroEre_CAF1 4734 120 + 1 CAAUCAGCUGGGCGAUAGUUGCCGUAUCGAUAGCCGAGCCAUCGAUAACGAUAUGUGUAUUUUCCUGCUCUCUCUGCACCAACACAAAACACGCAACUAAAUUUCGGUUAAAAACCCCAA .....(((((((...((((((((((((((((.((...)).)))))).)))...(((((.......(((.......)))...)))))......)))))))...)))))))........... ( -28.60) >DroYak_CAF1 4891 120 + 1 CAAUCAGCUGGGCGAUAGUUGCCGUGUCGAUAACCGUGGCAUCGAUAACGAUAUGUGUAUUUUCCUGCUCUCUCUGCACCAACACAAAACACGCAACUAAAUUUCGGUUAAAAACCCCAA .....(((((((...((((((((((((((((..(....).)))))).)))...(((((.......(((.......)))...)))))......)))))))...)))))))........... ( -26.20) >consensus CAAUCAGCUGGGCGAUAGUUGCCGUAUCGAUAGCCGACCCAUCGAUAACGAUAUGUGUAUUUUCCUGCUCUCUCUGCACCAACACAAAACACGCAACUAAAUUUCGGUUAAAAACCCCAA .....(((((((...((((((((((((((((.........)))))).)))...(((((.......(((.......)))...)))))......)))))))...)))))))........... (-25.66 = -25.26 + -0.40)

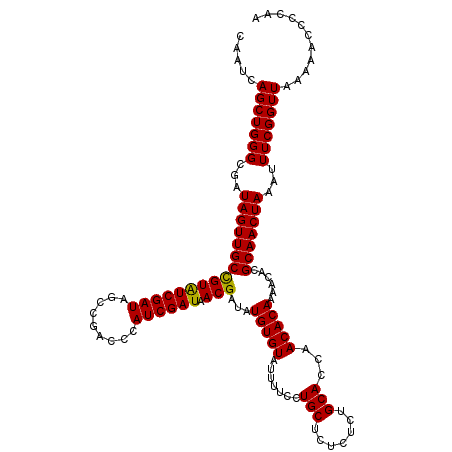
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:50:40 2006