Index >
Results for CNB 72798_0
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
72798_ENSMUSG00000043515_MOUSE_327_691/1-366 CACTTGGAGCTGGTGTACTTGGTGACGGCCTTGGTGCCCTCCGACACCGCGTGCTTGGCC
72798_ENSDARG00000022900_ZEBRAFISH_291_655/1-366 CACTTGGAGCTGGTGTATTTGGTCACGGCCTTTGTTCCCTCAGACACTGCGTGTTTGGCC
72798_SINFRUG00000141037_FUGU_315_680/1-366 TATTTGGAGCTGGTGTACTTGGTCACGGCCTTGGTGCCCTCGGACACGGCGTGCTTGGCC
72798_ENSRNOG00000018055_RAT_337_700/1-366 -ACTTGGAGCTGGTGTACTTGGTGACCGCCTTGGTGCCCTCGGACACAGCGTGCTTGGCC
72798_ENSG00000124518_HUMAN_319_684/1-366 TACTTGGAGCTGGTGTACTTGGTGACGGCCTTGGTGCCCTCCGACACCGCGTGCTTGGCC
72798_ENSMUSG00000043515_MOUSE_327_691/1-366 AGCTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGACGTGATGGTCGAG
72798_ENSDARG00000022900_ZEBRAFISH_291_655/1-366 AGTTCTCCGGGCAGGAGCAGGCGCACGGCTGTCTGGATCTCCCGGGATGTGATAGTGGAG
72798_SINFRUG00000141037_FUGU_315_680/1-366 AGCTCCCCCGGCAGCAGCAGCCTCACAGCGGTCTGGATCTCCCTGGAGGTGATGGTGGCG
72798_ENSRNOG00000018055_RAT_337_700/1-366 AGCTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGACGTGATGGTCGAG
72798_ENSG00000124518_HUMAN_319_684/1-366 AGCTCCCCCGGAAGCAGCAGGCGCACGGCGGTCTGGATCTCCCTGGAGGTGATGGTCGAG
72798_ENSMUSG00000043515_MOUSE_327_691/1-366 CGCTTGTTGTAATGCGCCAGGCGGGACGCCTCGCTCGCGATGCGCTCGAAGATGTCGTTC
72798_ENSDARG00000022900_ZEBRAFISH_291_655/1-366 CGCTTGTTGTAATGCGCCAGGCGCGACGCTTCTCCGGCGATGCGCTCGAAGATGTCGTTT
72798_SINFRUG00000141037_FUGU_315_680/1-366 CGCTTGTTGTAGTGCGCCAGGCGCGACCCTTCACTGGCGATGCGCTCGAATATGTCGTTC
72798_ENSRNOG00000018055_RAT_337_700/1-366 CGCTTGTTGTAATGCGCCAGGCGCGACGCCTCGCCCGCGATGCGCTCGAAGATGTCGTTC
72798_ENSG00000124518_HUMAN_319_684/1-366 CGCTTGTTGTAGTGCGCCAGGCGGGAAGCCTCGCTTGCGATGCGCTCGAAGATGTCGTTG
72798_ENSMUSG00000043515_MOUSE_327_691/1-366 ACGAACGAGTTCATGATGCCCATGGCCTTGGAGGAGATGCCGGTGTCGGGGTGCACTTGC
72798_ENSDARG00000022900_ZEBRAFISH_291_655/1-366 ACAAATGAGTTCATAATGCCCATCGCCTTTGAGGAGATGCCAGTGTCCGGGTGGACTTGT
72798_SINFRUG00000141037_FUGU_315_680/1-366 ACAAACGAGTTCATGATGCTCATGGCCTTGGAGGAGATGCCCGTGTCTGGGTGCACCTGC
72798_ENSRNOG00000018055_RAT_337_700/1-366 ACGAACGAGTTCATTATGCCCATGGCCTTGGAAGAGATGCCGGTGTCCGGGTGCACTTGC
72798_ENSG00000124518_HUMAN_319_684/1-366 ACGAAGGAGTTCATGATTCCCATGGCCTTAGAAGAGATGCCGGTGTCGGGGTGGACCTGC
72798_ENSMUSG00000043515_MOUSE_327_691/1-366 TTCAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTTG
72798_ENSDARG00000022900_ZEBRAFISH_291_655/1-366 TTTAGTACTTTGTACACGTAAATGGCGTAACTCTCTTTCCTGGTCTTACGCCTTTTCTTA
72798_SINFRUG00000141037_FUGU_315_680/1-366 TTCAGCACCTTGTACACGTAAATGGCGTAGCTCTCCTTCCTGTTTTTACGTCGTTTCTTA
72798_ENSRNOG00000018055_RAT_337_700/1-366 TTCAGTACCTTGTACACGTACACCGAGTAGCTCTCTTTGCGGCTGCGCTTGCGCTTCTTG
72798_ENSG00000124518_HUMAN_319_684/1-366 TTCAGCACCTTGTACACGTACACGGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTTG
72798_0.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 87.13
Mean single sequence MFE: -130.57
Consensus MFE: -105.56
Energy contribution: -107.76
Covariance contribution: 2.20
Mean z-score: -1.60
Structure conservation index: 0.81
SVM decision value: 0.34
SVM RNA-class probability: 0.694471
Prediction: RNA
######################################################################
>72798_ENSMUSG00000043515_MOUSE_327_691/1-366
CACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCCGACACCGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGACGCCUCGCUCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCGGUGUCGGGGUGCACUUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUG
.....(((((((((((((.(((((.(((....((...)).))).))))).))))...))))))))).((((.(((.(((((((((..(((((..(((((((((((((((..((..((((((((..(((((...(((..(((((...))))))))..)))))))))))))..))..))))).))..((((..((((.....))))..))))..)))))(((((((((((..(((((..(((....)))...))))).)).)))))).)))....)))..))))))))))))))))).)))) ( -142.60)
>72798_ENSDARG00000022900_ZEBRAFISH_291_655/1-366
CACUUGGAGCUGGUGUAUUUGGUCACGGCCUUUGUUCCCUCAGACACUGCGUGUUUGGCCAGUUCUCCGGGCAGGAGCAGGCGCACGGCUGUCUGGAUCUCCCGGGAUGUGAUAGUGGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCUUCUCCGGCGAUGCGCUCGAAGAUGUCGUUUACAAAUGAGUUCAUAAUGCCCAUCGCCUUUGAGGAGAUGCCAGUGUCCGGGUGGACUUGUUUUAGUACUUUGUACACGUAAAUGGCGUAACUCUCUUUCCUGGUCUUACGCCUUUUCUUA
.....((((...((((((..(((.((.......((((.(((.(((((((((((((((((((...((((.....))))(((((((.(.((((((.(.((((....)))).))))))).).)))))))......)).))))))))))..((.((((((((((((.((.....((..(((((....)))))..)).....)).))))))).....))))).))))))))).))).))))........)))))..))))))......(((((((..((.......))..)))))))..)))).. ( -113.26)
>72798_SINFRUG00000141037_FUGU_315_680/1-366
UAUUUGGAGCUGGUGUACUUGGUCACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCCGGCAGCAGCAGCCUCACAGCGGUCUGGAUCUCCCUGGAGGUGAUGGUGGCGCGCUUGUUGUAGUGCGCCAGGCGCGACCCUUCACUGGCGAUGCGCUCGAAUAUGUCGUUCACAAACGAGUUCAUGAUGCUCAUGGCCUUGGAGGAGAUGCCCGUGUCUGGGUGCACCUGCUUCAGCACCUUGUACACGUAAAUGGCGUAGCUCUCCUUCCUGUUUUUACGUCGUUUCUUA
.....(((((..((((((..((((..(((...(((((.((((((((((((((((((.(((((((((...)).)))....(((((.(((.((.........))))))))))..))))((((((((......)))))))))))))))..((((((..(((.(((.((((((((...(((((....)))))))))..)).)).))).))).))))))......))))))))))).))))).)))...).)))..))))))......(((((((..(........)...))))))))))))... ( -125.80)
>72798_ENSRNOG00000018055_RAT_337_700/1-366
ACUUGGAGCUGGUGUACUUGGUGACCGCCUUGGUGCCCUCGGACACAGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCCUCGCCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUUAUGCCCAUGGCCUUGGAAGAGAUGCCGGUGUCCGGGUGCACUUGCUUCAGUACCUUGUACACGUACACCGAGUAGCUCUCUUUGCGGCUGCGCUUGCGCUUCUUG
....(((((((((((((....(((..((...(((((.(((((((((.((((.(((..((((..((..((((((((((((((((.((((((((((..((....))..)).)))))))).)))))))))))...((((.(((((...))))).))..))((((.(((((......(((....))).))))).)))).)))))..))..))))..)))))))..))))))))).))))).)).)))((((...))))..)))))))).((((((........))))))......)))))... ( -133.60)
>72798_ENSG00000124518_HUMAN_319_684/1-366
UACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCCGACACCGCGUGCUUGGCCAGCUCCCCCGGAAGCAGCAGGCGCACGGCGGUCUGGAUCUCCCUGGAGGUGAUGGUCGAGCGCUUGUUGUAGUGCGCCAGGCGGGAAGCCUCGCUUGCGAUGCGCUCGAAGAUGUCGUUGACGAAGGAGUUCAUGAUUCCCAUGGCCUUAGAAGAGAUGCCGGUGUCGGGGUGGACCUGCUUCAGCACCUUGUACACGUACACGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUG
.....(((((((((((((.(((((.(((....((...)).))).))))).))))...)))))))))...((((((.((((((((((.((((..((((((((....(((((.((((((((((((..(((((((.(((..((((.....)))))))))))))))))))))).((.(((((.(((......))).))))))))))).)))))....))))).)))(((((((((((...(((...)))))))))).))))......((((.....)))))))).).))))))))))))))).. ( -137.60)
>consensus
CACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCCGACACCGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCCUCGCUCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCGGUGUCCGGGUGCACUUGCUUCAGCACCUUGUACACGUACACGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUUG
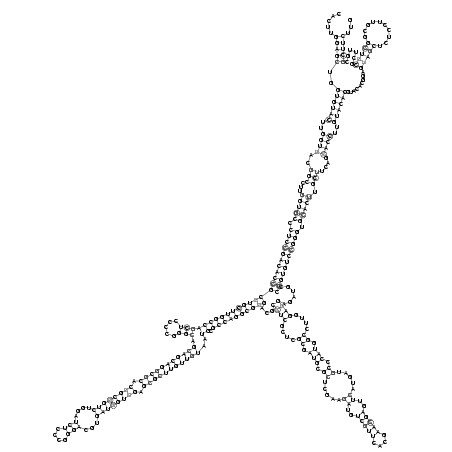
.....(((((..((((((..((((..(((...(((((.((((((((((((((((((((((((((.....))).(((((((((((.((((((((....(((....)))...)))))))).)))))))))))..)).))))))))))...((((....((.(((.((.....((..(((((....)))))..)).....)).))).))....))))......))))))))))).))))).)))....))))..)))))).........(((((..........)))))......)))))... (-105.56 = -107.76 + 2.20)
72798_0.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004