Index >
Results for CNB 72700_0
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
72700_ENSRNOG00000027856_RAT_335_700/1-367 -CACTTGGAGCTGGTGTACTTGGTGACCGCCTTGGTGCCCTCGGACACAGCGTGCTTGGC
72700_ENSG00000184825_HUMAN_291_657/1-367 TTACTTAGCGCTGGTGTACTTGGTGACGGCCTTGGTGCCCTCGGACACGGCGTGCTTGGC
72700_ENSMUSG00000054813_MOUSE_326_691/1-367 -CACTTGGAGCTGGTGTACTTGGTGACAGCCTTGGTGCCCTCCGACACCGCGTGCTTGGC
72700_ENSDARG00000022900_ZEBRAFISH_290_655/1-367 TCACTTGGAGCTGGTGTATTTGGTCACGGCCTTTGTTCCCTCAGACACTGCGTGTTTGGC
72700_SINFRUG00000141037_FUGU_314_680/1-367 CTATTTGGAGCTGGTGTACTTGGTCACGGCCTTGGTGCCCTCGGACACGGCGTGCTTGGC
72700_ENSRNOG00000027856_RAT_335_700/1-367 CAGCTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGACGTGATGGTCGA
72700_ENSG00000184825_HUMAN_291_657/1-367 CAACTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCTGGAGGTGATGGTCGA
72700_ENSMUSG00000054813_MOUSE_326_691/1-367 CAGCTCCCCGGGCAGCAGCAGGCGCACGGCCGTCTGGATCTCCCGGGACGTGATGGTCGA
72700_ENSDARG00000022900_ZEBRAFISH_290_655/1-367 CAGTTCTCCGGGCAGGAGCAGGCGCACGGCTGTCTGGATCTCCCGGGATGTGATAGTGGA
72700_SINFRUG00000141037_FUGU_314_680/1-367 CAGCTCCCCCGGCAGCAGCAGCCTCACAGCGGTCTGGATCTCCCTGGAGGTGATGGTGGC
72700_ENSRNOG00000027856_RAT_335_700/1-367 GCGCTTGTTGTAATGCGCTAGACGCGACGCCTCGCCCGCGATGCGCTCGAAGATGTCGTT
72700_ENSG00000184825_HUMAN_291_657/1-367 GCGCTTGTTGTAATGCGCCAGGCGGGAAGCCTCACCCGCGATGCGTTCGAAGATGTCGTT
72700_ENSMUSG00000054813_MOUSE_326_691/1-367 GCGCTTGTTGTAATGCGCCAGGCGGGACGCCTCGCTCGCGATGCGCTCGAAGATGTCGTT
72700_ENSDARG00000022900_ZEBRAFISH_290_655/1-367 GCGCTTGTTGTAATGCGCCAGGCGCGACGCTTCTCCGGCGATGCGCTCGAAGATGTCGTT
72700_SINFRUG00000141037_FUGU_314_680/1-367 GCGCTTGTTGTAGTGCGCCAGGCGCGACCCTTCACTGGCGATGCGCTCGAATATGTCGTT
72700_ENSRNOG00000027856_RAT_335_700/1-367 CACGAACGAGTTCATGATGCCCATGGCCTTAGAAGAGATGCCCGTGTCCGGGTGCACTTG
72700_ENSG00000184825_HUMAN_291_657/1-367 GACGAAGGAGTTCATGATTCCCATGGCCTTAGAGGAGATGCCGGTGTCGGGGTGGACCTG
72700_ENSMUSG00000054813_MOUSE_326_691/1-367 CACGAACGAGTTCATGATGCCCATGGCCTTGGAGGAGATGCCGGTGTCGGGGTGCACTTG
72700_ENSDARG00000022900_ZEBRAFISH_290_655/1-367 TACAAATGAGTTCATAATGCCCATCGCCTTTGAGGAGATGCCAGTGTCCGGGTGGACTTG
72700_SINFRUG00000141037_FUGU_314_680/1-367 CACAAACGAGTTCATGATGCTCATGGCCTTGGAGGAGATGCCCGTGTCTGGGTGCACCTG
72700_ENSRNOG00000027856_RAT_335_700/1-367 CTTCAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTT
72700_ENSG00000184825_HUMAN_291_657/1-367 CTTCAGCACCTTGTACACGTATACGGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTT
72700_ENSMUSG00000054813_MOUSE_326_691/1-367 CTTCAGCACCTTGTACACGTACACCGAGTAGCTCTCCTTGCGGCTGCGCTTGCGCTTCTT
72700_ENSDARG00000022900_ZEBRAFISH_290_655/1-367 TTTTAGTACTTTGTACACGTAAATGGCGTAACTCTCTTTCCTGGTCTTACGCCTTTTCTT
72700_SINFRUG00000141037_FUGU_314_680/1-367 CTTCAGCACCTTGTACACGTAAATGGCGTAGCTCTCCTTCCTGTTTTTACGTCGTTTCTT
72700_0.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 86.76
Mean single sequence MFE: -129.07
Consensus MFE: -102.52
Energy contribution: -105.32
Covariance contribution: 2.80
Mean z-score: -1.57
Structure conservation index: 0.79
SVM decision value: 0.20
SVM RNA-class probability: 0.630234
Prediction: RNA
######################################################################
>72700_ENSRNOG00000027856_RAT_335_700/1-367
CACUUGGAGCUGGUGUACUUGGUGACCGCCUUGGUGCCCUCGGACACAGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCUAGACGCGACGCCUCGCCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUAGAAGAGAUGCCCGUGUCCGGGUGCACUUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUU
.....(((((..((((((..((((...((...(((((.(((((((((.((((.((((((((((..(.(((((.(((((((((((.((((((((((..((....))..)).)))))))).)))))))))))..((((.....)))).......))))).)..))(((((..((..(((((....)))))..)).))).))...)))))....)))..))))..))))))))).))))).)).....))))..))))))(((......((((((........))))))...)))))))).. ( -129.30)
>72700_ENSG00000184825_HUMAN_291_657/1-367
UUACUUAGCGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAACUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCUGGAGGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGAAGCCUCACCCGCGAUGCGUUCGAAGAUGUCGUUGACGAAGGAGUUCAUGAUUCCCAUGGCCUUAGAGGAGAUGCCGGUGUCGGGGUGGACCUGCUUCAGCACCUUGUACACGUAUACGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUU
......(((((..((((((..((((..(((...(((.(((((.((((((((((((((((........)))))).(((((((((((.((((((((...(((((....))))))))))))).)))))))))))...))))).((((.....((((...(((((((((((....)))).))))).))...(((..((((.....))))..))).))))...)))).))))).))).))))).)))....))))..))))))......((.((((((........)))))).)).))))).... ( -135.70)
>72700_ENSMUSG00000054813_MOUSE_326_691/1-367
CACUUGGAGCUGGUGUACUUGGUGACAGCCUUGGUGCCCUCCGACACCGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGGGACGCCUCGCUCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUGGAGGAGAUGCCGGUGUCGGGGUGCACUUGCUUCAGCACCUUGUACACGUACACCGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUU
.....(((((..((((((..((((..(((...(((((.(((((((((((....)..(((...((((((((((((((((((((((.((((((((((..((....))..)).)))))))).)))))))))))..)))((((((((((.(((.(((....))).))))))...((..(((((....)))))..)).....)))..)))).)))).))))..))))))))))))).))))).)))....))))..))))))(((......((((((........))))))...)))))))).. ( -141.30)
>72700_ENSDARG00000022900_ZEBRAFISH_290_655/1-367
UCACUUGGAGCUGGUGUAUUUGGUCACGGCCUUUGUUCCCUCAGACACUGCGUGUUUGGCCAGUUCUCCGGGCAGGAGCAGGCGCACGGCUGUCUGGAUCUCCCGGGAUGUGAUAGUGGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCUUCUCCGGCGAUGCGCUCGAAGAUGUCGUUUACAAAUGAGUUCAUAAUGCCCAUCGCCUUUGAGGAGAUGCCAGUGUCCGGGUGGACUUGUUUUAGUACUUUGUACACGUAAAUGGCGUAACUCUCUUUCCUGGUCUUACGCCUUUUCUU
......((((...((((((..(((.((.......((((.(((.(((((((((((((((((((...((((.....))))(((((((.(.((((((.(.((((....)))).))))))).).)))))))......)).))))))))))..((.((((((((((((.((.....((..(((((....)))))..)).....)).))))))).....))))).))))))))).))).))))........)))))..))))))......(((((((..((.......))..)))))))..)))). ( -113.26)
>72700_SINFRUG00000141037_FUGU_314_680/1-367
CUAUUUGGAGCUGGUGUACUUGGUCACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCCGGCAGCAGCAGCCUCACAGCGGUCUGGAUCUCCCUGGAGGUGAUGGUGGCGCGCUUGUUGUAGUGCGCCAGGCGCGACCCUUCACUGGCGAUGCGCUCGAAUAUGUCGUUCACAAACGAGUUCAUGAUGCUCAUGGCCUUGGAGGAGAUGCCCGUGUCUGGGUGCACCUGCUUCAGCACCUUGUACACGUAAAUGGCGUAGCUCUCCUUCCUGUUUUUACGUCGUUUCUU
......(((((..((((((..((((..(((...(((((.((((((((((((((((((.(((((((((...)).)))....(((((.(((.((.........))))))))))..))))((((((((......)))))))))))))))..((((((..(((.(((.((((((((...(((((....)))))))))..)).)).))).))).))))))......))))))))))).))))).)))...).)))..))))))......(((((((..(........)...)))))))))))).. ( -125.80)
>consensus
_CACUUGGAGCUGGUGUACUUGGUGACGGCCUUGGUGCCCUCGGACACGGCGUGCUUGGCCAGCUCCCCGGGCAGCAGCAGGCGCACGGCCGUCUGGAUCUCCCGGGACGUGAUGGUCGAGCGCUUGUUGUAAUGCGCCAGGCGCGACGCCUCACCCGCGAUGCGCUCGAAGAUGUCGUUCACGAACGAGUUCAUGAUGCCCAUGGCCUUAGAGGAGAUGCCCGUGUCCGGGUGCACUUGCUUCAGCACCUUGUACACGUAAACGGAGUAGCUCUCCUUGCGGCUGCGCUUGCGCUUCUU
......(((((..((((((..((((..(((...(((((.((((((((((((((((((((((((((.....))).(((((((((((.((((((((....(((....)))...)))))))).)))))))))))..)).))))))))))...((((....((.(((.((.....((..(((((....)))))..)).....)).))).))....))))......))))))))))).))))).)))....))))..)))))).........(((((..........)))))......))))).. (-102.52 = -105.32 + 2.80)
72700_0.rnaz
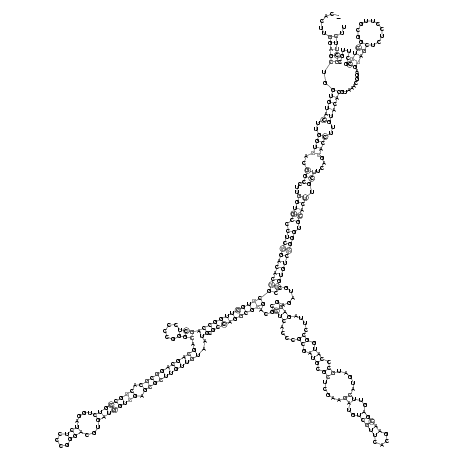
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004