Index >
Results for CNB 501424_6-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
501424_ENSDARG00000025235_ZEBRAFISH_368_1002/1-684 ACCAC--TACTACTTGCACTGATGCTGCTCTGGCTCCTTGGACGAAGCATCCAGGGGTCC
501424_ENSMUSG00000039825_MOUSE_135_716/1-684 CAAGC--TACTACCCTCGCTAACAATGCTCTGGCTTCTGGACCTGGGGAGCCAGGGTTCC
501424_ENSRNOG00000011386_RAT_135_730/1-684 CAAGC--TACTACCCTCGCTAACAATGCTCTGGCTTCTGGACCTGGGGAGCCAGGGTTCC
501424_SINFRUG00000136522_FUGU_3941_4616/1-684 ACCACCGAGCTCCCTGCACTGATGCTGCTCTGACTGCGAGGCCTTAAGAGCCAGGGGTCA
501424_ENSG00000135540_HUMAN_9405_10000/1-684 CATGC--TGCTGCCAGCACTAACTGTGCTCTGGCTCCGGGAGCGGGGCAGCCAGGGCTCT
* ** * * ** * ******* ** * * * * ****** **
501424_ENSDARG00000025235_ZEBRAFISH_368_1002/1-684 TCATAGTCACTACAAGGGCTATGAGATGGTGACGTGGAGGAT---CCTTTGCCTTT----
501424_ENSMUSG00000039825_MOUSE_135_716/1-684 TCAAAGTCACTGCAGGGGCTCTGGGAAGG---GGAGCTGCCG---CCCTTGCCAGG----
501424_ENSRNOG00000011386_RAT_135_730/1-684 TCACAGTCACTGCAGGGGCTCTGGGAAGG---GGAACCGCTG---CCTTGGCCAGG----
501424_SINFRUG00000136522_FUGU_3941_4616/1-684 TCATAGTCGCTGCTGGGGCTGTGAGACGGCGAGGAAGGCGATGCCCCTTTTCCTTTCCCT
501424_ENSG00000135540_HUMAN_9405_10000/1-684 TCCAAGTCACTGCAGGGGCTCTGGGAGGG---CGAGCTGCCA---CTCTTGCCTGG----
** **** ** * ***** ** ** ** * * * **
501424_ENSDARG00000025235_ZEBRAFISH_368_1002/1-684 -----AAGCCCATTCTGAAGTGACTGTTGGAGAGTGGCAATCAATGATTCATTGAGAAGA
501424_ENSMUSG00000039825_MOUSE_135_716/1-684 -----GAGGCTTAGCTGCAGTGAATGCTGGAGCGAGGCGATCAGGCTCTCATT-----GA
501424_ENSRNOG00000011386_RAT_135_730/1-684 -----GAGGCTTAGCTGCAGAGAATGCTGGAGTGAGGCGATCAGGCTCTCGTT-----GA
501424_SINFRUG00000136522_FUGU_3941_4616/1-684 CCCCCCAGACCCATCTGTAGACTCTGCTGCAGGCTGGCAATGAGCGTCTCACTGAGCATG
501424_ENSG00000135540_HUMAN_9405_10000/1-684 -----GAGGCTCAGCTGCAGCGAGTGCTGGAGTGTGGCGATCAGGCTCTCGTT-----GA
** * *** ** ** ** ** *** ** * ** *
501424_ENSDARG00000025235_ZEBRAFISH_368_1002/1-684 GAACCATT--CCT-AATATTTGACTGCCCAATATTGGCA-GAGGGA-GAGC-TGTTTTTC
501424_ENSMUSG00000039825_MOUSE_135_716/1-684 GCAC-------------------CTGACC---GTT-TGT-CTG------GT-TGTTCTTT
501424_ENSRNOG00000011386_RAT_135_730/1-684 GCAC-------------------CTGCCC---ATT-TGT-CTG------GT-TGTTCTTC
501424_SINFRUG00000136522_FUGU_3941_4616/1-684 GCTCCATTTGCATCGTTAGCTCCATTCACCGTGCTGACACCTCCGACGGGCTTTTTCGGG
501424_ENSG00000135540_HUMAN_9405_10000/1-684 GCAC-------------------CTGCCC---GTT-GCA-CTG------GC-TGCTCTTC
* * * * * * * *
501424_ENSDARG00000025235_ZEBRAFISH_368_1002/1-684 CCAGGCTTTCTCCGTAGAGAGTCTGTGCGTGCAGGTGGAAGGGGTGGTTTCTTAGGCTTG
501424_ENSMUSG00000039825_MOUSE_135_716/1-684 TTGGGAATCCTTCGCAGAGAATCGGTCCGGGATGGTGGCAGGGGTGGTTTCTTTGCTTTC
501424_ENSRNOG00000011386_RAT_135_730/1-684 TTGGGAATCCTTCGCAGAGAATCTGTCCGGGATGGTGGCGGTGGTGGTTTTTTTGCTTTC
501424_SINFRUG00000136522_FUGU_3941_4616/1-684 CCTGGCTTGCGTCTGAGAGAGTCTGTTCGAGCCGGAGGCAGCGGTGGCTTCTTAGACTTG
501424_ENSG00000135540_HUMAN_9405_10000/1-684 TTGGGAATCCTGCGGAGGGAGTCTGTCCGGGAGGGTGGCAGGGGAGGCTTCTTTGCTTTC
** * * * ** ** ** ** ** * ** ** * ** ** ** ** * **
501424_6-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 63.14
Mean single sequence MFE: -114.56
Consensus MFE: -41.91
Energy contribution: -43.55
Covariance contribution: 1.64
Mean z-score: -2.35
Structure conservation index: 0.37
SVM decision value: 1.15
SVM RNA-class probability: 0.922539
Prediction: RNA
######################################################################
>501424_ENSDARG00000025235_ZEBRAFISH_368_1002/1-684
ACCACUACUACUUGCACUGAUGCUGCUCUGGCUCCUUGGACGAAGCAUCCAGGGGUCCUCAUAGUCACUACAAGGGCUAUGAGAUGGUGACGUGGAGGAUCCUUUGCCUUUAAGCCCAUUCUGAAGUGACUGUUGGAGAGUGGCAAUCAAUGAUUCAUUGAGAAGAGAACCAUUCCUAAUAUUUGACUGCCCAAUAUUGGCAGAGGGAGAGCUGUUUUUCCCAGGCUUUCUCCGUAGAGAGUCUGUGCGUGCAGGUGGAAGGGGUGGUUUCUUAGGCUUG
((((((.((((((((((((((((..(((((((.(((((((.......))))))))))((.(((((((((.((.(((((..(((..(....)(..(((....)))..))))..)))))....)).))))))))).))))))..)).))))..(((((.(((.(.(((((.((...............(((((.......))))).(((((((....))))))).)).))))).).))).))))).....))))))))))....))))))............ ( -108.80)
>501424_ENSMUSG00000039825_MOUSE_135_716/1-684
CAAGCUACUACCCUCGCUAACAAUGCUCUGGCUUCUGGACCUGGGGAGCCAGGGUUCCUCAAAGUCACUGCAGGGGCUCUGGGAAGGGGAGCUGCCGCCCUUGCCAGGGAGGCUUAGCUGCAGUGAAUGCUGGAGCGAGGCGAUCAGGCUCUCAUUGAGCACCUGACCGUUUGUCUGGUUGUUCUUUUUGGGAAUCCUUCGCAGAGAAUCGGUCCGGGAUGGUGGCAGGGGUGGUUUCUUUGCUUUC
.((((.(((((((..((((.((..((....))(((((((((((((((((....))))))))...((.((((((((..(((.(((((((((((((((.((((....)))).)))...(((.((((....)))).)))..........)))))))...(((((((.(((.....))).)).)))))))))).)))..)))).)))).))...))))))))))).))))..)))))))......)))).. ( -110.30)
>501424_ENSRNOG00000011386_RAT_135_730/1-684
CAAGCUACUACCCUCGCUAACAAUGCUCUGGCUUCUGGACCUGGGGAGCCAGGGUUCCUCACAGUCACUGCAGGGGCUCUGGGAAGGGGAACCGCUGCCUUGGCCAGGGAGGCUUAGCUGCAGAGAAUGCUGGAGUGAGGCGAUCAGGCUCUCGUUGAGCACCUGCCCAUUUGUCUGGUUGUUCUUCUUGGGAAUCCUUCGCAGAGAAUCUGUCCGGGAUGGUGGCGGUGGUGGUUUUUUUGCUUUC
.(((((((((((.((((((.....((((((((((((.......))))))))))))((((.((((((.((((..(((.((..(((((((.(((((......(((.((((((.((((.(((.(((......))).))).))))..))..((((.....)))).)))).)))......))))).)))))))..))...)))..)))).))..))))..)))))))))).))))))))))).......... ( -106.20)
>501424_SINFRUG00000136522_FUGU_3941_4616/1-684
ACCACCGAGCUCCCUGCACUGAUGCUGCUCUGACUGCGAGGCCUUAAGAGCCAGGGGUCAUCAUAGUCGCUGCUGGGGCUGUGAGACGGCGAGGAAGGCGAUGCCCCUUUUCCUUUCCCUCCCCCCAGACCCAUCUGUAGACUCUGCUGCAGGCUGGCAAUGAGCGUCUCACUGAGCAUGGCUCCAUUUGCAUCGUUAGCUCCAUUCACCGUGCUGACACCUCCGACGGGCUUUUUCGGGCCUGGCUUGCGUCUGAGAGAGUCUGUUCGAGCCGGAGGCAGCGGUGGCUUCUUAGACUUG
.((((((.((..((.((...(((((.((((.....(.(((((((((...((((((((((.((((((((.(....).))))))))((.((.(((((((((...)))....)))))).)).))......))))).(((((((......))))))))))))..)))).))))).).))))(((....)))..)))))((((((.(........).))))))...((.((((((((((((((((((......).))))))))))))))))).)))).))..))..))))))............. ( -125.60)
>501424_ENSG00000135540_HUMAN_9405_10000/1-684
CAUGCUGCUGCCAGCACUAACUGUGCUCUGGCUCCGGGAGCGGGGCAGCCAGGGCUCUUCCAAGUCACUGCAGGGGCUCUGGGAGGGCGAGCUGCCACUCUUGCCUGGGAGGCUCAGCUGCAGCGAGUGCUGGAGUGUGGCGAUCAGGCUCUCGUUGAGCACCUGCCCGUUGCACUGGCUGCUCUUCUUGGGAAUCCUGCGGAGGGAGUCUGUCCGGGAGGGUGGCAGGGGAGGCUUCUUUGCUUUC
....(((..((((((((.....))))..))))..)))((((((((.((((...(((((((((((((........)))).)))))))))...((((((((((..((((((((((((..((((((.(((((((..((((..(((..(((((((.....))))..)))..)))..))))))).))))(((....)))..))))))...)))))).))))))))))))))))....)))).)))))))).. ( -121.90)
>consensus
CAAGC__UACUACCCGCACUAACAAUGCUCUGGCUCCUGGACCUGGGGAGCCAGGGGUCCUCAAAGUCACUGCAGGGGCUCUGGGAAGG___GGAGCUGCAG___CCUUUGCCUGG_________GAGGCUCAGCUGCAGAGAAUGCUGGAGUGUGGCGAUCAGGCUCUCAUU_____GAGCAC___________________CUGCCC___GUU_GCA_CUG______GC_UGUUCUUCUUGGGAAUCCUUCGCAGAGAGUCUGUCCGGGAUGGUGGCAGGGGUGGUUUCUUUGCUUUC
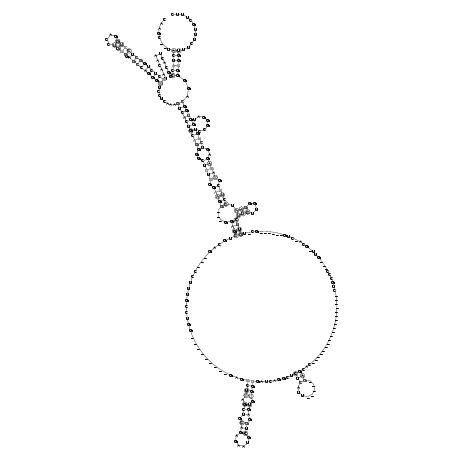
........((((((............(((((((((((((....)))).)))))))))........(((((((((((..(((((.(((((....((((...............................((((.(((.(((......))).))).)))).........(((........)))....................................................))))(((....))).))))).)))))..))))).......))))))...))))))............ (-41.91 = -43.55 + 1.64)
501424_6-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004