Index >
Results for CNB 467058_3-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
467058_ENSRNOG00000020045_RAT_7890_8393/1-507 GTTTGATTTTGTTCTCAGCAAGGGGTCCCTGAATTTATGTTCGCTTCTCCCTGCAAATGA
467058_ENSG00000165606_HUMAN_8456_8959/1-507 GTTTGATTTTGTTCTCAGCAAGGGGTCCCTGAATTTATGTTCGCTTCTCGATGCAAATGA
467058_SINFRUG00000142795_FUGU_775_1264/1-507 TTTTGATTTTGTTCACAGCAAGGGGTCCCGCGATTTA-TTTCGCTTCCTCAGATAAATGA
467058_ENSDARG00000011919_ZEBRAFISH_2243_2727/1-507 TTTTGATTTTGTTCACAGCAAGGGGTCCGAGGATTTATTTCCCACTCTCG-CGTAAATGA
467058_ENSMUSG00000041730_MOUSE_457_960/1-507 GTTTGATTTTGTTCTCAGCAAGGGGTCCCTGAATTTATGTTCGCTTCTCCCTGCAAATGA
************* ************* ***** * * ** ******
467058_ENSRNOG00000020045_RAT_7890_8393/1-507 TATGCAGTGGGACCCTTCGCAATTATTTTTAAAATGCATCCGAGGCAGACAC-TCAGCGG
467058_ENSG00000165606_HUMAN_8456_8959/1-507 TATGCGGTGGGACCCTTTGCAATTACTTTTAAAATGCATCCGAGGCAGACAC-TCAACGG
467058_SINFRUG00000142795_FUGU_775_1264/1-507 TCTGCTGCGGGACCCTTT-CAATTAGTTTTAAAGCGCA--TCAGG--G--ACGTCAGAGC
467058_ENSDARG00000011919_ZEBRAFISH_2243_2727/1-507 TATGCAGCGGGACCCTTT-CAATTACTTTTAAAACGCA--CGCGG--G--AC-GCCGCGC
467058_ENSMUSG00000041730_MOUSE_457_960/1-507 TATGCAGTGGGACCCTTCGCAATTACTTTTAAAATGCATCCGAGGCAGACAC-TCAGCGG
* *** * ********* ****** ******* *** ** * ** * *
467058_ENSRNOG00000020045_RAT_7890_8393/1-507 AGAGAGACCGCCCTTCTCTCTGCCTG-GGTGAAAATTAAACTCTAAAGTACCGGGTTGAA
467058_ENSG00000165606_HUMAN_8456_8959/1-507 AGAGAGACCGCCCTTCTCTCTGCCTG-GGTGAAAATTAAACTCTAAAGTACCGGGCTGAA
467058_SINFRUG00000142795_FUGU_775_1264/1-507 GCAGACGCCATGCGTCTCTGAG-GTGTAGGCAAAATGAGGCTT-CGAGAAGC-AACTGAA
467058_ENSDARG00000011919_ZEBRAFISH_2243_2727/1-507 GCAGACACCGCCGCTC-CTCTG-CT--GCTGGAAATGAGGCTC-CGAGTAGC-AGCTGAA
467058_ENSMUSG00000041730_MOUSE_457_960/1-507 AGAGAGACCGCCCTTCTCTCTGCCTG-GGTGAAAATTAAACTCTAAAGTACCGGGTTGAA
*** ** ** ** * * **** * ** ** * * ****
467058_ENSRNOG00000020045_RAT_7890_8393/1-507 ATTTTCAAGTCAGGGGCGCGGGATTGGATCAAATCACATAAACTGCAAAAAAAGCAAGTC
467058_ENSG00000165606_HUMAN_8456_8959/1-507 ATTTTCAAGTCAGGGGCGCGGGATTGGATCAAATCACATAAACTGCAAAAAAAGCAAGTC
467058_SINFRUG00000142795_FUGU_775_1264/1-507 ATTTAGAAGA--GGAGTGCGGGATTGGACTAAACCACATAAAGTGC-AGAAAATCCCTTC
467058_ENSDARG00000011919_ZEBRAFISH_2243_2727/1-507 ATTTTGAAGT--GCGGGACGGTATTGGATGAAATCACATAAACTGC-AAAAAAGCAAGTG
467058_ENSMUSG00000041730_MOUSE_457_960/1-507 ATTTTCAAGTCAGGGGCGCGGGATTGGATCAAATCACATAAACTGCAAAAAAAGCAAGTC
**** *** * * *** ****** *** ******** *** * **** * *
467058_ENSRNOG00000020045_RAT_7890_8393/1-507 TAATGGCGCATAATTGGTTCTGTAATAGCATTACACCAGGATAATAGCCCGTTACGGCCC
467058_ENSG00000165606_HUMAN_8456_8959/1-507 TAATGGCGCATAATTGGTTCTGTAATAGCATTACACCAGGATAATAGCCCGTTACGGCCC
467058_SINFRUG00000142795_FUGU_775_1264/1-507 TAATGGCGCGTAATTGGTTCTGTAATATCATTACAGGCGGATAATGGCCAGTTACAGGGC
467058_ENSDARG00000011919_ZEBRAFISH_2243_2727/1-507 TAATGGTGCATAATTGGTTCTCTAATAGCATTACACAAGGATAATAGCCTGTTATGGCCC
467058_ENSMUSG00000041730_MOUSE_457_960/1-507 TAATGGCGCATAATTGGTTCTGTAATAGCATTACACCAGGATAATAGCCCGTTACGGCCC
****** ** *********** ***** ******* ******* *** **** * *
467058_3-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 300
Mean pairwise identity: 81.82
Mean single sequence MFE: -92.94
Consensus MFE: -48.87
Energy contribution: -51.88
Covariance contribution: 3.01
Mean z-score: -2.06
Structure conservation index: 0.53
SVM decision value: 0.07
SVM RNA-class probability: 0.570399
Prediction: RNA
######################################################################
>467058_ENSRNOG00000020045_RAT_7890_8393/1-507
GUUUGAUUUUGUUCUCAGCAAGGGGUCCCUGAAUUUAUGUUCGCUUCUCCCUGCAAAUGAUAUGCAGUGGGACCCUUCGCAAUUAUUUUUAAAAUGCAUCCGAGGCAGACACUCAGCGGAGAGAGACCGCCCUUCUCUCUGCCUGGGUGAAAAUUAAACUCUAAAGUACCGGGUUGAAAUUUUCAAGUCAGGGGCGCGGGAUUGGAUCAAAUCACAUAAACUGCAAAAAAAGCAAGUCUAAUGGCGCAUAAUUGGUUCUGUAAUAGCAUUACACCAGGAUAAUAGCCCGUUACGGCCC
(((((((((...((((.((.(((((((((.((((....))))........(((((.......))))).))))))))).))...............((..((..(((....(((((((((((((((.......))))))))).))))))(((((((.(((((.........)))))..)))))))..)))..))..))))))..))))))))).........(((.......))).(((((((((.((....(((((...(((((...)))))))))).......)))))))).))).. ( -92.80)
>467058_ENSG00000165606_HUMAN_8456_8959/1-507
GUUUGAUUUUGUUCUCAGCAAGGGGUCCCUGAAUUUAUGUUCGCUUCUCGAUGCAAAUGAUAUGCGGUGGGACCCUUUGCAAUUACUUUUAAAAUGCAUCCGAGGCAGACACUCAACGGAGAGAGACCGCCCUUCUCUCUGCCUGGGUGAAAAUUAAACUCUAAAGUACCGGGCUGAAAUUUUCAAGUCAGGGGCGCGGGAUUGGAUCAAAUCACAUAAACUGCAAAAAAAGCAAGUCUAAUGGCGCAUAAUUGGUUCUGUAAUAGCAUUACACCAGGAUAAUAGCCCGUUACGGCCC
((((((((((.......((((((((((((.((((....)))).........((((.......))))..))))))))))))................(((((.(((((((..(((....))).((((.......))))))))))))))))))))))))))......(((.((((((((.....))..(((..((((((..((((......)))).(((..(((((.......)).)))...))))))).......((..(((....)))..)).))..)))...)))))).)))..... ( -93.20)
>467058_SINFRUG00000142795_FUGU_775_1264/1-507
UUUUGAUUUUGUUCACAGCAAGGGGUCCCGCGAUUUAUUUCGCUUCCUCAGAUAAAUGAUCUGCUGCGGGACCCUUUCAAUUAGUUUUAAAGCGCAUCAGGGACGUCAGAGCGCAGACGCCAUGCGUCUCUGAGGUGUAGGCAAAAUGAGGCUUCGAGAAGCAACUGAAAUUUAGAAGAGGAGUGCGGGAUUGGACUAAACCACAUAAAGUGCAGAAAAUCCCUUCUAAUGGCGCGUAAUUGGUUCUGUAAUAUCAUUACAGGCGGAUAAUGGCCAGUUACAGGGC
..((.(((((((..(((.(((((((((((((((......)).......(((((.....)))))..))))))))))))...................(((((((((.((..(((....)))..)))))))))))).)))..))))))).))((((....))))..(((....(((((((.((((..(...(((((......))).))...)..)......))))))))))((((.(((.(((.((.(((((((...))))))))).))).)))))))....)))... ( -97.90)
>467058_ENSDARG00000011919_ZEBRAFISH_2243_2727/1-507
UUUUGAUUUUGUUCACAGCAAGGGGUCCGAGGAUUUAUUUCCCACUCUCGCGUAAAUGAUAUGCAGCGGGACCCUUUCAAUUACUUUUAAAACGCACGCGGGACGCCGCGCGCAGACACCGCCGCUCCUCUGCUGCUGGAAAUGAGGCUCCGAGUAGCAGCUGAAAUUUUGAAGUGCGGGACGGUAUUGGAUGAAAUCACAUAAACUGCAAAAAAGCAAGUGUAAUGGUGCAUAAUUGGUUCUCUAAUAGCAUUACACAAGGAUAAUAGCCUGUUAUGGCCC
...((((((..((((..(.((((((((((.(((......)))).(.((.(((((.....))))))).)))))))))))...............((.(((((....))))).))....((((((((((..(((((((((((........))).))))))))..))...........))))..))))..))))..)))))).......(((......))).((((((((.(.....(((((....)))))).))))))))..........(((......))).. ( -88.00)
>467058_ENSMUSG00000041730_MOUSE_457_960/1-507
GUUUGAUUUUGUUCUCAGCAAGGGGUCCCUGAAUUUAUGUUCGCUUCUCCCUGCAAAUGAUAUGCAGUGGGACCCUUCGCAAUUACUUUUAAAAUGCAUCCGAGGCAGACACUCAGCGGAGAGAGACCGCCCUUCUCUCUGCCUGGGUGAAAAUUAAACUCUAAAGUACCGGGUUGAAAUUUUCAAGUCAGGGGCGCGGGAUUGGAUCAAAUCACAUAAACUGCAAAAAAAGCAAGUCUAAUGGCGCAUAAUUGGUUCUGUAAUAGCAUUACACCAGGAUAAUAGCCCGUUACGGCCC
(((((((((...((((.((.(((((((((.((((....))))........(((((.......))))).))))))))).))...............((..((..(((....(((((((((((((((.......))))))))).))))))(((((((.(((((.........)))))..)))))))..)))..))..))))))..))))))))).........(((.......))).(((((((((.((....(((((...(((((...)))))))))).......)))))))).))).. ( -92.80)
>consensus
GUUUGAUUUUGUUCUCAGCAAGGGGUCCCUGAAUUUAUGUUCGCUUCUCCAUGCAAAUGAUAUGCAGUGGGACCCUUUGCAAUUACUUUUAAAAUGCAUCCGAGGCAGACAC_UCAGCGGAGAGAGACCGCCCUUCUCUCUGCCUG_GGUGAAAAUUAAACUCUAAAGUACCGGGCUGAAAUUUUCAAGUCAGGGGCGCGGGAUUGGAUCAAAUCACAUAAACUGCAAAAAAAGCAAGUCUAAUGGCGCAUAAUUGGUUCUGUAAUAGCAUUACACCAGGAUAAUAGCCCGUUACGGCCC
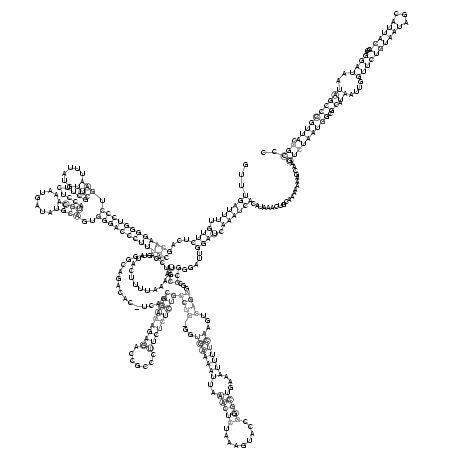
...((((((.((((...((((((((((((.(((......))).........((((.......))))..))))))))))))..............(((....................(((((((((.......)))))))))((((...(((((((..(((((.........)))))...)))))))...))))...))).....)))).)))))).....................(((((((((.((.((....((((((((((...))))))...))))..)))))))))).))).. (-48.87 = -51.88 + 3.01)
467058_3-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004