Index >
Results for CNB 467045_3-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
467045_ENSMUSG00000041730_MOUSE_457_903/1-449 -TTTGATTTTGTTCTCAGCAAGGGGTCCCTGAATTTATGTTCGCTTCTCCCTGCAAATGA
467045_ENSG00000165606_HUMAN_8456_8903/1-449 GTTTGATTTTGTTCTCAGCAAGGGGTCCCTGAATTTATGTTCGCTTCTCGATGCAAATGA
467045_ENSRNOG00000020045_RAT_7890_8336/1-449 -TTTGATTTTGTTCTCAGCAAGGGGTCCCTGAATTTATGTTCGCTTCTCCCTGCAAATGA
467045_SINFRUG00000142795_FUGU_773_1208/1-449 -TTTGATTTTGTTCACAGCAAGGGGTCCCGCGATTTAT-TTCGCTTCCTCAGATAAATGA
467045_ENSDARG00000011919_ZEBRAFISH_2244_2674/1-449 TTTTGATTTTGTTCACAGCAAGGGGTCCGAGGATTTAT-TTCCCACTCTCGCGTAAATGA
************* ************* ****** *** * ******
467045_ENSMUSG00000041730_MOUSE_457_903/1-449 TATGCAGTGGGACCCTTCGCAATTACTTTTAAAATGCATCCGAGGCAGACACTCAGCGGA
467045_ENSG00000165606_HUMAN_8456_8903/1-449 TATGCGGTGGGACCCTTTGCAATTACTTTTAAAATGCATCCGAGGCAGACACTCAACGGA
467045_ENSRNOG00000020045_RAT_7890_8336/1-449 TATGCAGTGGGACCCTTCGCAATTATTTTTAAAATGCATCCGAGGCAGACACTCAGCGGA
467045_SINFRUG00000142795_FUGU_773_1208/1-449 TCTGCTGCGGGACCCTTT-CAATTAGTTTTAAAGCGCATC--AGG--GAC-GTCAGAGCG
467045_ENSDARG00000011919_ZEBRAFISH_2244_2674/1-449 TATGCAGCGGGACCCTTT-CAATTACTTTTAAAACGCACG--CGG--GAC-G-CCGCGCG
* *** * ********* ****** ******* *** ** *** * *
467045_ENSMUSG00000041730_MOUSE_457_903/1-449 GAGAGACCGCCCTTCTCTCTGCCTGGGTGAAAATTAAACTCTAAAGTACCGGGTTGAAAT
467045_ENSG00000165606_HUMAN_8456_8903/1-449 GAGAGACCGCCCTTCTCTCTGCCTGGGTGAAAATTAAACTCTAAAGTACCGGGCTGAAAT
467045_ENSRNOG00000020045_RAT_7890_8336/1-449 GAGAGACCGCCCTTCTCTCTGCCTGGGTGAAAATTAAACTCTAAAGTACCGGGTTGAAAT
467045_SINFRUG00000142795_FUGU_773_1208/1-449 CAGACGCCATGCGTCTCTGAGGTGTAGGCAAAATGAGGCT-TCGAGAAGC-AACTGAAAT
467045_ENSDARG00000011919_ZEBRAFISH_2244_2674/1-449 CAGACACCGCCGCTC-CTCTG-CT-GCTGGAAATGAGGCT-CCGAGTAGC-AGCTGAAAT
*** ** ** ** * **** * ** ** * * ******
467045_ENSMUSG00000041730_MOUSE_457_903/1-449 TTTCAAGTCAGGGGCGCGGGATTGGATCAAATCACATAAACTGCAAAAAAAGCAAGTCTA
467045_ENSG00000165606_HUMAN_8456_8903/1-449 TTTCAAGTCAGGGGCGCGGGATTGGATCAAATCACATAAACTGCAAAAAAAGCAAGTCTA
467045_ENSRNOG00000020045_RAT_7890_8336/1-449 TTTCAAGTCAGGGGCGCGGGATTGGATCAAATCACATAAACTGCAAAAAAAGCAAGTCTA
467045_SINFRUG00000142795_FUGU_773_1208/1-449 TTAGAAG--AGGAGTGCGGGATTGGACTAAACCACATAAAGTGC-AGAAAATCCCTTCTA
467045_ENSDARG00000011919_ZEBRAFISH_2244_2674/1-449 TTTGAAG--TGCGGGACGGTATTGGATGAAATCACATAAACTGC-AAAAAAGCAAGTGTA
** *** * * *** ****** *** ******** *** * **** * * **
467045_ENSMUSG00000041730_MOUSE_457_903/1-449 ATGGCGCATAATTGGTTCTGTAATAGCATTACACCAGGATAATAGCCCGTTACGGCCCC
467045_ENSG00000165606_HUMAN_8456_8903/1-449 ATGGCGCATAATTGGTTCTGTAATAGCATTACACCAGGATAATAGCCCGTTACGGCCCC
467045_ENSRNOG00000020045_RAT_7890_8336/1-449 ATGGCGCATAATTGGTTCTGTAATAGCATTACACCAGGATAATAGCCCGTTACGGCCCC
467045_SINFRUG00000142795_FUGU_773_1208/1-449 ATGGCGCGTAATTGGTTCTGTAATATCATTACAGGCGGATAATGGCCAGTTACAGGGCC
467045_ENSDARG00000011919_ZEBRAFISH_2244_2674/1-449 ATGGTGCATAATTGGTTCTCTAATAGCATTACACAAGGATAATAGCCTGTTATGGCCCC
**** ** *********** ***** ******* ******* *** **** * **
467045_3-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 299
Mean pairwise identity: 81.85
Mean single sequence MFE: -92.66
Consensus MFE: -50.44
Energy contribution: -54.68
Covariance contribution: 4.24
Mean z-score: -2.01
Structure conservation index: 0.54
SVM decision value: 0.07
SVM RNA-class probability: 0.569489
Prediction: RNA
######################################################################
>467045_ENSMUSG00000041730_MOUSE_457_903/1-449
UUUGAUUUUGUUCUCAGCAAGGGGUCCCUGAAUUUAUGUUCGCUUCUCCCUGCAAAUGAUAUGCAGUGGGACCCUUCGCAAUUACUUUUAAAAUGCAUCCGAGGCAGACACUCAGCGGAGAGAGACCGCCCUUCUCUCUGCCUGGGUGAAAAUUAAACUCUAAAGUACCGGGUUGAAAUUUUCAAGUCAGGGGCGCGGGAUUGGAUCAAAUCACAUAAACUGCAAAAAAAGCAAGUCUAAUGGCGCAUAAUUGGUUCUGUAAUAGCAUUACACCAGGAUAAUAGCCCGUUACGGCCCC
((((((((...((((.((.(((((((((.((((....))))........(((((.......))))).))))))))).))...............((..((..(((....(((((((((((((((.......))))))))).))))))(((((((.(((((.........)))))..)))))))..)))..))..))))))..))))))))..........(((.......))).(((((((((.((....(((((...(((((...)))))))))).......)))))))).)))... ( -91.90)
>467045_ENSG00000165606_HUMAN_8456_8903/1-449
GUUUGAUUUUGUUCUCAGCAAGGGGUCCCUGAAUUUAUGUUCGCUUCUCGAUGCAAAUGAUAUGCGGUGGGACCCUUUGCAAUUACUUUUAAAAUGCAUCCGAGGCAGACACUCAACGGAGAGAGACCGCCCUUCUCUCUGCCUGGGUGAAAAUUAAACUCUAAAGUACCGGGCUGAAAUUUUCAAGUCAGGGGCGCGGGAUUGGAUCAAAUCACAUAAACUGCAAAAAAAGCAAGUCUAAUGGCGCAUAAUUGGUUCUGUAAUAGCAUUACACCAGGAUAAUAGCCCGUUACGGCCCC
((((((((((.......((((((((((((.((((....)))).........((((.......))))..))))))))))))................(((((.(((((((..(((....))).((((.......))))))))))))))))))))))))))......(((.((((((((.....))..(((..((((((..((((......)))).(((..(((((.......)).)))...))))))).......((..(((....)))..)).))..)))...)))))).)))...... ( -93.20)
>467045_ENSRNOG00000020045_RAT_7890_8336/1-449
UUUGAUUUUGUUCUCAGCAAGGGGUCCCUGAAUUUAUGUUCGCUUCUCCCUGCAAAUGAUAUGCAGUGGGACCCUUCGCAAUUAUUUUUAAAAUGCAUCCGAGGCAGACACUCAGCGGAGAGAGACCGCCCUUCUCUCUGCCUGGGUGAAAAUUAAACUCUAAAGUACCGGGUUGAAAUUUUCAAGUCAGGGGCGCGGGAUUGGAUCAAAUCACAUAAACUGCAAAAAAAGCAAGUCUAAUGGCGCAUAAUUGGUUCUGUAAUAGCAUUACACCAGGAUAAUAGCCCGUUACGGCCCC
((((((((...((((.((.(((((((((.((((....))))........(((((.......))))).))))))))).))...............((..((..(((....(((((((((((((((.......))))))))).))))))(((((((.(((((.........)))))..)))))))..)))..))..))))))..))))))))..........(((.......))).(((((((((.((....(((((...(((((...)))))))))).......)))))))).)))... ( -91.90)
>467045_SINFRUG00000142795_FUGU_773_1208/1-449
UUUGAUUUUGUUCACAGCAAGGGGUCCCGCGAUUUAUUUCGCUUCCUCAGAUAAAUGAUCUGCUGCGGGACCCUUUCAAUUAGUUUUAAAGCGCAUCAGGGACGUCAGAGCGCAGACGCCAUGCGUCUCUGAGGUGUAGGCAAAAUGAGGCUUCGAGAAGCAACUGAAAUUUAGAAGAGGAGUGCGGGAUUGGACUAAACCACAUAAAGUGCAGAAAAUCCCUUCUAAUGGCGCGUAAUUGGUUCUGUAAUAUCAUUACAGGCGGAUAAUGGCCAGUUACAGGGCC
.((.(((((((..(((.(((((((((((((((......)).......(((((.....)))))..))))))))))))...................(((((((((.((..(((....)))..)))))))))))).)))..))))))).))((((....)))).........(((((((.((((..(...(((((......))).))...)..)......)))))))))).(((.((((((((((((((((((...)))))))).........))))))))).).))) ( -98.30)
>467045_ENSDARG00000011919_ZEBRAFISH_2244_2674/1-449
UUUUGAUUUUGUUCACAGCAAGGGGUCCGAGGAUUUAUUUCCCACUCUCGCGUAAAUGAUAUGCAGCGGGACCCUUUCAAUUACUUUUAAAACGCACGCGGGACGCCGCGCGCAGACACCGCCGCUCCUCUGCUGCUGGAAAUGAGGCUCCGAGUAGCAGCUGAAAUUUUGAAGUGCGGGACGGUAUUGGAUGAAAUCACAUAAACUGCAAAAAAGCAAGUGUAAUGGUGCAUAAUUGGUUCUCUAAUAGCAUUACACAAGGAUAAUAGCCUGUUAUGGCCCC
...((((((..((((..(.((((((((((.(((......)))).(.((.(((((.....))))))).)))))))))))...............((.(((((....))))).))....((((((((((..(((((((((((........))).))))))))..))...........))))..))))..))))..)))))).......(((......))).((((((((.(.....(((((....)))))).))))))))..........(((......)))... ( -88.00)
>consensus
_UUUGAUUUUGUUCUCAGCAAGGGGUCCCUGAAUUUAUGUUCGCUUCUCCAUGCAAAUGAUAUGCAGUGGGACCCUUUGCAAUUACUUUUAAAAUGCAUCCGAGGCAGACACUCAGCGGAGAGAGACCGCCCUUCUCUCUGCCUGGGUGAAAAUUAAACUCUAAAGUACCGGGCUGAAAUUUUCAAGUCAGGGGCGCGGGAUUGGAUCAAAUCACAUAAACUGCAAAAAAAGCAAGUCUAAUGGCGCAUAAUUGGUUCUGUAAUAGCAUUACACCAGGAUAAUAGCCCGUUACGGCCCC
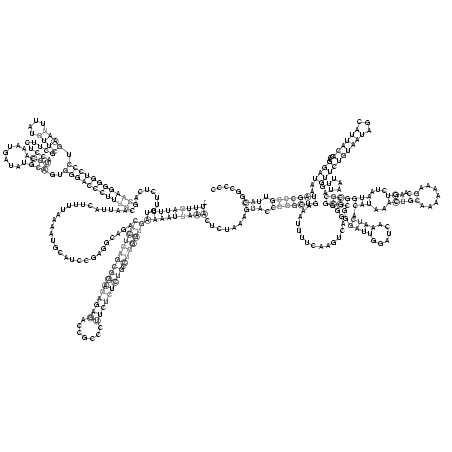
.(((((((((.......((((((((((((.((((....)))).........((((.......))))..)))))))))))).............................((((((((((((((((.......))))))))).)))))))))))))))).......(((.(((((((.................((((..((((......)))).(((..(((((.......)).)))...))))))).......((((((((((...))))))...))))..))))))).)))...... (-50.44 = -54.68 + 4.24)
467045_3-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004