Index >
Results for CNB 453969-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
453969_ENSMUSG00000007817_MOUSE_796_1002/1-208 CGTGTGTGCCAAAGGGGCTGATGCCGGGTGGTCGGGGCTGGTTCATGCCCATGGGAGGGC
453969_ENSDARG00000013677_ZEBRAFISH_9793_10000/1-208 CATGGCCACCAAAGGGCCCCATGCCTGGGCCTCTGGGCTGGTTCATACCCATCGGTGGGC
453969_SINFRUG00000145892_FUGU_8334_8524/1-208 -ATGGGCACCAAAAGGCCCCATGCCTTGGCCCCTGGGCTGGTTCATTCCCATGGGAGGCC
453969_ENSRNOG00000010488_RAT_796_1002/1-208 CGTGTGTGCCAAAGGGGCTGATGCCAGGCGGTCGGGGCTGGTTCATGCCCATGGGAGGGC
** ***** ** * ***** * * ************ ***** ** ** *
453969_ENSMUSG00000007817_MOUSE_796_1002/1-208 CGCTCATGCCCGAGGGTGTCATGCCAGCTGCCATGCCGGCAGGGTTCAGGCTGCCCCCCA
453969_ENSDARG00000013677_ZEBRAFISH_9793_10000/1-208 CGCTCATGTTGGAGCTATTCATTCCACCACCCATAGCAGCTGGGTTCATGCCTCCTGCCA
453969_SINFRUG00000145892_FUGU_8334_8524/1-208 CACTCATGTTGGACGCATTCATGCCTGCGCCCA-------TGTTTCCAGGGAGC------
453969_ENSRNOG00000010488_RAT_796_1002/1-208 CGCTCATGCCCGAGGGTGTCATGCCAGCTGCCATGCTGGCAGGGTTCATGCTGCCCCCCA
* ****** ** **** ** * *** * * ** * *
453969_ENSMUSG00000007817_MOUSE_796_1002/1-208 TGGAGGCAGGCCCCCGAGGCCCGGGTTGGTTCATGAATTGGCTGTTATACGCCTGGGTGG
453969_ENSDARG00000013677_ZEBRAFISH_9793_10000/1-208 TGGATGGGGGTCCGCGAGGCCCTGGCTGGTTCATAAACTGGCTGTTGTAGCCTTGCGTAG
453969_SINFRUG00000145892_FUGU_8334_8524/1-208 --GATGCGGGGCCGCGAGGCCCTGGCTGGTTCATGAACTGGCTGTTGTATGCCTGGTTTG
453969_ENSRNOG00000010488_RAT_796_1002/1-208 TGGAGGCAGGCCCCCGAGGCCCGGGTTGGTTCATGAATTGGCTGTTATACGCCTGGGTGG
** * ** ** ******** ** ******** ** ******** ** * ** * *
453969_ENSMUSG00000007817_MOUSE_796_1002/1-208 GACCCATCTGGAAAGAGGAACACACCT-
453969_ENSDARG00000013677_ZEBRAFISH_9793_10000/1-208 GCCCCATCTGGAATGAGGAGCAAACCTG
453969_SINFRUG00000145892_FUGU_8334_8524/1-208 GTGCCATCTGAAAAGAGGAGCTCATCT-
453969_ENSRNOG00000010488_RAT_796_1002/1-208 GACCCATCTGGAAAGAGGAACACACCT-
* ******* ** ***** * * **
453969-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 208
Mean pairwise identity: 75.34
Mean single sequence MFE: -97.75
Consensus MFE: -60.96
Energy contribution: -61.27
Covariance contribution: 0.31
Mean z-score: -2.75
Structure conservation index: 0.62
SVM decision value: 3.33
SVM RNA-class probability: 0.999016
Prediction: RNA
######################################################################
>453969_ENSMUSG00000007817_MOUSE_796_1002/1-208
CGUGUGUGCCAAAGGGGCUGAUGCCGGGUGGUCGGGGCUGGUUCAUGCCCAUGGGAGGGCCGCUCAUGCCCGAGGGUGUCAUGCCAGCUGCCAUGCCGGCAGGGUUCAGGCUGCCCCCCAUGGAGGCAGGCCCCCGAGGCCCGGGUUGGUUCAUGAAUUGGCUGUUAUACGCCUGGGUGGGACCCAUCUGGAAAGAGGAACACACCU
.((((((.((...(((.((.((.(((((((...(.(((..(((((((.(((((((.((((.(((...(((((..(((.....)))..)((((.....))))))))...))).))))))))))).....((((((((.....))))..)))))))))))..))).)....))))))))).)).))).(((....))))).)))))).. ( -101.90)
>453969_ENSDARG00000013677_ZEBRAFISH_9793_10000/1-208
CAUGGCCACCAAAGGGCCCCAUGCCUGGGCCUCUGGGCUGGUUCAUACCCAUCGGUGGGCCGCUCAUGUUGGAGCUAUUCAUUCCACCACCCAUAGCAGCUGGGUUCAUGCCUCCUGCCAUGGAUGGGGGUCCGCGAGGCCCUGGCUGGUUCAUAAACUGGCUGUUGUAGCCUUGCGUAGGCCCCAUCUGGAAUGAGGAGCAAACCUG
...((...((((.((((((.......)))))).(((((.(((((((........))))))))))))..)))).(((.(((((((((.((.((((.((((..((((....)))).)))).)))).))((((((((((((((...(((..(((....)))..)))......))))))))..))))))...))))))))).)))...)).. ( -93.70)
>453969_SINFRUG00000145892_FUGU_8334_8524/1-208
AUGGGCACCAAAAGGCCCCAUGCCUUGGCCCCUGGGCUGGUUCAUUCCCAUGGGAGGCCCACUCAUGUUGGACGCAUUCAUGCCUGCGCCCAUGUUUCCAGGGAGCGAUGCGGGGCCGCGAGGCCCUGGCUGGUUCAUGAACUGGCUGUUGUAUGCCUGGUUUGGUGCCAUCUGAAAAGAGGAGCUCAUCU
((((((.((....(((.(((.(((..((((((((((.(((..(..(((....))))..)))..((((..((.((((........))))))))))..))))))).(((((.(((((((....)))))))((..(((....)))..)).)))))..))).))).))).))).(((....))))).)))))).. ( -87.50)
>453969_ENSRNOG00000010488_RAT_796_1002/1-208
CGUGUGUGCCAAAGGGGCUGAUGCCAGGCGGUCGGGGCUGGUUCAUGCCCAUGGGAGGGCCGCUCAUGCCCGAGGGUGUCAUGCCAGCUGCCAUGCUGGCAGGGUUCAUGCUGCCCCCCAUGGAGGCAGGCCCCCGAGGCCCGGGUUGGUUCAUGAAUUGGCUGUUAUACGCCUGGGUGGGACCCAUCUGGAAAGAGGAACACACCU
.((((((.((...(((.((.((.(((((((...(.(((..(((((((.(((((((.((((.((....((((((.....)).(((((((......)))))))))))....)).))))))))))).....((((((((.....))))..)))))))))))..))).)....))))))))).)).))).(((....))))).)))))).. ( -107.90)
>consensus
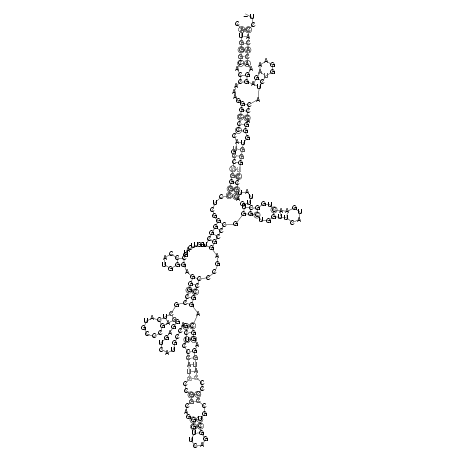
CAUGGGCACCAAAGGGCCCCAUGCCUGGGCCUCGGGGCUGGUUCAUGCCCAUGGGAGGGCCGCUCAUGCCCGAGGGAGUCAUGCCAGCUCCCAUGCCGGCAGGGUUCAGGCUGCCCCCCAUGGAGGCAGGCCCCCGAGGCCCGGGCUGGUUCAUGAACUGGCUGUUAUACGCCUGGGUGGGACCCAUCUGGAAAGAGGAACACACCU_
.((((((.((....(((((.((.(((((((....(((((........((....)).(((((.(((......)))............(((.(((((..((...(((....)))..))..))))).))).)))))....))))).(((..(((....)))..)))......))))))))).)).))).(((....))))).))))))... (-60.96 = -61.27 + 0.31)
453969-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004