Index >
Results for CNB 407017_4
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
407017_ENSG00000184486_HUMAN_1441_1832/1-455 --TTTTA-CTGGATGTGAG-----AGCG----CTCT------TGGTCTTAGGAGTGA---
407017_ENSRNOG00000006908_RAT_1055_1446/1-455 --TTTTA-CTGGATGTGAG-----AGCA----CTCT------TGGTATTAGGAGTGA---
407017_SINFRUG00000149835_FUGU_6576_7027/1-455 GGTTTCAGCTGCACAAGAGGAGGCAGCGTGGCCCCTAACATTGGCCCCTCTGAGTGATTG
407017_ENSMUSG00000052621_MOUSE_1366_1756/1-455 --TTTTA-CTGGATGTGAG-----AGCA----CTCT------TGGTATTAGGAGCGA---
407017_ENSDARG00000026918_ZEBRAFISH_7280_7673/1-455 --TTTCA-CTGTATTTGAG-----AGCG----CGCT------GGCCCTTCCAAATGA---
407017_ENSG00000184486_HUMAN_1441_1832/1-455 -CTCACCACAAACCCACCCTTCCCAATAACAAGA-TCACATCATGTATGCATTATTAAGC
407017_ENSRNOG00000006908_RAT_1055_1446/1-455 -CTCACTGAAAACCTTCCTAACCCAATAACAAGA-TCACATCATGTATGCATTATTAAGC
407017_SINFRUG00000149835_FUGU_6576_7027/1-455 AATAACTGAGGACCCCGCAGTCCAAATAACAAGGCTCGCATCATTTATGCATTGTTAAGC
407017_ENSMUSG00000052621_MOUSE_1366_1756/1-455 -CTCACTGCAAACCTTCCTATCCCAATAACAAGA-TCACATCATGTATGCATTATTAAGC
407017_ENSDARG00000026918_ZEBRAFISH_7280_7673/1-455 -ATTACCGTAGACCCCACAGTCCAAATAACAAGACTCGCATCATTTATGCGTTGTTAAGC
407017_ENSG00000184486_HUMAN_1441_1832/1-455 CACATAATTGAA-CGGTCCATAGTTTGCATATCAGACTATTAAAATAATGTGTTTTGCTT
407017_ENSRNOG00000006908_RAT_1055_1446/1-455 TACATAATTGAA-CAGTCTATAGTTTGCATATCAGACTATTAAAATAATGTGTTTTGCTT
407017_SINFRUG00000149835_FUGU_6576_7027/1-455 CATGTAATTGAAACCAGCCATAGTTTGCATATTACACCATTAAAATAATGTGCTTTGCGT
407017_ENSMUSG00000052621_MOUSE_1366_1756/1-455 TACATAATTGAA-CAGTCTATAGTTTGCATATCAGACTATTAAAATAATGTGTTTTGCTT
407017_ENSDARG00000026918_ZEBRAFISH_7280_7673/1-455 CATGTAATTGAA-CGATCCATAGTTTGCATATTATACCATTAAAGTAATGTGCTTTGGTT
407017_ENSG00000184486_HUMAN_1441_1832/1-455 TTCTCTGAACACGTATTGTTCAAGCCTCTGCGAGTATGTAGTGATTCTGATAATTACCTC
407017_ENSRNOG00000006908_RAT_1055_1446/1-455 TTCTCTGAACACGTATTGTTCAAGCCTCTGCGAATATGTGGTGATTCTGATAATTATCAC
407017_SINFRUG00000149835_FUGU_6576_7027/1-455 TTCTCCCAACACG-ACTATTTAAGCCTGTGTGGCGATGTGGTAATTCCTTGAATTACTGC
407017_ENSMUSG00000052621_MOUSE_1366_1756/1-455 TTCTCTGAACACGTATTGTTCAAGCCTCTGCGAATATGTGGTGATTCTGATAATTATCAC
407017_ENSDARG00000026918_ZEBRAFISH_7280_7673/1-455 TTCTCCCAACACG-ACTATTTAAGCTTCTGTGGCGTTGTGGTAATTCTCTGAATTACAAC
407017_ENSG00000184486_HUMAN_1441_1832/1-455 CA-AACTACTGCAAA
407017_ENSRNOG00000006908_RAT_1055_1446/1-455 CA-AATTACAGCAAA
407017_SINFRUG00000149835_FUGU_6576_7027/1-455 CT-AATTACCACAA-
407017_ENSMUSG00000052621_MOUSE_1366_1756/1-455 CA-AATTACAGCAAA
407017_ENSDARG00000026918_ZEBRAFISH_7280_7673/1-455 CTTAATTGCCATAAA
407017_4.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 5
Columns: 255
Mean pairwise identity: 77.02
Mean single sequence MFE: -57.53
Consensus MFE: -16.96
Energy contribution: -18.64
Covariance contribution: 1.68
Mean z-score: -2.00
Structure conservation index: 0.29
SVM decision value: 0.65
SVM RNA-class probability: 0.811535
Prediction: RNA
######################################################################
>407017_ENSG00000184486_HUMAN_1441_1832/1-455
UUUUACUGGAUGUGAGAGCGCUCUUGGUCUUAGGAGUGACUCACCACAAACCCACCCUUCCCAAUAACAAGAUCACAUCAUGUAUGCAUUAUUAAGCCACAUAAUUGAACGGUCCAUAGUUUGCAUAUCAGACUAUUAAAAUAAUGUGUUUUGCUUUUCUCUGAACACGUAUUGUUCAAGCCUCUGCGAGUAUGUAGUGAUUCUGAUAAUUACCUCCAAACUACUGCAAA
......((((.(((((((((((((((....)))))))).))).........................((..(((((..((((..((((((((........))))(((((((((..((((((((.....)))))))).......((((((((...........))))))))))))))))).....))))..))))..)))))..))....)))).))))............ ( -53.20)
>407017_ENSRNOG00000006908_RAT_1055_1446/1-455
UUUUACUGGAUGUGAGAGCACUCUUGGUAUUAGGAGUGACUCACUGAAAACCUUCCUAACCCAAUAACAAGAUCACAUCAUGUAUGCAUUAUUAAGCUACAUAAUUGAACAGUCUAUAGUUUGCAUAUCAGACUAUUAAAAUAAUGUGUUUUGCUUUUCUCUGAACACGUAUUGUUCAAGCCUCUGCGAAUAUGUGGUGAUUCUGAUAAUUAUCACCAAAUUACAGCAAA
........((((((((((((((((((....)))))))).)))...(((....))).................)))))))(((((.((........)))))))..(((((((((..((((((((.....)))))))).......((((((((...........))))))))))))))))).....(((.......(((((((..........))))))).......))).. ( -58.74)
>407017_SINFRUG00000149835_FUGU_6576_7027/1-455
GGUUUCAGCUGCACAAGAGGAGGCAGCGUGGCCCCUAACAUUGGCCCCUCUGAGUGAUUGAAUAACUGAGGACCCCGCAGUCCAAAUAACAAGGCUCGCAUCAUUUAUGCAUUGUUAAGCCAUGUAAUUGAAACCAGCCAUAGUUUGCAUAUUACACCAUUAAAAUAAUGUGCUUUGCGUUUCUCCCAACACGACUAUUUAAGCCUGUGUGGCGAUGUGGUAAUUCCUUGAAUUACUGCCUAAUUACCACAA
(((((..((.((((((((((.(((...(((........)))..))))))))..(((((((...(((((.((((......)))).........((((.(..(((....(((((.(.....).)))))..)))..).)))).)))))..)).))))).............)))))...))(((......)))..........)))))..(((((..(((..(((((((...)))))))..)...))..))))). ( -68.10)
>407017_ENSMUSG00000052621_MOUSE_1366_1756/1-455
UUUUACUGGAUGUGAGAGCACUCUUGGUAUUAGGAGCGACUCACUGCAAACCUUCCUAUCCCAAUAACAAGAUCACAUCAUGUAUGCAUUAUUAAGCUACAUAAUUGAACAGUCUAUAGUUUGCAUAUCAGACUAUUAAAAUAAUGUGUUUUGCUUUUCUCUGAACACGUAUUGUUCAAGCCUCUGCGAAUAUGUGGUGAUUCUGAUAAUUAUCACCAAAUUACAGCAAA
........(((((((........((((...((((((((......)))......)))))..))))........)))))))(((((.((........)))))))..(((((((((..((((((((.....)))))))).......((((((((...........))))))))))))))))).....(((.......(((((((..........))))))).......))).. ( -55.63)
>407017_ENSDARG00000026918_ZEBRAFISH_7280_7673/1-455
UUUCACUGUAUUUGAGAGCGCGCUGGCCCUUCCAAAUGAAUUACCGUAGACCCCACAGUCCAAAUAACAAGACUCGCAUCAUUUAUGCGUUGUUAAGCCAUGUAAUUGAACGAUCCAUAGUUUGCAUAUUAUACCAUUAAAGUAAUGUGCUUUGGUUUUCUCCCAACACGACUAUUUAAGCUUCUGUGGCGUUGUGGUAAUUCUCUGAAUUACAACCUUAAUUGCCAUAAA
.((((....(((((..((.((....)).))..)))))((((((((...(((.((((((......((((((....(((((.....)))))))))))(((..(((......)))....((((((.(((((((((.........))))))))).((((.......))))...))))))....))).)))))).)))..))))))))..))))...................... ( -52.00)
>consensus
__UUUUA_CUGGAUGUGAG_____AGCG____CUCU______UGGUCUUAGGAGUGA____CUCACUGAAAACCCCCCAAUCCCAAUAACAAGA_UCACAUCAUGUAUGCAUUAUUAAGCCACAUAAUUGAA_CAGUCCAUAGUUUGCAUAUCAGACUAUUAAAAUAAUGUGUUUUGCUUUUCUCUGAACACGUAUUGUUCAAGCCUCUGCGAAUAUGUGGUGAUUCUGAUAAUUACCACCA_AAUUACAGCAAA
.........................((..............................................................(((((..(((((((..(((((........))...)))..)))........((((((((.....))))))))........)))))))))........((((((.....)))))).))...........((((((((((.((...........)).)))))))))).. (-16.96 = -18.64 + 1.68)
407017_4.rnaz
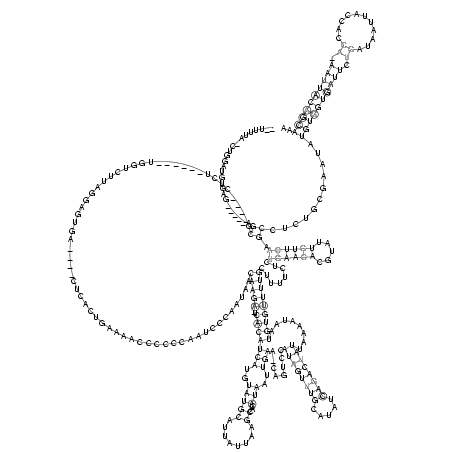
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004