Index >
Results for CNB 376858_9-rev
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
376858_SINFRUG00000142471_FUGU_3228_3898/1-798 ATCACAGACTCCTGAGTGATCTTCAGGTCTCCCTCCAGCTTCCTCTTGGCTCGCTCCAGG
376858_ENSRNOG00000002987_RAT_2681_3395/1-798 ATTGTGGACTCTTGGGCCAGTTTCAGGTCCCCCTCCAGTTTCCTCTTGGCTCTTTCCAGA
376858_ENSDARG00000014514_ZEBRAFISH_217_1012/1-798 ATAATAGACTCTTGGGCCAATTTCAAATCTCCTTCAAGCTTTCTCTTGGCTCTCTCCAGG
376858_ENSMUSG00000055724_MOUSE_2723_3438/1-798 ATTGTGGACTCTTGGGCCAGTTTCAGGTCCCCCTCCAGTTTCCTCTTGGCTCTTTCTAAG
** ***** ** * * **** ** ** ** ** ** ********** ** *
376858_SINFRUG00000142471_FUGU_3228_3898/1-798 TCCATCCGAATCTTCTTCTCCTGCTCCAGTGAACCTTCAAGCT-TGGTGGCAA-ATCAAA
376858_ENSRNOG00000002987_RAT_2681_3395/1-798 TCCATCCTCAGCTTCTTTTCTTGCTCTAGCGATCCTTCAAGCTGATGATAGAAGAGAAAG
376858_ENSDARG00000014514_ZEBRAFISH_217_1012/1-798 TCCATACGAAGCTTCTTTTCTTGTTCAAGGGAACCCTCAAGCTGTGGGACAATGATATTT
376858_ENSMUSG00000055724_MOUSE_2723_3438/1-798 TCCATCCTCAGCTTCTTTTCTTGCTCTAGCGATCCCTCAAGCTGATGGGGAAAGAGAAAC
***** * * ****** ** ** ** ** ** ** ******* * * *
376858_SINFRUG00000142471_FUGU_3228_3898/1-798 CAGTGATGT--GAGATGCCAAACATG-GCCGTTGCTTCCCGGCAGC-AA-CAGATACTCA
376858_ENSRNOG00000002987_RAT_2681_3395/1-798 CCATTAAAATCGAAATTCACTTAAAGAAATGTTTCAGGAGGGCATTTGA-AACATGCTCA
376858_ENSDARG00000014514_ZEBRAFISH_217_1012/1-798 AAAATATGTTCG-AAGTAAAAAAGGTAAATAATTTACTTCCATGTTTAATGAAGAGCTCA
376858_ENSMUSG00000055724_MOUSE_2723_3438/1-798 CTGTTAAAATCGAAATGCACTTAAAGAAATACTTCAGAAGGCCATCTGA-ACCGTGCTTA
* * * * * ** *
376858_SINFRUG00000142471_FUGU_3228_3898/1-798 CGTCATTCACTTGCTGTTCCAGTTTGGACTTGGCTTTGGTCAAGGTGTTGACCTTATCTT
376858_ENSRNOG00000002987_RAT_2681_3395/1-798 CATCATCCACTTGCTGTTCGAGCTTGGTTTTGGCTTTGGTCAGGGTGTTGACTTTGTCCT
376858_ENSDARG00000014514_ZEBRAFISH_217_1012/1-798 CATCATCAACTTGCTGCTCAAGCTTTGTCTTGGATTTGGTCAGGGTGTTGACTTTGTCCT
376858_ENSMUSG00000055724_MOUSE_2723_3438/1-798 CGTCATCCACTTGCTGTTCCAGCTTGGTTTTGGCTTTGGTCAGGGTGTTGACTTTGTCCT
* **** ******** ** ** ** * **** ******** ********* ** ** *
376858_SINFRUG00000142471_FUGU_3228_3898/1-798 CCTCAGCCTGAAGGTCATCCAGAACCTGCTGATGGGCTTCCTGAAGGGCTCTCTTTTCCT
376858_ENSRNOG00000002987_RAT_2681_3395/1-798 CCTCTGCCTGCAGGTCATCCAGGGTCTGCTGGTGGGCCTCCTGCAAGGCCTTCTTCTCCT
376858_ENSDARG00000014514_ZEBRAFISH_217_1012/1-798 CCTCAGCCTGAAGATCATCCAGAGTCTGCTGATGTGCCTCTTGGAGGGCTTTCTTCTCCT
376858_ENSMUSG00000055724_MOUSE_2723_3438/1-798 CCTCTGCCTGCAGGTCATCCAGGGTCTGCTGGTGGGCCTCCTGGAGGGCCTTCTTCTCCT
**** ***** ** ******** ****** ** ** ** ** * *** **** ****
376858_9-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 300
Mean pairwise identity: 74.51
Mean single sequence MFE: -93.58
Consensus MFE: -56.21
Energy contribution: -56.65
Covariance contribution: 0.44
Mean z-score: -2.53
Structure conservation index: 0.60
SVM decision value: 2.77
SVM RNA-class probability: 0.996946
Prediction: RNA
######################################################################
>376858_SINFRUG00000142471_FUGU_3228_3898/1-798
AUCACAGACUCCUGAGUGAUCUUCAGGUCUCCCUCCAGCUUCCUCUUGGCUCGCUCCAGGUCCAUCCGAAUCUUCUUCUCCUGCUCCAGUGAACCUUCAAGCUUGGUGGCAAAUCAAACAGUGAUGUGAGAUGCCAAACAUGGCCGUUGCUUCCCGGCAGCAACAGAUACUCACGUCAUUCACUUGCUGUUCCAGUUUGGACUUGGCUUUGGUCAAGGUGUUGACCUUAUCUUCCUCAGCCUGAAGGUCAUCCAGAACCUGCUGAUGGGCUUCCUGAAGGGCUCUCUUUUCCU
.............(((.(..(((((((...(((..((((....((((((((....(((((((((..((((.....)))....((...(((((........(((....)))..........((((((((((..((((....)))).((((((....))))))........))))))))))))))).)).......)..)))))))))....))))))))...(((((((..((.....))....)))))))..........))))..)))...)))))))..).)))....... ( -96.40)
>376858_ENSRNOG00000002987_RAT_2681_3395/1-798
AUUGUGGACUCUUGGGCCAGUUUCAGGUCCCCCUCCAGUUUCCUCUUGGCUCUUUCCAGAUCCAUCCUCAGCUUCUUUUCUUGCUCUAGCGAUCCUUCAAGCUGAUGAUAGAAGAGAAAGCCAUUAAAAUCGAAAUUCACUUAAAGAAAUGUUUCAGGAGGGCAUUUGAAACAUGCUCACAUCAUCCACUUGCUGUUCGAGCUUGGUUUUGGCUUUGGUCAGGGUGUUGACUUUGUCCUCCUCUGCCUGCAGGUCAUCCAGGGUCUGCUGGUGGGCCUCCUGCAAGGCCUUCUUCUCCU
...(..(((.((((((((((((((((((..((((((..........(((((.((((....((.(((.(((((((......((((....))))......))))))).))).)).)))).)))))........(((((..............))))).)))))).)))))))))..(((((((.((......)).)))..)))).))))..((((((.((.(((((.(..(((...)))..))))))))...)))))).))))))))..).((.((((((......)))))).))...... ( -97.74)
>376858_ENSDARG00000014514_ZEBRAFISH_217_1012/1-798
AUAAUAGACUCUUGGGCCAAUUUCAAAUCUCCUUCAAGCUUUCUCUUGGCUCUCUCCAGGUCCAUACGAAGCUUCUUUUCUUGUUCAAGGGAACCCUCAAGCUGUGGGACAAUGAUAUUUAAAAUAUGUUCGAAGUAAAAAAGGUAAAUAAUUUACUUCCAUGUUUAAUGAAGAGCUCACAUCAUCAACUUGCUGCUCAAGCUUUGUCUUGGAUUUGGUCAGGGUGUUGACUUUGUCCUCCUCAGCCUGAAGAUCAUCCAGAGUCUGCUGAUGUGCCUCUUGGAGGGCUUUCUUCUCCU
....((((((((.(((((((....(((.((......)).)))...)))))))((((((((((((.(((((((((.((..(((....)))..))......(((.(..((...(((((.....(((((((...((((((((............)))))))))))))))..(((.....))).)))))...))..).))).)))))))))..)))))))))(((((.((..((........))..)).))))))))......))))))))..((...((((......))))..))....... ( -79.70)
>376858_ENSMUSG00000055724_MOUSE_2723_3438/1-798
AUUGUGGACUCUUGGGCCAGUUUCAGGUCCCCCUCCAGUUUCCUCUUGGCUCUUUCUAAGUCCAUCCUCAGCUUCUUUUCUUGCUCUAGCGAUCCCUCAAGCUGAUGGGGAAAGAGAAACCUGUUAAAAUCGAAAUGCACUUAAAGAAAUACUUCAGAAGGCCAUCUGAACCGUGCUUACGUCAUCCACUUGCUGUUCCAGCUUGGUUUUGGCUUUGGUCAGGGUGUUGACUUUGUCCUCCUCUGCCUGCAGGUCAUCCAGGGUCUGCUGGUGGGCCUCCUGGAGGGCCUUCUUCUCCU
.....(((.....(((((...((((((...(((.(((((....((((((((((((((....(((...(((((((......((((....))))......))))))))))))))))))..((((((.................................(((((((...((((((.(((.(((.((......)).)))...))).)))))))))))))((.(((((.(..(((...)))..)))))))).))))))...))))))...))))).)))...)))))).))))).....))). ( -100.50)
>consensus
AUUAUAGACUCUUGGGCCAGUUUCAGGUCCCCCUCCAGCUUCCUCUUGGCUCUCUCCAGGUCCAUCCGAAGCUUCUUUUCUUGCUCUAGCGAACCCUCAAGCUGAGGGGGAAAGAGAAAACAAUUAAAAUCGAAAUGCAAAAAAAGAAAUAUUUCAGAACGGCAUCUAA_AACAUGCUCACAUCAUCCACUUGCUGUUCCAGCUUGGUCUUGGCUUUGGUCAGGGUGUUGACUUUGUCCUCCUCAGCCUGAAGGUCAUCCAGAGUCUGCUGAUGGGCCUCCUGGAGGGCCUUCUUCUCCU
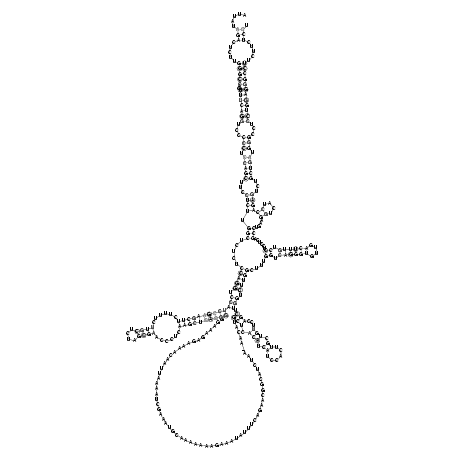
.....(((.....(((((..(((((((...(((.(((((...((((.(((.....(((((.(((((((((((((......((((....))))......))))))))))...................................................................(((.(((.((......)).)))...))).))).)))))....((.((((((....)))))).))......)))....((....))))))...))))).)))...)))))))))))).....))). (-56.21 = -56.65 + 0.44)
376858_9-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004